分子生物学导论/DNA 生命的单位
基因由一个叫做 DNA 的长分子组成,它在世代之间被复制和遗传。DNA 由简单的单元组成,这些单元在该大分子中以特定的顺序排列。这些单元的顺序携带着遗传信息,类似于页面上字母的顺序携带着信息。DNA 使用的语言称为遗传密码,它允许生物体读取基因中的信息。该信息是构建和运行活生物体的指令。
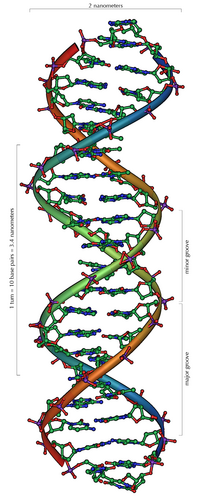
脱氧核糖核酸 (DNA):脱氧核糖核酸 (/diˌɒksiˌraɪbɵ.njuːˌkleɪ.ɨk ˈæsɪd/,或 DNA,是一种核酸,包含所有已知活生物体(除 RNA 病毒外)发育和功能中使用的遗传指令。DNA 分子的主要作用是长期存储信息。DNA 通常被比作一组蓝图,就像食谱或代码一样,因为它包含构建细胞其他成分(如蛋白质和 RNA 分子)所需的指令。携带这种遗传信息的 DNA 片段称为基因,但其他 DNA 序列具有结构目的,或参与调节这种遗传信息的利用。DNA 由两个简单的单元组成的长聚合物组成,称为核苷酸,其骨架由糖和磷酸基团组成,通过酯键连接。这两条链彼此朝相反方向运行,因此是反平行的。连接到每种糖上的是一种称为碱基的四种类型的分子之一。正是沿着骨架的这四种碱基的顺序编码了信息。该信息使用遗传密码被读取,该密码指定蛋白质中氨基酸的顺序。通过将 DNA 片段复制到相关的核酸 RNA 中的过程(称为转录)来读取该代码。DNA 的结构首先被 **詹姆斯·D·沃森和弗朗西斯·克里克** 发现。对于所有物种来说都是一样的,由两条螺旋链组成,每条螺旋链围绕同一个轴线盘旋,每条螺旋链的螺距为 34 埃(3.4 纳米),半径为 10 埃(1.0 纳米)。
在细胞内,DNA 被组织成称为染色体的长结构。在细胞分裂之前,这些染色体会复制,这一过程称为 DNA 复制。真核生物(动物、植物、真菌和原生生物)将大部分 DNA 存储在细胞核内,并将一些 DNA 存储在细胞器中,例如线粒体或叶绿体。相反,原核生物(细菌和古细菌)仅在细胞质中存储 DNA。在染色体内,组蛋白等染色质蛋白压缩和组织 DNA。这些紧凑的结构指导 DNA 与其他蛋白质之间的相互作用,帮助控制 DNA 的哪些部分被转录。DNA 双螺旋通过连接到两条链上的碱基之间的氢键而稳定。DNA 中发现的四种碱基是腺嘌呤(缩写为 A)、胞嘧啶(C)、鸟嘌呤(G)和胸腺嘧啶(T)。这四种碱基与糖/磷酸连接形成完整的核苷酸,如腺苷一磷酸所示。[1]


格里菲斯实验由弗雷德里克·格里菲斯于 1928 年进行,是第一个表明细菌能够通过称为转化的过程转移遗传信息的实验之一。
格里菲斯使用了两种感染小鼠的肺炎链球菌菌株——III-S(光滑)型和 II-R(粗糙)型菌株。III-S 菌株用多糖荚膜包裹自身,使其免受宿主免疫系统的攻击,导致宿主死亡,而 II-R 菌株没有这种保护性荚膜,被宿主免疫系统击败。德国细菌学家弗雷德·诺伊费尔德发现了三种肺炎球菌类型(I 型、II 型和 III 型),并发现了奎尔隆反应,以便在体外识别它们。在格里菲斯实验之前,细菌学家认为这些类型是固定的,从一代到一代不会改变。在这个实验中,来自 III-S 菌株的细菌被热杀死,它们的残骸被添加到 II-R 菌株细菌中。虽然它们单独都不能伤害小鼠,但它们的组合能够杀死宿主。格里菲斯还能够从小鼠血液中分离出活的 II-R 和活的 III-S 肺炎球菌菌株。格里菲斯得出结论,II-R 类型已被“转化”为致命的 III-S 菌株,这是由于来自死亡的 III-S 菌株细菌的“转化因子”所致。今天,我们知道格里菲斯观察到的“转化因子”是 III-S 菌株细菌的 DNA。虽然细菌已经被杀死,但 DNA 已经存活了加热过程,并被 II-R 菌株细菌吸收。III-S 菌株 DNA 包含形成保护性多糖荚膜的基因。配备了这个基因,以前的 II-R 菌株细菌现在可以免受宿主免疫系统的攻击,并可以杀死宿主。转化因子的确切性质(DNA)是在艾弗里、麦克劳德和麦卡蒂以及赫尔希和蔡斯进行的实验中验证的。[2]
首次确认
阿尔弗雷德·赫尔希和玛莎·蔡斯在 1952 年进行了一系列实验,证实了 DNA 是遗传物质,这在 1944 年的艾弗里-麦克劳德-麦卡蒂实验中首次得到证明。这些实验被称为 **赫尔希-蔡斯实验**。生物学家自 1869 年以来就知道 DNA 的存在,他们中的大多数人当时都认为蛋白质携带遗传信息。赫尔希和蔡斯在 T2 噬菌体上进行了他们的实验。噬菌体由一个包含其遗传物质的蛋白质外壳组成。噬菌体通过附着在细菌的外膜上并注入其遗传物质来感染细菌,留下其空壳附着在细菌上。
在他们的第一组实验中,赫尔希和蔡斯用放射性磷-32 (p32) 对噬菌体的 DNA 进行了标记(磷元素存在于 DNA 中,但不存在于作为蛋白质成分的 20 种氨基酸中的任何一种)。他们让噬菌体感染大肠杆菌,并通过几个巧妙的实验能够观察到标记有 P32 的噬菌体 DNA 转移到细菌的细胞质中。在他们的第二组实验中,他们用放射性硫-35 对噬菌体进行了标记(硫存在于氨基酸半胱氨酸和蛋氨酸中,但不存在于 DNA 中)。在感染大肠杆菌后,他们使用高速搅拌器将病毒蛋白外壳从受感染的细胞中剪切下来,并使用离心机分离细胞和病毒外壳。分离后,在蛋白质外壳中观察到放射性 S35 示踪剂,但在受感染的细菌中没有观察到,这支持了感染细菌的遗传物质是 DNA 而不是蛋白质的假设。[3][4] **赫尔希因其“关于病毒遗传结构的发现”而获得了 1969 年诺贝尔生理学或医学奖。**
奥斯瓦尔德·T·艾弗里、科林·麦克劳德、麦克林·麦卡蒂以及弗朗西斯·克里克和詹姆斯·D·沃森 [5]


两条螺旋链形成DNA骨架。另一条双螺旋可以通过追踪链之间的空间或沟槽找到。这些空隙与碱基对相邻,可以提供一个结合位点。由于链不是完全相对的,因此沟槽的大小也不相同。一个沟槽,主沟,宽 22 Å,另一个,小沟,宽 12 Å。小沟的狭窄意味着碱基的边缘在主沟中更容易接近。因此,像转录因子这样的蛋白质可以结合到双链 DNA 中的特定序列,通常与主沟中暴露的碱基侧面接触。这种情况在细胞内 DNA 的异常构象中有所不同,但主沟和小沟始终被命名以反映如果 DNA 扭回到普通的 B 形状所看到的尺寸差异。
查伽夫法则由埃尔温·查伽夫提出,其指出来自所有生物任何细胞的DNA应该具有嘧啶和嘌呤碱基的1:1比例,更具体地说,鸟嘌呤的量等于胞嘧啶,腺嘌呤的量等于胸腺嘧啶。这种模式存在于DNA的两条链中。它们是由奥地利化学家埃尔温·查伽夫发现的。
在分子生物学中,通过氢键连接在相反互补 DNA 链上的两个核苷酸称为碱基对(通常缩写为bp)。在规范的沃森-克里克 DNA 碱基配对中,腺嘌呤 (A) 与胸腺嘧啶 (T) 形成碱基对,鸟嘌呤 (G) 与胞嘧啶 (C) 形成碱基对。在 RNA 中,胸腺嘧啶被尿嘧啶 (U) 代替。交替氢键模式,如摆动碱基对和霍格斯汀碱基对,也存在——特别是在 RNA 中——导致复杂的和功能性的三级结构。[6]
示例
5'CTCGTTTGCGCTCTATCG3' 3'GAGCAAACGCGAGATAGC5'
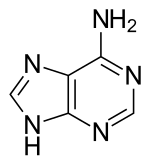
德国化学家埃米尔·费舍尔在 1884 年给出了“嘌呤”(purum uricum)这个名字。他于 1899 年首次通过尿酸合成了它,尿酸是由舍勒在 1776 年从肾结石中分离出来的。除了 DNA 和 RNA 之外,嘌呤也是许多其他重要生物分子中的组成部分,如 ATP、GTP、环状 AMP、NADH 和辅酶 A。嘌呤本身在自然界中没有被发现,但可以通过有机合成来生产。嘌呤是一种杂环芳香族有机化合物,由一个嘧啶环与一个咪唑环融合而成。
示例
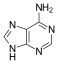
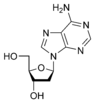
腺嘌呤是构成核酸(DNA 或 RNA)核苷酸的两种嘌呤核碱基之一(另一种是鸟嘌呤)。在 DNA 中,腺嘌呤通过两个氢键与胸腺嘧啶结合,以帮助稳定核酸结构。腺嘌呤与核糖结合形成腺苷,一种核苷,与脱氧核糖结合形成脱氧腺苷。当三个磷酸基团添加到腺苷时,它会形成腺嘌呤三磷酸 (ATP),一种核苷酸。
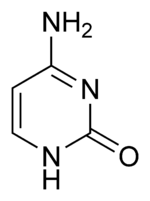
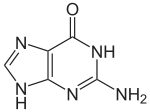
鸟嘌呤与腺嘌呤和胞嘧啶一起存在于 DNA 和 RNA 中,而胸腺嘧啶通常只在 DNA 中看到,而尿嘧啶只在 RNA 中看到。在 DNA 中,鸟嘌呤与胞嘧啶配对。以 C5H5N5O 的化学式,鸟嘌呤是嘌呤的衍生物,由一个融合的嘧啶-咪唑环系统组成,具有共轭双键。
鸟嘌呤有两种互变异构体,主要酮式和罕见的烯醇式。它通过三个氢键与胞嘧啶结合。在胞嘧啶中,氨基作为氢供体,C-2 羰基和 N-3 胺作为氢键受体。鸟嘌呤在 C-6 处有一个基团作为氢受体,而在 N-1 和 C-2 处的氨基作为氢供体。



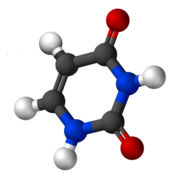
嘧啶是一种杂环芳香族有机化合物,类似于苯和吡啶,在六元环的 1 和 3 位包含两个氮原子。它与二嗪的两种其他形式互为异构体。在核酸中发现的三种核碱基,胞嘧啶 (C)、胸腺嘧啶 (T) 和尿嘧啶 (U),是嘧啶衍生物。
嘧啶与吡啶有很多共同的性质,因为环中氮原子数量的增加使环 pi 电子变得不那么有能量和亲电子性,芳香族亲电取代变得更加困难,而芳香族亲核取代变得更容易。最后一种反应类型的例子是在 2-氨基嘧啶中用氯取代氨基,以及它的逆反应。嘧啶共振稳定性的降低可能会导致加成和开环反应,而不是取代反应。一种这样的表现形式在迪莫斯重排中观察到。与吡啶相比,N-烷基化和 N-氧化更难,嘧啶的碱性也更弱:质子化嘧啶的 pKa 值为1.23,而吡啶的 pKa 值为5.30。[7]嘧啶也存在于陨石中,但科学家仍然不知道它的起源。嘧啶在紫外线下也会光解成尿嘧啶。

胞嘧啶可以作为 DNA 的一部分、RNA 的一部分或核苷酸的一部分找到。作为胞嘧啶三磷酸 (CTP),它可以作为酶的辅因子,并且可以转移一个磷酸基团来将二磷酸腺苷 (ADP) 转化为三磷酸腺苷 (ATP)。胞嘧啶的核苷是胞苷。在 DNA 和 RNA 中,胞嘧啶与鸟嘌呤配对。然而,它本质上是不稳定的,可以变成尿嘧啶(自发脱氨)。如果不对其进行修复,则会导致点突变,例如尿嘧啶糖基化酶之类的 DNA 修复酶可以修复它,尿嘧啶糖基化酶会切除 DNA 中的尿嘧啶。
胞嘧啶也可以被一种称为 DNA 甲基转移酶的酶甲基化为 5-甲基胞嘧啶,或者被甲基化和羟基化为 5-羟甲基胞嘧啶。胞嘧啶或 5-甲基胞嘧啶的活性酶脱氨作用(由 APOBEC 家族的胞嘧啶脱氨酶完成)可能会对各种细胞过程以及生物进化产生有益和有害的影响。另一方面,脱氨作用对 5-羟甲基胞嘧啶的影响仍然不太了解。[8]
胸腺嘧啶 (T, Thy) 是 DNA 核酸中的四种核碱基之一,由字母 G–C–A–T 表示。其他三种是腺嘌呤、鸟嘌呤和胞嘧啶。胸腺嘧啶也被称为 5-甲基尿嘧啶,一种嘧啶核碱基。顾名思义,胸腺嘧啶可以通过在第 5 个碳原子上甲基化尿嘧啶来获得。在 RNA 中,胸腺嘧啶在大多数情况下被尿嘧啶取代。在 DNA 中,胸腺嘧啶 (T) 通过两个氢键与腺嘌呤 (A) 结合,从而稳定核酸结构。
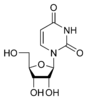
尿嘧啶存在于 RNA 中,它与腺嘌呤配对,并在 DNA 转录过程中取代胸腺嘧啶。尿嘧啶的甲基化产生胸腺嘧啶。它转化为胸腺嘧啶以保护 DNA 并提高 DNA 复制的效率。尿嘧啶可以与任何碱基配对,这取决于分子在螺旋上的排列方式,但容易与腺嘌呤配对,因为甲基被排斥到固定位置。尿嘧啶通过氢键与腺嘌呤配对。尿嘧啶是氢键受体,可以形成两个氢键。尿嘧啶还可以与核糖结合形成核糖核苷尿苷。当磷酸附着在尿苷上时,就会产生尿苷 5'-单磷酸。
| 含氮碱基 | 核苷 | 脱氧核苷 |
|---|---|---|
 腺嘌呤 |
 腺苷 A |
 脱氧腺苷 dA |
 鸟嘌呤 |
 鸟苷 G |
 脱氧鸟苷 dG |
 胸腺嘧啶 |
 5-甲基尿苷 m5U |
 胸腺嘧啶核苷 dT |
 尿嘧啶 |
 尿苷 U |
 脱氧尿苷 dU |
 胞嘧啶 |
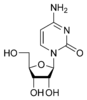 胞嘧啶核苷 C |
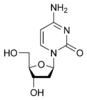 脱氧胞嘧啶核苷 dC |
核苷是糖基胺,由一个核碱基(通常简称为碱基)通过β-糖苷键连接到一个核糖或脱氧核糖糖上。核苷的例子包括胞嘧啶核苷、尿苷、腺苷、鸟苷、胸腺嘧啶核苷和肌苷。核苷可以在细胞中被特异性激酶磷酸化,在糖的伯醇基(-CH2-OH)上产生核苷酸,它是DNA和RNA的分子构建块。
核苷可以通过从头合成途径产生,特别是在肝脏中,但它们主要通过饮食中核酸的摄入和消化来供应,其中核苷酸酶将核苷酸(如胸腺嘧啶核苷酸)分解为核苷(如胸腺嘧啶核苷)和磷酸盐。
1. 腺苷是一种核苷,由一个腺嘌呤分子通过β-N9-糖苷键连接到一个核糖糖分子(核糖呋喃糖)上。
2. 胞嘧啶核苷是一种核苷分子,当胞嘧啶通过β-N1-糖苷键连接到一个核糖环(也称为核糖呋喃糖)上时形成。胞嘧啶核苷是RNA的组成部分。
3. 鸟苷是一种嘌呤核苷,由鸟嘌呤通过β-N9-糖苷键连接到一个核糖(核糖呋喃糖)环上。鸟苷可以被磷酸化,形成鸟苷一磷酸 (GMP)、环鸟苷一磷酸 (cGMP)、鸟苷二磷酸 (GDP) 和鸟苷三磷酸 (GTP)。
4. 胸腺嘧啶核苷(更准确地称为脱氧胸腺嘧啶核苷;也可以标记为脱氧核糖基胸腺嘧啶,和胸腺嘧啶脱氧核苷)是一种化学化合物,更准确地说是嘧啶脱氧核苷。脱氧胸腺嘧啶核苷是DNA核苷T,它在双链DNA中与脱氧腺苷(A)配对。
如果胞嘧啶连接到一个脱氧核糖环上,它被称为脱氧胞嘧啶核苷[9]
一个核苷酸由一个核碱基(含氮碱基)、一个五碳糖(核糖或2'-脱氧核糖)和一个到三个磷酸基团组成。核碱基和糖一起构成一个核苷。磷酸基团与糖的2、3或5-碳形成键,其中5-碳位点最常见。环状核苷酸是在磷酸基团与糖的两个羟基结合时形成的。核糖核苷酸是糖为核糖的核苷酸,脱氧核糖核苷酸含有糖脱氧核糖。核苷酸可以包含嘌呤或嘧啶碱基。核酸是由核苷酸单体组成的聚合大分子。在DNA中,嘌呤碱基是腺嘌呤和鸟嘌呤,而嘧啶是胸腺嘧啶和胞嘧啶。RNA使用尿嘧啶代替胸腺嘧啶。腺嘌呤总是通过2个氢键与胸腺嘧啶配对,而鸟嘌呤通过3个氢键与胞嘧啶配对,每个配对都是由于它们独特的结构。
脱氧核糖核苷酸是DNA或脱氧核糖核酸的单体或单个单元。每个脱氧核糖核苷酸包含三个部分:一个含氮碱基、一个脱氧核糖糖和一个或多个磷酸基团。含氮碱基总是与脱氧核糖的1'碳相连,脱氧核糖与核糖的区别在于2'碳上存在一个质子而不是一个-OH基团。磷酸基团与糖的5'碳相连。当脱氧核糖核苷酸聚合形成DNA时,一个核苷酸的磷酸基团将与另一个核苷酸的3'碳相连,通过脱水合成形成磷酸二酯键。新的核苷酸总是被添加到最后一个核苷酸的3'碳上,因此合成总是从5'到3'进行。[10]
磷酸二酯键
磷酸二酯键是通过两个酯键将一个磷酸基团连接到两个5-碳环碳水化合物(戊糖)之间的一组强共价键。磷酸二酯键对地球上的大多数生命至关重要,因为它们构成了DNA链的骨架。在DNA和RNA中,磷酸二酯键是连接一个糖分子的3'碳原子和另一个糖分子的5'碳原子的连接键,在DNA中为脱氧核糖,在RNA中为核糖。磷酸二酯键中的磷酸基团带负电。因为磷酸基团的pKa接近0,所以在pH 7时它们带负电。这种排斥力迫使磷酸盐占据DNA链的相反侧,并被蛋白质(组蛋白)、金属离子(如镁)和多胺中和。为了形成磷酸二酯键和连接核苷酸,核苷酸构建块的三磷酸或二磷酸形式被分解,释放出驱动酶催化反应所需的能量。当一个单磷酸或两个磷酸(称为焦磷酸盐)脱离并催化反应时,磷酸二酯键就形成。磷酸二酯键的水解可以被磷酸二酯酶的作用催化,磷酸二酯酶在修复DNA序列中起重要作用。在生物系统中,两个核糖核苷酸之间的磷酸二酯键可以被碱性水解破坏,因为存在游离的2'羟基。[11]
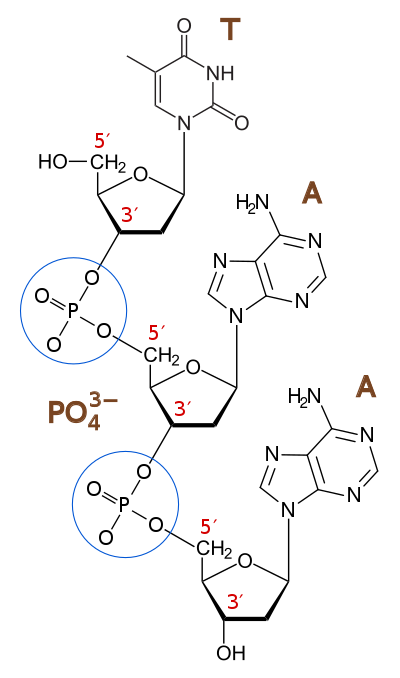
 腺嘌呤一磷酸 AMP |
 腺嘌呤二磷酸 ADP |
 腺嘌呤三磷酸 ATP |
 鸟嘌呤一磷酸 GMP |
 鸟嘌呤二磷酸 GDP |
 鸟嘌呤三磷酸 GTP |
 核糖胸腺嘧啶一磷酸 rTMP |
 核糖胸腺嘧啶二磷酸 rTDP |
 核糖胸腺嘧啶三磷酸 rTTP |
 尿嘧啶一磷酸 UMP |
 尿嘧啶二磷酸 UDP |
 尿嘧啶三磷酸 UTP |
 胞嘧啶一磷酸 CMP |
 胞嘧啶二磷酸 CDP |
 胞嘧啶三磷酸 CTP |
A-DNA: A-DNA是DNA许多可能的双螺旋结构之一。A-DNA被认为是三种生物活性双螺旋结构之一,另外两种是B-DNA和Z-DNA。它是一种右手双螺旋,与更常见且更著名的B-DNA形式非常相似,但螺旋结构更短、更紧凑。它似乎只出现在脱水的DNA样本中,如那些用于晶体学实验的样本,并且可能也存在于DNA-RNA杂交螺旋和双链RNA区域中。[12]
B-DNA最常见的DNA形式是B DNA。DNA双螺旋是核酸的螺旋聚合物,通过碱基配对在一起的核苷酸连接在一起。在B-DNA中,最常见的双螺旋结构,双螺旋是右手螺旋,每圈大约有10-10.5个核苷酸。DNA的双螺旋结构包含一个主沟和一个次沟,主沟比次沟更宽。鉴于主沟和次沟宽度不同,许多与DNA结合的蛋白质都是通过更宽的主沟结合的。
Z-DNA: Z-DNA是DNA许多可能的双螺旋结构之一。它是一种左手双螺旋结构,其中双螺旋以之字形模式向左螺旋(而不是像更常见的B-DNA形式那样向右螺旋)。Z-DNA被认为是三种生物活性双螺旋结构之一,另外两种是A-DNA和B-DNA。Z-DNA与右手形式截然不同。事实上,Z-DNA经常与B-DNA进行比较,以说明主要差异。Z-DNA螺旋是左手螺旋,其结构每2个碱基对重复一次。与A-DNA和B-DNA不同,主沟和次沟的宽度几乎没有差异。这种结构的形成通常是不利的,尽管某些条件可以促进它的形成;例如交替的嘌呤-嘧啶序列(尤其是poly(dGC)2)、负DNA超螺旋或高盐和一些阳离子(所有这些都在生理温度37 °C和pH 7.3-7.4下)。Z-DNA可以与B-DNA形成连接(称为“B-to-Z连接盒”),在一个涉及碱基对挤出的结构中。Z-DNA构象很难研究,因为它不是双螺旋的稳定特征。相反,它是一种瞬态结构,偶尔被生物活性诱导,然后迅速消失。[13]

| A-DNA | B-DNA | Z-DNA | |
|---|---|---|---|
| 螺旋方向 | 右手 | 右手 | 左手 |
| 直径 | 23 Å (2.3 nm) | 20 Å (2.0 nm) | 18 Å (1.8 nm) |
| 重复单元 | 1 bp | 1 bp | 2 bp |
| 每bp旋转 | 32.7° | 35.9° | 60°/2 |
| 每圈bp | 11 | 10.5 | 12 |
| bp相对于轴线的倾斜 | +19° | −1.2° | −9° |
| 沿轴线的每bp上升 | 2.3 Å (0.23 nm) | 3.32 Å (0.332 nm) | 3.8 Å (0.38 nm) |
| 螺旋的节距 | 28.2 Å (2.82 nm) | 33.2 Å (3.32 nm) | 45.6 Å (4.56 nm) |
| 平均螺旋扭曲 | +18° | +16° | 0° |
| 糖苷键角 | 反式 | 反式 | C: 反式, G: 顺式 |
| 糖的皱褶 | C3'-内式 | C2'-内式 | C: C2'-内式, G: C2'-外式 |
bp-碱基对,nm-纳米
在分子生物学中,非编码 DNA 描述了生物体 DNA 序列中不编码蛋白质序列的部分。
假基因 假基因是与已知基因相关的 DNA 序列,它们已经失去了蛋白质编码能力,或者在细胞中不再表达。假基因产生于功能基因的逆转座或基因组复制,并成为“基因组化石”,由于阻止基因转录的突变(如基因启动子区域内的突变)或致命地改变基因翻译的突变(如早停密码子或移码突变)而失去功能。由 RNA 中间体的逆转座产生的假基因被称为加工过的假基因;由复制基因的基因组残留或失活基因残留产生的假基因是非加工过的假基因。虽然 Dollo 法则表明假基因的功能丧失可能是永久性的,但沉默基因实际上可能保留数百万年的功能,并且可以“重新激活”为蛋白质编码序列,并且大量假基因被积极转录。由于假基因被认为是在没有进化约束的情况下进化的,因此它们可以作为各种自发性遗传突变的类型和频率的有效模型。[14]


DNA 超螺旋对于所有细胞中的 DNA 包装很重要。由于 DNA 的长度可以是细胞长度的数千倍,因此将这种遗传物质包装到细胞或细胞核(在真核生物中)是一项艰巨的任务。DNA 的超螺旋减少了空间并允许包装更多的 DNA。在原核生物中,由于环状染色体和相对较少的遗传物质,螺旋状超螺旋占主导地位。在真核生物中,DNA 超螺旋存在于螺旋状和螺线管状超螺旋的多个水平上,其中螺线管状超螺旋在压缩 DNA 方面最为有效。螺线管状超螺旋通过组蛋白实现,形成 10 nm 纤维。这种纤维进一步螺旋成 30 nm 纤维,然后自身再次螺旋多次。在核分裂事件(如有丝分裂或减数分裂)期间,DNA 包装会大大增加,此时 DNA 必须被压缩并分离到子细胞中。凝聚素和粘连素是染色体结构维持蛋白,它们有助于姐妹染色单体的凝聚和姐妹染色单体着丝粒的连接。这些 SMC 蛋白诱导正超螺旋。DNA/RNA 合成也需要超螺旋。由于 DNA 必须解旋才能进行 DNA/RNA 聚合酶作用,因此会产生超螺旋。聚合酶复合体前方的区域将被解旋;这种应力通过复合体前方的正超螺旋来补偿。在复合体后方,DNA 被重新缠绕,并且会有补偿性的负超螺旋。需要注意的是,拓扑异构酶(如 DNA 旋转酶(II 型拓扑异构酶))在 DNA/RNA 合成过程中起着缓解部分应力的作用。[15]
NA 超螺旋可以通过“联结数” Lk 的变化在数值上描述。联结数是超螺旋 DNA 最具描述性的属性。Lko,即松弛(B 型)DNA 质粒/分子的转数,通过将分子的总碱基对除以松弛 bp/转来确定,具体取决于参考值,为 10.4-10.5。
Lk 仅仅是单链在平面投影中穿过另一链的次数。DNA 的拓扑结构由下面的等式描述,其中联结数等效于 TW 的总和,TW 是双螺旋的转数或匝数,以及 Wr,即螺旋数或“缠绕数”。如果存在闭合的 DNA 分子,则 TW 和 Wr 的总和,或联结数,不会改变。但是,TW 和 Wr 可以进行互补变化,而不会改变它们的总和。
联结数的变化 ΔLk 是质粒/分子中的实际转数 Lk 减去松弛质粒/分子 Lko 中的转数。
如果 DNA 是负超螺旋,则 ΔLk < 0。负超螺旋意味着 DNA 被欠缠绕。
一个独立于分子大小的标准表达是“比联结差异”或“超螺旋密度”,用 σ 表示。σ 代表相对于松弛分子/质粒中的总转数添加或移除的转数,表明超螺旋的程度。
联结数是一个数值不变量,描述了三维空间中两条闭合曲线的联结。直观地说,联结数表示每条曲线绕另一条曲线的次数。联结数始终是整数,但根据两条曲线的取向,可以是正数或负数。由于超螺旋 DNA 的联结数 *L* 是两条链相互缠绕的次数(并且两条链都保持共价完整),因此 *L* 不会改变。环状 DNA 双链体的参考状态(或参数) *L0* 是它的松弛状态。在这种状态下,它的缠绕数 *W* = 0。由于 *L = T + W*,在松弛状态下 *T = L*。因此,如果我们有一个 400 bp 松弛的环状 DNA 双链体,则 *L ~ 40*(假设 B-DNA 每圈约 10 bp)。那么 *T ~ 40*。
- 正超螺旋
- T = 0,W = 0,则 L = 0
- T = +3,W = 0,则 L = +3
- T = +2,W = +1,则 L = +3
- 负超螺旋
- T = 0,W = 0,则 L = 0
- T = -3,W = 0,则 L = -3
- T = -2,W = -1,则 L = -3
负超螺旋有利于 DNA 的局部解旋,从而允许进行诸如转录、DNA 复制 和 重组 等过程。负超螺旋也被认为有利于 B-DNA 与 Z-DNA 之间的转变,并调节参与基因调控的 DNA 结合蛋白的相互作用。[17]
组蛋白于 1884 年由 阿尔布雷希特·科塞尔 发现。 “组蛋白”一词源于 19 世纪末,来自德语“Histon”,其起源尚不清楚:可能是来自希腊语 histanai 或 histos。 直到 20 世纪 90 年代初,组蛋白被大多数人认为是真核细胞核 DNA 的惰性包装材料,部分原因是 马克·普塔什内 等人提出的“球棍模型”,他们认为转录是由蛋白质-DNA 和蛋白质-蛋白质相互作用在很大程度上裸露的 DNA 模板上激活的,就像在细菌中一样。 在 20 世纪 80 年代,迈克尔·格伦斯坦 [18] 的工作表明真核组蛋白抑制基因转录,而转录激活因子的功能是克服这种抑制。 我们现在知道组蛋白在基因表达中起着正负两方面的作用,构成了组蛋白密码的基础。
H5 组蛋白的发现似乎可以追溯到 20 世纪 70 年代,[19][20] 在分类中它被归类为 核小体核心由两个 H2A-H2B 二聚体和一个 H3-H4 四聚体组成,通过三级结构形成两个几乎对称的半部分(C2 对称性;一个大分子是另一个的镜像)。H2A-H2B 二聚体和 H3-H4 四聚体也表现出假二元对称性。 4 个“核心”组蛋白(H2A、H2B、H3 和 H4)在结构上相对相似,并且在进化过程中高度保守,所有都具有“螺旋转螺旋转螺旋”基序(这允许轻松二聚化)。 它们还共有一个特征,即在氨基酸结构一端有长“尾”——这是翻译后修饰的位置(见下文)。

有人提出组蛋白在进化上与扩展 AAA+ ATPase 结构域的螺旋部分、C 结构域以及 Clp/Hsp100 蛋白的 N 端底物识别结构域相关。 尽管它们在拓扑结构上存在差异,但这三种折叠共享一个同源螺旋-链-螺旋 (HSH) 基序。
英国研究人员使用电子顺磁共振自旋标记技术测量了真核细胞在其周围缠绕 DNA 的线轴之间的距离。 他们确定间距范围为 59 到 70 Å。 总之,组蛋白与 DNA 进行五种类型的相互作用
来自 H2B、H3 和 H4 中α-螺旋的螺旋偶极子导致净正电荷在与 DNA 上带负电荷的磷酸基团相互作用的点处积累
DNA 骨架与组蛋白中主链上的酰胺基团之间的氢键
组蛋白与 DNA 上的脱氧核糖糖之间的非极性相互作用
碱性氨基酸(特别是赖氨酸和精氨酸)侧链与 DNA 上的磷酸氧之间的盐桥和氢键
H3 和 H2B N 端尾部进入 DNA 分子上两个小沟的非特异性小沟插入
组蛋白的高度碱性性质除了促进 DNA-组蛋白相互作用外,还有助于组蛋白的水溶性。 组蛋白主要在其 N 端尾部,但也在其球状结构域中受到酶的翻译后修饰。 这些修饰包括甲基化、瓜氨酸化、乙酰化、磷酸化、SUMO 化、泛素化和 ADP 核糖基化。 这会影响它们对基因调控的功能。 一般来说,活跃的基因结合的组蛋白较少,而非活跃基因在间期与组蛋白高度相关。 组蛋白的结构似乎也已在进化上得到保存,因为任何有害的突变都会严重不利。
核心组蛋白包含一个称为“组蛋白折叠”的特征性结构基序,该基序由三个α-螺旋(α1-3)组成,它们由两个环(L1-2)隔开。 在溶液中,组蛋白形成 H2A-H2B 异二聚体和 H3-H4 异四聚体。 组蛋白在其长的α2螺旋周围以反平行方向二聚化,在 H3 和 H4 的情况下,两个这样的二聚体形成了一个由广泛的 H3-H3’相互作用稳定的 4 螺旋束。 H2A/H2B 二聚体由于 H4 和 H2B 之间的相互作用而结合到 H3/H4 四聚体上,包括疏水簇的形成。 组蛋白八聚体由夹在两个 H2A/H2B 二聚体之间的中心 H3/H4 四聚体形成。 由于所有四个核心组蛋白都具有高度碱性电荷,组蛋白八聚体仅在存在 DNA 或非常高的盐浓度的情况下稳定。
核小体构成真核染色质的基本重复单元,用于将大型真核基因组包装到细胞核中,同时仍然确保对其的适当访问(在哺乳动物细胞中,大约 2 米的线性 DNA 必须包装到大约 10 微米直径的细胞核中)。 核小体通过一系列不断增加的高阶结构折叠,最终形成染色体; 这既压缩了 DNA,又创建了一个额外的调控层,确保基因表达正确。 核小体被认为以核心组蛋白的共价修饰形式携带表观遗传继承的信息。 核小体假说由 Don 和 Ada Olins 于 1974 年提出,以及 Roger Kornberg。
核小体核心颗粒)由大约 146 bp 的 DNA 组成,围绕组蛋白八聚体(由核心组蛋白 H2A、H2B、H3 和 H4 的 2 个拷贝组成)以 1.67 个左手超螺旋周数缠绕。 相邻的核小体由一段称为“连接 DNA”的游离 DNA 连接(长度因物种和组织类型而异,从 10 到 80 bp 不等)。

一个或多个 DNA 结合域通常是包含具有不同功能的额外结构域的更大蛋白质的一部分。 额外的结构域通常调节 DNA 结合域的活性。 DNA 结合的功能要么是结构性的,要么涉及转录调控,这两种作用有时会重叠。 具有涉及 DNA 结构功能的 DNA 结合域在 DNA 的复制、修复、储存和修饰(例如甲基化)中具有生物学作用。 许多参与基因表达调控的蛋白质包含 DNA 结合域。 例如,通过结合 DNA 调节转录的蛋白质称为转录因子。 大多数细胞信号级联的最终输出是基因调控。 DBD 以 DNA 序列特异性或非序列特异性方式与 DNA 的核苷酸相互作用,但即使是非序列特异性识别也涉及蛋白质和 DNA 之间的某种分子互补性。 DBD 对 DNA 的识别可以发生在 DNA 的大沟或小沟,或发生在糖磷酸 DNA 骨架上(参见 DNA 的结构)。 每种特定类型的 DNA 识别都针对蛋白质的功能而定制。 例如,DNA 切割酶 DNAse I 几乎随机切割 DNA,因此必须以非序列特异性方式结合 DNA。 但是,即使如此,DNAse I 也识别特定 3-D DNA 结构,从而产生某种特定的 DNA 切割模式,这对于通过称为 DNA 足迹法的技术研究 DNA 识别很有用。 许多 DNA 结合域必须识别特定的 DNA 序列,例如激活特定基因的转录因子的 DBD,或在特定位点修饰 DNA 的酶的 DBD,如限制性内切酶和端粒酶。 DNA 大沟中的氢键模式比 DNA 小沟中的氢键模式退化程度更低,为序列特异性 DNA 识别提供了更具吸引力的位点。可以使用许多生化和生物物理技术来研究 DNA 结合蛋白的特异性,例如凝胶电泳、分析超速离心、量热法、DNA 突变、蛋白质结构突变或修饰、核磁共振、X 射线晶体学、表面等离子共振、电子顺磁共振、交联和微量热泳动 (MST)。[21]
DNA 结合域的类型
螺旋-转角-螺旋
最初在细菌中发现,螺旋-转角-螺旋基序通常存在于阻遏蛋白中,长度约为 20 个氨基酸。 在真核生物中,同源域包含 2 个螺旋,其中一个识别 DNA(又名识别螺旋)。 它们在调节发育过程的蛋白质中很常见(PROSITE HTH)。[22]
锌指
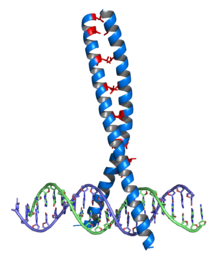
糖皮质激素受体(上)的锌指结构域(DBD)二聚体与DNA(下)结合的晶体结构(PDB 1R4O)。锌原子用灰色球体表示,配位的半胱氨酸侧链用棒状表示。锌指这个结构域通常有23到28个氨基酸长,通过与规则间隔的锌配位残基(组氨酸或半胱氨酸)配位锌离子而稳定。最常见的锌指类型(Cys2His2)配位一个锌离子,由一个识别螺旋和一个2链β折叠组成。在转录因子中,这些结构域通常成串排列(通常由短连接序列隔开),相邻的指在与DNA结合时间隔3个碱基对。

| 折叠组 | 代表性结构 | 配体放置 |
|---|---|---|
| Cys2His2 |  |
两个配体来自一个指节,另外两个来自螺旋的C端。 |
| Gag指节 |  |
两个配体来自一个指节,另外两个来自一个短螺旋或环。 |
| 高音谱号 | 两个配体来自一个指节,另外两个来自螺旋的N端。 | |
| 锌带 |  |
两个指节分别提供两个配体。 |
| Zn2/Cys6 |  |
两个配体来自螺旋的N端,另外两个来自一个环。 |
| TAZ2结构域样 | 两个配体来自两个螺旋的末端。 |
亮氨酸拉链
碱性亮氨酸拉链(bZIP)结构域包含一个α螺旋,每7个氨基酸有一个亮氨酸。如果两个这样的螺旋相互靠近,亮氨酸就可以像拉链的齿一样相互作用,使两个蛋白质二聚化。在与DNA结合时,碱性氨基酸残基与糖磷酸骨架结合,而螺旋则位于主沟中。它调节基因表达。bZip家族的转录因子包含一个通过氢键与DNA分子主沟相互作用的碱性区域,以及一个负责二聚化的疏水亮氨酸拉链区域。
翼螺旋
翼螺旋(WH)结构域包含大约110个氨基酸,具有四个螺旋和一个两链β折叠。
翼螺旋转螺旋 翼螺旋转螺旋结构域(wHTH)SCOP 46785 通常有85-90个氨基酸长。它由一个3螺旋束和一个4链β折叠(翼)组成。
螺旋-环-螺旋
螺旋-环-螺旋结构域存在于一些转录因子中,其特征是两个由环连接的α螺旋。一个螺旋通常较小,由于环的柔性,允许通过折叠和包装到另一个螺旋上而二聚化。较大的螺旋通常包含DNA结合区域。
HMG盒
HMG盒结构域存在于高迁移率基团蛋白中,这些蛋白参与各种依赖DNA的过程,如复制和转录。该结构域由三个由环隔开的α螺旋组成。
DNA测序
[edit | edit source]RNA测序是最早的核苷酸测序形式之一。RNA测序的主要里程碑是第一个完整基因的序列和噬菌体MS2的完整基因组的序列,由Walter Fiers及其在根特大学(比利时根特)的同事在1972年至1976年间确定并发表。在20世纪70年代初Frederick Sanger在英国剑桥大学以及Walter Gilbert和Allan Maxam在哈佛大学开发出快速DNA测序方法之前,曾使用过一些繁琐的方法。例如,1973年,Gilbert和Maxam使用一种称为“漫游斑点分析”的方法报道了24个碱基对的序列。Sanger及其同事在1975年开发的链终止法很快成为首选方法,因为它相对容易且可靠。[23]
Maxam和Gilbert方法
[edit | edit source]1976-1977年,Allan Maxam和Walter Gilbert开发了一种基于DNA化学修饰和随后在特定碱基处裂解的DNA测序方法。虽然Maxam和Gilbert在Sanger和Coulson关于加减测序的开创性论文发表两年后才发表了他们的化学测序方法,但Maxam-Gilbert测序很快就变得更加流行,因为可以直接使用纯化的DNA,而最初的Sanger方法则要求将每个读取的起点克隆以便产生单链DNA。然而,随着链终止方法的改进(见下文),Maxam-Gilbert测序已经不受欢迎,因为它技术复杂,禁止其在标准分子生物学试剂盒中使用,大量使用危险化学品以及难以扩展。该方法需要在DNA的一个5'端进行放射性标记(通常通过使用γ-32P ATP进行激酶反应)并纯化要测序的DNA片段。化学处理会在四个反应(G、A+G、C、C+T)中的一到两个碱基中的一小部分产生断裂。例如,嘌呤(A+G)使用甲酸进行脱嘌呤,鸟嘌呤(以及在某种程度上腺嘌呤)使用硫酸二甲酯进行甲基化,嘧啶(C+T)使用肼进行甲基化。在肼反应中加入盐(氯化钠)会抑制胸腺嘧啶的甲基化,从而进行C-only反应。然后,修饰的DNA在修饰碱基的位置用热哌啶裂解。修饰化学品的浓度控制在平均每个DNA分子引入一个修饰。因此,会产生一系列标记片段,从放射性标记的末端到每个分子中的第一个“切割”位点。在四个反应中的片段在变性丙烯酰胺凝胶中并排进行电泳,以进行大小分离。为了可视化片段,将凝胶暴露于X射线胶片上进行放射自显影,产生一系列深色条带,每个条带对应于一个放射性标记的DNA片段,可以从中推断出序列。该方法也称为“化学测序”,它导致了用于绘制DNA结合蛋白的DNA结合位点的甲基化干扰分析。[24]
双脱氧核苷酸链终止法
[edit | edit source]
由于链终止法(或以其开发者[Frederick Sanger](/w/index.php?title=Frederick_Sanger&action=edit&redlink=1" class="new" title="Frederick Sanger (does not exist)">命名的Sanger方法)比Maxam和Gilbert的方法效率更高,使用的有毒化学品更少,放射性物质也更少,因此它迅速成为首选方法。Sanger方法的关键原理是使用双脱氧核苷酸三磷酸(ddNTP)作为DNA链终止剂。
经典的链终止法需要单链DNA模板、DNA引物、DNA聚合酶、正常的脱氧核苷酸磷酸(dNTP)以及修饰的核苷酸(双脱氧核苷酸),这些核苷酸会终止DNA链的延伸。这些ddNTP也会进行放射性或荧光标记,以便在自动测序仪中检测。DNA样本被分成四个独立的测序反应,每个反应包含所有四个标准脱氧核苷酸(dATP、dGTP、dCTP和dTTP)以及DNA聚合酶。在每个反应中只添加四个双脱氧核苷酸中的一个(ddATP、ddGTP、ddCTP或ddTTP),它们是链终止核苷酸,缺少形成两个核苷酸之间磷酸二酯键所需的3'-羟基(OH)基团,从而终止DNA链的延伸,并产生不同长度的DNA片段。
新合成的标记DNA片段被热变性,并通过大小(分辨率仅为一个核苷酸)在变性丙烯酰胺-尿素凝胶上进行电泳,四个反应中的每个反应都在四个独立的泳道(泳道A、T、G、C)中进行;然后通过放射自显影或紫外光可视化DNA条带,可以直接从X射线胶片或凝胶图像上读出DNA序列。在右侧的图像中,X射线胶片暴露于凝胶,深色条带对应于不同长度的DNA片段。泳道中的深色条带表示一个DNA片段,该片段是在掺入双脱氧核苷酸(ddATP、ddGTP、ddCTP或ddTTP)后链终止的结果。然后使用四个泳道中不同条带的相对位置(从下到上)读出DNA序列。[25]

链终止测序的技术变异包括使用包含放射性磷的核苷酸进行放射性标记,或使用在5'端用荧光染料标记的引物。染料引物测序便于在光学系统中读取,从而实现更快、更经济的分析和自动化。[Leroy Hood](/w/index.php?title=Leroy_Hood&action=edit&redlink=1" class="new" title="Leroy Hood (does not exist)">及其同事[26][27]后来开发的荧光标记的ddNTP和引物为自动化、高通量DNA测序奠定了基础。

链终止法极大地简化了DNA测序。例如,市售的基于链终止的试剂盒包含测序所需的试剂,预先分装并可以使用。局限性包括引物与DNA的非特异性结合,影响DNA序列的准确读出,以及DNA二级结构影响序列的保真度。

染料终止子测序利用链终止剂 ddNTP 的标记,使得测序可以在单一反应中完成,而不是像标记引物法那样需要四种反应。在染料终止子测序中,四种双脱氧核苷酸链终止剂中的每一种都被标记上荧光染料,每种染料都在不同的波长发射光。
由于其更高的便利性和速度,染料终止子测序现在已成为自动化测序的主流。其局限性包括由于染料标记的链终止剂掺入 DNA 片段的差异而产生的染料效应,导致电子 DNA 序列跟踪 色谱图 中峰高和峰形不等 毛细管电泳(见左侧图)。
这个问题已通过使用改进的 DNA 聚合酶酶系统和最大限度地减少掺入差异的染料以及消除“染料斑点”的方法得到解决。染料终止子测序方法与自动化高通量 DNA 序列分析仪一起,现在正被用于绝大多数测序项目。
DNA 测序的常见挑战包括序列前 15-40 个碱基的质量差以及 700-900 个碱基后测序跟踪质量下降。 碱基识别 软件通常会提供质量估计,以帮助进行质量修剪。[28][29]
在 DNA 片段在测序之前被克隆的情况下,得到的序列可能包含克隆载体的部分。相反,基于 PCR 的克隆和新兴的基于焦磷酸测序的测序技术通常避免使用克隆载体。最近,已经开发出单步桑格测序(结合扩增和测序)方法,如 Ampliseq 和 SeqSharp,这些方法允许快速测序目标基因,而无需克隆或事先扩增。[30][31]
目前的方法只能直接测序相对较短的(300-1000 核苷酸 长)DNA 片段。在单一反应中测序超过此尺寸限制的 DNA 片段的主要障碍是,对于长度仅相差一个核苷酸的大 DNA 片段,分离能力不足以进行分辨。在所有情况下,使用具有游离 5' 末端的引物都是必不可少的。

自动化 DNA 测序仪(DNA 测序仪)可以一次批次(运行)测序多达 384 个 DNA 样本,每天最多可运行 24 次。DNA 测序仪进行毛细管电泳以进行尺寸分离、检测和记录染料荧光,并将数据输出为荧光峰跟踪色谱图。通过热循环进行测序反应,在加载到测序仪上之前进行清理和重新悬浮在缓冲溶液中。许多商业和非商业软件包可以自动修剪低质量 DNA 跟踪。这些程序会对每个峰的质量进行评分,并删除低质量的碱基峰(通常位于序列的末端)。这种算法的准确性低于人工操作员的视觉检查,但足以进行大型序列数据集的自动化处理。

PCR
PCR 用于扩增 DNA 链的特定区域(DNA 目标)。大多数 PCR 方法通常扩增高达 ~10 千碱基对 (kb) 的 DNA 片段,尽管一些技术允许扩增高达 40 kb 的片段。基本的 PCR 设置需要几个组分和试剂。这些组分包括
包含要扩增的 DNA 区域(目标)的 DNA 模板。
两个引物,它们与 DNA 目标的正义链和反义链的 3'(三素)末端互补。Taq 聚合酶或其他 DNA 聚合酶,其最佳温度在 70 °C 左右。脱氧核苷酸三磷酸 (dNTP),DNA 聚合酶合成新的 DNA 链的构建块。缓冲溶液,为 DNA 聚合酶的最佳活性提供合适的化学环境。二价阳离子,镁或锰离子;通常使用 Mg2+,但 Mn2+ 可用于 PCR 介导的 DNA 诱变,因为更高的 Mn2+ 浓度会增加 DNA 合成过程中的错误率。一价阳离子钾离子。PCR 通常在热循环仪中,在 10-200 μl 的反应体积内,在小反应管(0.2-0.5 ml 体积)中进行。热循环仪对反应管进行加热和冷却,以在反应的每个步骤中实现所需的温度(见下文)。许多现代热循环仪利用珀耳帖效应,通过简单地反转电流来实现容纳 PCR 管的模块的加热和冷却。薄壁反应管允许良好的热传导,从而实现快速热平衡。大多数热循环仪都具有加热盖,以防止反应管顶部的冷凝。缺乏加热盖的旧热循环仪需要在反应混合物顶部添加一层油或在管内添加一块蜡。[32]
程序
图 1:PCR 循环示意图。 (1) 在 94–96 °C 下变性。 (2) 在 ~65 °C 下退火 (3) 在 72 °C 下延伸。 此处显示了四个循环。 蓝色线表示 DNA 模板,引物(红色箭头)与其退火,并由 DNA 聚合酶(浅绿色圆圈)延伸,以产生较短的 DNA 产物(绿色线),这些产物本身在 PCR 过程中被用作模板。 通常,PCR 包含一系列 20-40 次重复的温度变化,称为循环,每个循环通常包含 2-3 个不同的温度步骤,通常为三个。 循环通常以在高温 (>90 °C) 下的单个温度步骤(称为保持)开始,并在最后以一个保持步骤结束,用于最终产物延伸或短暂储存。 所使用的温度以及它们在每个循环中应用的时间取决于多种参数。 这些参数包括用于 DNA 合成的酶、反应中二价离子和 dNTP 的浓度以及引物的熔解温度 (Tm)。 初始化步骤:此步骤包括将反应加热到 94–96 °C(或如果使用极耐热的聚合酶则为 98 °C),并保持 1–9 分钟。 它仅适用于需要通过热启动 PCR 进行热激活的 DNA 聚合酶。 变性步骤:此步骤是第一个常规循环事件,包括将反应加热到 94–98 °C,持续 20–30 秒。 它通过破坏互补碱基之间氢键引起 DNA 模板的 DNA 熔化,产生单链 DNA 分子。 退火步骤:反应温度降低到 50–65 °C,持续 20–40 秒,允许引物退火到单链 DNA 模板。 通常,退火温度比所用引物的 Tm 低约 3-5 摄氏度。 稳定的 DNA-DNA 氢键仅在引物序列与模板序列非常匹配时形成。 聚合酶结合到引物-模板杂交体并开始 DNA 合成。 延伸/延长步骤:此步骤的温度取决于所使用的 DNA 聚合酶;Taq 聚合酶的最佳活性温度为 75–80 °C,通常在这种酶的情况下使用 72 °C 的温度。 在此步骤中,DNA 聚合酶通过添加与模板互补的 dNTP,以 5' 到 3' 的方向合成与 DNA 模板链互补的新 DNA 链,将 dNTP 的 5'-磷酸基团与新生(延伸)DNA 链末端的 3'-羟基团缩合。 延伸时间既取决于所使用的 DNA 聚合酶,也取决于要扩增的 DNA 片段的长度。 作为经验法则,在最佳温度下,DNA 聚合酶每分钟会聚合一千个碱基。 在最佳条件下,即如果没有由于限制性底物或试剂引起的限制,在每个延伸步骤中,DNA 目标的数量都会翻倍,导致特定 DNA 片段的指数(几何)扩增。 最终延伸:此单个步骤偶尔在最后一个 PCR 循环后以 70–74 °C 的温度进行 5–15 分钟,以确保任何剩余的单链 DNA 能够完全延伸。 最终保持:此步骤在 4–15 °C 的温度下进行无限时间,可用于对反应进行短期储存。
为了检查 PCR 是否产生了预期的 DNA 片段(有时也称为扩增子或扩增片段),采用琼脂糖凝胶电泳来分离 PCR 产物的大小。 PCR 产物的大小通过与 DNA 梯(分子量标记)进行比较来确定,DNA 梯包含已知大小的 DNA 片段,与 PCR 产物一起在凝胶上运行。
需要记住的事实
[edit | edit source]DNA 聚合酶是催化从核苷三磷酸合成多核苷酸链并制造 DNA 的酶。 1865 年,格雷戈尔·孟德尔的论文《植物杂交实验》
1869 年,瑞士医生弗里德里希·米歇尔首次分离出 DNA,他在废弃的绷带的脓液中发现了一种显微镜下可见的物质。
从 1880 年到 1890 年,沃尔特·弗莱明、爱德华·施特拉斯伯格和埃德华·范·贝内登阐明了细胞分裂过程中染色体的分布。
1889 年,休戈·德·弗里斯假设“生物体中特定性状的遗传是以颗粒的形式存在的”,并称这些颗粒为“(泛)基因”。
1903 年,沃尔特·萨顿假设以孟德尔方式分离的染色体是遗传单位。
1905 年,威廉·贝特森在一封给亚当·塞奇威克的信中和 1906 年的一次会议上创造了“遗传学”一词。
1908 年,推导出哈代-温伯格定律。
1910 年,托马斯·亨特·摩根证明基因位于染色体上。
1913 年,阿尔弗雷德·斯特蒂文特制作了第一张染色体遗传图。
1913 年,基因图谱显示染色体包含线性排列的基因。
1918 年,罗纳德·费舍尔发表了《关于孟德尔遗传假设下的亲属之间的相关性》一书,现代遗传学与进化生物学合成开始。 请参见群体遗传学。
1928 年,弗雷德里克·格里菲斯发现,来自死亡细菌的遗传物质可以整合到活细菌中(请参见格里菲斯实验)。
1931 年,交叉被确定为重组的原因。
1933 年,让·布拉谢特能够证明 DNA 存在于染色体中,而 RNA 存在于所有细胞的细胞质中。
1937 年,威廉·阿斯特伯里产生了第一个 X 射线衍射图谱,显示 DNA 具有规则的结构。
1928 年,弗雷德里克·格里菲斯发现,肺炎链球菌的“光滑”形式的性状可以通过将杀死的“光滑”细菌与活的“粗糙”形式混合来转移到相同细菌的“粗糙”形式。
1952 年,阿尔弗雷德·赫希和玛莎·蔡斯在赫希-蔡斯实验中表明,DNA 是 T2 噬菌体的遗传物质。
1953 年,詹姆斯·D·沃森和弗朗西斯·克里克提出了 DNA 结构的双螺旋模型。
嘌呤在肉类和肉制品中含量很高,尤其是在肝脏和肾脏等内脏中。
高嘌呤来源的示例包括:胸腺、凤尾鱼、沙丁鱼、肝脏、牛肉肾脏、脑、肉汁(例如 Oxo、Bovril)、鲱鱼、鲭鱼、扇贝、野味、啤酒(来自酵母)和肉汁。
bp = 碱基对。 一个 bp 对应于沿链约 3.4 Å 的长度。
kb (= kbp) = 千碱基对 = 1,000 bp
Mb = 兆碱基对 = 1,000,000 bp
DNA 拓扑结构分析使用三个值
L = 连接数 - 一条 DNA 链绕另一条 DNA 链缠绕的次数。 对于闭合环,它是整数,对于闭合拓扑域,它是常数。
T = 扭曲 - 双链 DNA 螺旋中总的转数。 通常,这将趋向于接近在溶液中自由形成的拓扑开放双链 DNA 螺旋的转数:碱基数/10.5,假设没有嵌入剂(例如,氯喹)或其他改变 DNA 刚度的元素。
W = 缠绕 - 双链 DNA 螺旋绕超螺旋轴缠绕的次数
L = T + W 且 ΔL = ΔT + ΔW
在闭合拓扑域中,T 的任何变化都必须由 W 的变化来平衡,反之亦然。 这导致 DNA 的更高阶结构。 具有 0 缠绕的环状 DNA 分子将是圆形的。 如果该分子的扭曲随后通过超螺旋而增加或减少,则缠绕将相应地改变,使分子经历曲折或环状超螺旋缠绕。 当一段双链螺旋 DNA 的末端连接在一起形成一个圆时,链条在拓扑上是打结的。 这意味着单链无法通过任何不涉及断裂链条(例如加热)的过程来分离。 解开拓扑连接的 DNA 链的任务落到了称为拓扑异构酶的酶上。 这些酶专门通过切割一条或两条链来解开环状 DNA,以便另一段双链或单链可以通过。 这种解开对于环状 DNA 的复制以及具有类似拓扑约束的线性 DNA 中的各种类型的重组是必需的。
Gb = 吉碱基对 = 1,000,000,000 bp。
1972年,重组DNA技术的开发允许分离出明确的DNA片段;在此之前,唯一可用于测序的样本来自噬菌体或病毒DNA。 1977年,第一个完整测序的DNA基因组是噬菌体φX174。 1977年,Allan Maxam和Walter Gilbert发表了“化学降解法测序DNA”。 同时,Frederick Sanger独立发表了“利用链终止抑制剂测序DNA”。 1984年,英国医学研究委员会的科学家破译了170kb的EB病毒的完整DNA序列。 1986年,加州理工学院Leroy E. Hood实验室和Smith宣布了第一个半自动化的DNA测序仪。 1987年,应用生物系统公司推出了第一台自动测序仪,型号为ABI 370。 1990年,美国国立卫生研究院(NIH)开始对支原体(Mycoplasma capricolum)、大肠杆菌(Escherichia coli)、秀丽隐杆线虫(Caenorhabditis elegans)和酿酒酵母(Saccharomyces cerevisiae)进行大规模测序试验(每碱基0.75美元)。 1991年,Craig Venter实验室开始对人类表达序列标签进行测序,试图捕捉人类基因组的编码部分。 1995年,Craig Venter、Hamilton Smith和基因组研究所(TIGR)的同事发表了第一个自由生活生物体的完整基因组,即嗜血杆菌(Haemophilus influenzae)。 环状染色体包含1,830,137个碱基,其发表在《科学》杂志上标志着首次使用全基因组鸟枪法测序,消除了对初始作图工作的需求。 1996年,瑞典皇家理工学院的Pål Nyrén和他的学生Mostafa Ronaghi发表了他们的焦磷酸测序方法。 1998年,华盛顿大学的Phil Green和Brent Ewing发布了用于测序数据分析的“phred”软件。 2001年,人类基因组的草图序列发表。 2004年,454 Life Sciences公司推出了焦磷酸测序的并行版本。 他们机器的第一版与自动化的Sanger测序相比,测序成本降低了6倍,是继MPSS之后,新一代测序技术的第二种。
DNA和RNA中发现的碱基列表
| 名称 | 3D结构 | 缩写 | 结构式 | 分类 | 发现于 |
|---|---|---|---|---|---|
 |
 |
嘧啶 | DNA,RNA | ||
 |
 |
嘧啶 | DNA | ||
 |
 |
嘧啶 | RNA | ||
 |
 |
嘌呤 | DNA,RNA | ||
 |
 |
嘌呤 | DNA,RNA |
- ↑ DNA
- ↑ 格里菲斯实验
- ↑ 赫希-蔡斯实验
- ↑ Hershey, A.D. and Chase, M. (1952) 噬菌体生长中病毒蛋白和核酸的独立功能。J Gen Physiol. 36:39–56.
- ↑ 艾弗里-麦克莱德-麦卡锡实验
- ↑ 碱基对
- ↑ 嘧啶
- ↑ 胞嘧啶
- ↑ 核苷
- ↑ 核苷酸
- ↑ 磷酸二酯键
- ↑ A-DNA
- ↑ Z-DNA
- ↑ 非编码DNA
- ↑ DNA超螺旋
- ↑ Vologodskii AV,Lukashin AV,Anshelevich VV 等。 (1979). “超螺旋DNA的波动”。核酸研究. 6: 967–682. doi:10.1093/nar/6.3.967.
{{引用期刊}}: 在“|作者=”中显式使用“等”。 (帮助)CS1维护:作者列表中的多个名称 (链接) - ↑ H. S. Chawla (2002). 植物生物技术导论. 科学出版社. ISBN 1578082285.
- ↑ Kayne PS, Kim UJ, Han M, Mullen JR, Yoshizaki F, Grunstein M. 酵母中高度保守的组蛋白H4 N端对于生长是可有可无的,但对于抑制沉默的交配位点是必不可少的。细胞. 1988年10月7日;55(1):27-39. PMID 3048701
- ↑ Crane-Robinson C,Dancy SE,Bradbury EM,Garel A,Kovacs AM,Champagne M,Daune M (1976). “鸡红细胞组蛋白H5的结构研究”。欧洲生物化学杂志. 67 (2): 379–88. doi:10.1111/j.1432-1033.1976.tb10702.x. PMID 964248.
{{引用期刊}}: 未知参数“|月=”被忽略 (帮助)CS1维护:作者列表中的多个名称 (链接) - ↑ Aviles FJ,Chapman GE,Kneale GG,Crane-Robinson C,Bradbury EM (1978). “组蛋白H5的构象。球状片段的隔离和表征”。欧洲生物化学杂志. 88 (2): 363–71. doi:10.1111/j.1432-1033.1978.tb12457.x. PMID 689022.
{{引用期刊}}: 未知参数“|月=”被忽略 (帮助)CS1维护:作者列表中的多个名称 (链接) - ↑ DNA结合域
- ↑ DNA结合域
- ↑ DNA测序
- ↑ DNA测序
- ↑ DNA测序
- ↑ Smith LM, Sanders JZ, Kaiser RJ 等人。 (1986). “自动化 DNA 测序分析中的荧光检测”。自然. 321 (6071): 674–9. doi:10.1038/321674a0. PMID 3713851.
我们开发了一种用于 DNA 测序分析部分自动化的方法。 DNA 片段的荧光检测是通过将荧光团共价连接到用于酶促 DNA 测序分析的寡核苷酸引物来实现的。 每个特定于碱基 A、C、G 和 T 的反应使用不同的彩色荧光团。 反应混合物被组合并共同电泳通过单个聚丙烯酰胺凝胶管,分离的 DNA 荧光带在管底部附近被检测到,并且序列信息直接由计算机获取。
{{cite journal}}: 在|author=中明确使用 et al. (help)CS1 维护:作者列表的多个名称 (link) - ↑ Smith LM, Fung S, Hunkapiller MW, Hunkapiller TJ, Hood LE (1985). "合成在 5' 末端含有脂肪族氨基的寡核苷酸:合成用于 DNA 测序分析的荧光 DNA 引物". 核酸研究. 13 (7): 2399–412. doi:10.1093/nar/13.7.2399. PMC 341163. PMID 4000959.
{{cite journal}}: 未知参数|month=被忽略 (help)CS1 维护:作者列表的多个名称 (link) - ↑ "Phred - 质量碱基调用". 检索于 2011-02-24.
- ↑ "下一代测序平台的碱基调用 - 简短生物信息学". 检索于 2011-02-24.
- ↑ Murphy, K.;Berg, K.;Eshleman, J. (2005). “通过组合扩增和循环测序反应对基因组 DNA 进行测序”。临床化学 51 (1): 35–39。
- ↑ Sengupta, D.;Cookson, B. (2010). “SeqSharp:一种用于改进循环测序的通用方法,它促进稳健的一步组合扩增和测序方法”。分子诊断杂志:JMD 12 (3): 272–277。
- ↑ 聚合酶链式反应









