分子生物学/基因表达简介
基因表达是指基因中的信息被用来合成功能性基因产物的过程。这些产物通常是蛋白质,但在非蛋白质编码基因(如核糖体RNA(rRNA)基因或转移RNA(tRNA)基因)中,产物是功能性RNA。基因表达过程被所有已知生命形式使用 - 真核生物(包括多细胞生物)、原核生物(细菌和古细菌)和病毒 - 来产生维持生命的生物大分子机制。基因表达过程中的几个步骤可能会受到调节,包括蛋白质的转录、RNA剪接、翻译和翻译后修饰。基因调控使细胞能够控制其结构和功能,并且是细胞分化、形态发生以及任何生物体灵活性和适应性的基础。基因调控也可能作为进化变化的基质,因为对基因表达时间、位置和量的控制可以对基因在细胞或多细胞生物体中的功能(作用)产生深远的影响。在遗传学中,基因表达是基因型产生表型的最基本层次。以核苷酸序列形式存储在 DNA 中的遗传密码通过基因表达被“解释”,而表达产物的特性导致了生物体的表型。[1] 最初由弗朗索瓦·雅各布和雅克·莫诺提出,允许遗传物质被实现为蛋白质的分子。1965年,几个实验室在体外建立了RNA聚合酶的RNA合成;然而,这些酶合成的RNA具有表明存在一个额外的因子,需要正确终止转录。1972年,沃尔特·菲尔斯成为第一个真正证明终止酶存在的科学家。罗杰·D·科恩伯格因“他对真核生物转录的分子基础的研究”获得2006年诺贝尔化学奖。
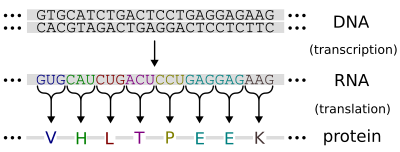
从DNA生成RNA被称为转录。换句话说,转录是创建DNA序列的互补RNA副本的过程。在转录过程中,DNA序列被RNA聚合酶读取,它产生一个互补的反向RNA链。与DNA复制相反,转录产生的RNA互补链包含尿嘧啶(U),在DNA互补链中所有出现胸腺嘧啶(T)的地方都会出现尿嘧啶(U)。[2] 转录可以通过4或5个简单的步骤来轻松解释,每个步骤就像波浪一样沿着DNA移动。
As the Hydrogen Bonds Break DNA unwinds. The free nucleotides of the RNA, pair with complementary DNA bases. RNA sugar-phosphate backbone forms. (by RNA Polymerase.) Hydrogen bonds of the untwisted RNA-DNA "ladder" break, freeing the new RNA. The RNA is further processed and then moves through the small nuclear pores to the cytoplasm.
转录是导致基因表达的第一步。转录成RNA分子的DNA片段被称为转录单位,它至少编码一个基因。如果被转录的基因编码蛋白质,那么转录的结果是信使RNA(mRNA),然后将通过翻译过程被用来创建该蛋白质。或者,被转录的基因可能编码核糖体RNA(rRNA)或转移RNA(tRNA),这是蛋白质组装过程的其他组分,或其他核酶。
编码蛋白质的DNA转录单位不仅包含最终将直接翻译成蛋白质的序列(编码序列),还包含指导和调节该蛋白质合成的调控序列。编码序列之前的调控序列(上游)被称为5'UTR(5'非翻译区),编码序列之后的序列(下游)被称为3'UTR(3'非翻译区)。转录具有一些校对机制,但这些机制少于且不如复制DNA的控制机制有效;因此,转录的复制保真度低于DNA复制。与DNA复制一样,DNA在转录过程中从3'→5'方向读取。同时,互补RNA从5'→3'方向创建。这意味着它的5'端首先在碱基配对中被创建。虽然DNA以双螺旋形式排列成两条反平行链,但只有两条DNA链中的一条被称为模板链用于转录。这是因为RNA是单链的,而DNA是双链的。另一条DNA链被称为编码链,因为它的序列与新创建的RNA转录本相同(除了尿嘧啶取代胸腺嘧啶)。只使用3'→5'链可以消除对DNA复制中观察到的冈崎片段的需要。转录分为5个阶段:起始前、起始、启动子清除、延伸和终止。
一个基因一个酶假说是指基因通过产生酶来发挥作用,每个基因负责产生一种特定的酶,而这种酶又会影响代谢途径中的一个特定步骤。这个概念是由乔治·比德尔和爱德华·塔特姆在他们1941年关于真菌Neurospora crassa的遗传突变的一篇有影响力的论文中提出的,[3] 后来被他们的合作者诺曼·霍洛维茨称为“一个基因一个酶假说”。它通常被认为是后来被称为分子生物学的第一项重大成果。虽然这个假说影响深远,但在它被提出后不久就被认识到它过于简单化了。甚至后来提出的“一个基因一个多肽”假说也被认为过于简单,无法描述基因和蛋白质之间的关系。[4]
什么是Neurospora?Neurospora crassa是一种属于子囊菌门的红色面包霉。属名意为“神经孢子”,指的是孢子上的特征条纹。
N. crassa被用作模式生物,因为它易于培养,并且具有单倍体生命周期,这使得遗传分析变得简单,因为隐性性状会在后代中显现出来。由于Neurospora的子囊孢子中减数分裂产物的排列有序,因此可以方便地分析遗传重组。它的整个包含7条染色体的基因组已经测序。爱德华·塔特姆和乔治·威尔斯·比德尔在他们的实验中使用Neurospora,他们因此获得了1958年的诺贝尔生理学或医学奖。比德尔和塔特姆用X射线照射N. crassa,导致突变。然后,他们观察了由于特定酶出现错误而导致的代谢途径中的故障。这导致他们提出了“一个基因一个酶”假说,即特定基因编码特定的蛋白质。他们的假说后来由诺曼·霍洛维茨进一步阐述,他也从事Neurospora的研究。
到20世纪50年代初,生物化学遗传学的进展——部分源于最初的假说——使一个基因一个酶假说看起来不太可能(至少在最初的形式上)。从1957年开始,弗农·英格拉姆等人通过蛋白质指纹图谱表明,蛋白质中的遗传变异(如镰状细胞血红蛋白)可能仅限于多聚体蛋白质中单个多肽链的差异,从而导致了“一个基因一个多肽”假说的提出。根据遗传学家罗兰·H·戴维斯的说法,“到1958年——实际上,即使到1948年——一个基因一个酶不再是需要坚决捍卫的假说;它仅仅是一个研究项目的名称。”目前,一个基因一个多肽的观点无法解释许多真核生物中的各种剪接版本,这些真核生物使用剪接体根据各种细胞间和细胞内环境信号分别准备RNA转录本。这种剪接是由菲利普·夏普和理查德·J·罗伯茨在1977年发现的。
操纵子是基因组材料的功能单位,包含一个基因簇,受单个调控信号或启动子的控制。这些基因一起转录成一个 mRNA 链,并在细胞质中一起翻译,或者进行转剪接以创建单顺反子 mRNA,这些 mRNA 被单独翻译,即几个 mRNA 链,每个链编码一个单一的基因产物。其结果是操纵子中包含的基因要么一起表达,要么根本不表达。几个基因必须同时转录和共调节以定义一个操纵子。最初认为操纵子只存在于原核生物中,但自从 1990 年代初在真核生物中发现第一个操纵子以来,越来越多的证据表明它们比以前认为的更常见。
操纵子主要存在于原核生物中,但也存在于一些真核生物中,包括线虫,如秀丽隐杆线虫,以及果蝇。 rRNA 基因通常存在于操纵子中,这些操纵子已在包括脊索动物在内的多种真核生物中被发现。操纵子由多个结构基因组成,这些基因排列在一个共同的启动子下,并受一个共同的操纵子的调节。它被定义为一组相邻的结构基因,加上影响结构基因转录的相邻调控信号。给定操纵子的调控因子,包括阻遏物、共阻遏物和激活物,不一定由该操纵子编码。调控因子、启动子、操纵子和结构 DNA 序列的位置和条件可以决定常见突变的影响。操纵子与调节子、刺激子和调控子相关。操纵子包含一组受相同操纵子调节的基因,而调节子包含一组受单个调控蛋白调节的基因,刺激子包含一组受单个细胞刺激调节的基因。[5]
操纵子的结构
[edit | edit source]启动子 - 使基因能够被转录的核苷酸序列。启动子被 RNA 聚合酶识别,然后启动转录。在 RNA 合成中,启动子指示哪些基因应该用于信使 RNA 的创建 - 并且,通过扩展,控制细胞制造哪些蛋白质。
操纵子 - DNA 片段,调控因子与之结合。它在乳糖操纵子中被经典地定义为启动子和操纵子基因之间的片段。在阻遏物的情况下,阻遏蛋白在物理上阻止 RNA 聚合酶转录基因。
结构基因 - 由操纵子共调节的基因。
原核生物启动子
[edit | edit source]在原核生物中,启动子包含两个短序列,分别位于转录起始位点上游的 -10 和 -35 位置。σ因子不仅有助于增强 RNAP 与启动子的结合,还有助于 RNAP 靶向特定基因进行转录。-10 的序列称为 Pribnow 盒,或 -10 元件,通常由六个核苷酸 TATAAT 组成。Pribnow 盒对于启动原核生物中的转录至关重要。-35 处的另一个序列(-35 元件)通常由七个核苷酸 TTGACAT 组成。它的存在允许非常高的转录速率。上述两个共有序列,虽然平均而言是保守的,但在大多数启动子中并没有完整地找到。平均而言,在每个共有序列中只有 6 个碱基对中的 3 个在任何给定的启动子中被发现。迄今为止,尚未发现任何启动子在 -10 和 -35 处都具有完整的共有序列;发现具有 -10/-35 六聚体的完全保守性的合成启动子以非常高的效率促进 RNA 链起始。一些启动子包含一个 UP 元件(共有序列 5'-AAAWWTWTTTTNNNAAANNN-3';W = A 或 T;N = 任何碱基),以 -50 为中心;-35 元件的存在似乎对于来自 UP 元件包含的启动子的转录并不重要。应该注意的是,上述启动子序列仅被与原核生物 RNA 聚合酶相互作用的 σ-70 蛋白识别。原核生物 RNA 聚合酶与其他 σ 因子的复合物识别完全不同的核心启动子序列。
<-- upstream downstream -->
5'-XXXXXXXPPPPPXXXXXXPPPPPPXXXXGGGCCGGGTTGGTTGGGCCGAAGGGTTGGCCGGGGGGGGXXXX-3'
-35 -10 Gene to be transcribed
真核生物启动子
[edit | edit source]真核生物启动子极其多样,难以表征。它们通常位于基因上游,并且可以具有距离转录起始位点几千个碱基对的调控元件(增强子)。在真核生物中,转录复合物可以导致 DNA 弯曲回到自身,这使得调控序列可以放置在远离实际转录位点的区域。许多真核生物启动子,在所有基因中占 10% 到 20%,包含一个TATA 盒(序列 TATAAA),进而结合一个TATA 结合蛋白,它有助于形成RNA 聚合酶转录复合物。TATA 盒通常位于非常靠近转录起始位点的区域(通常在 50 个碱基对内)。
真核生物启动子调控序列通常结合称为转录因子的蛋白质,这些蛋白质参与转录复合物的形成。一个例子是E 盒(序列 CACGTG),它与碱性螺旋-环-螺旋(bHLH)家族(例如BMAL1-Clock、cMyc)中的转录因子结合。
增强子
[edit | edit source]增强子是 DNA 的一个短区域,可以与蛋白质(即顺式作用因子,非常像一组转录因子)结合,以增强基因簇中基因的转录水平(因此得名)。虽然增强子通常是顺式作用的,但增强子不需要特别靠近它作用的基因,也不需要位于同一条染色体上。
在真核细胞中,DNA 的染色质复合体的结构以一种功能上模拟原核生物 DNA 特征的超螺旋状态的方式折叠,因此,尽管增强子 DNA 在核苷酸数量方面远离基因,但它在几何上靠近启动子和基因。这使它能够与通用转录因子和 RNA 聚合酶 II 相互作用。增强子可以位于其调控的基因的上游或下游。
此外,增强子不需要位于靠近转录起始位点的位置以影响基因的转录,因为一些增强子被发现结合在起始位点上游或下游数十万个碱基对处。增强子本身并不作用于启动子区域,而是被激活蛋白结合。这些激活蛋白与介质复合物相互作用,介质复合物招募聚合酶 II 和通用转录因子,然后开始转录基因。增强子也可以存在于内含子中。增强子的方向甚至可以反转,而不会影响其功能。此外,增强子可以被切除并插入染色体的其他位置,并且仍然影响基因转录。这就是为什么内含子多态性被检查,尽管它们没有被翻译。
共阻遏物
[edit | edit source]共阻遏物是一种蛋白质,通过与包含 DNA 结合域的转录因子结合来降低基因表达。共阻遏物本身无法结合 DNA。共阻遏物可以通过招募组蛋白脱乙酰酶来抑制转录起始,组蛋白脱乙酰酶催化从赖氨酸残基去除乙酰基。这增加了组蛋白的正电荷,从而增强了组蛋白与 DNA 之间的相互作用,使后者难以被转录。
核糖开关
[edit | edit source]在分子生物学中,核糖开关是 mRNA 分子的一部分,它可以直接结合一个小分子靶标,并且结合靶标会影响基因的活性。因此,包含核糖开关的 mRNA 直接参与调节自身的活性,以响应其靶标分子的浓度。现代生物体利用 RNA 结合小分子,并区分密切相关的类似物这一发现,显著扩展了人们对 RNA 天然能力的认识,超出了其编码蛋白质或结合其他 RNA 或蛋白质大分子的能力。该术语“核糖开关”的最初定义规定了它们直接感知小分子代谢物的浓度。虽然此定义仍然在普遍使用中,但一些生物学家使用更广泛的定义,包括其他顺式调控 RNA。但是,本文将只讨论代谢物结合核糖开关。大多数已知的核糖开关存在于细菌中,但已在植物和某些真菌中发现了类型为 TPP 核糖开关的功能性核糖开关。TPP 核糖开关也被预测存在于古细菌中,但尚未经过实验验证。[6]
lac 操纵子是大肠杆菌和其他一些肠杆菌中运输和代谢乳糖所需的操纵子。它包含三个相邻的结构基因,lacZ、lacY 和 lacA。lac 操纵子受多种因素调节,包括葡萄糖和乳糖的可用性。lac 操纵子的基因调控是第一个被阐明的复杂遗传调控机制,也是原核生物基因调控的最主要例子之一。
在自然环境中,lac 操纵子允许有效消化乳糖。细胞可以通过产生酶β-半乳糖苷酶来将乳糖消化成葡萄糖和半乳糖,从而利用乳糖作为能源。然而,在没有乳糖的情况下或存在更易获得的能量来源(如葡萄糖)的情况下,产生酶将是低效的。lac 操纵子使用两部分控制机制来确保细胞只有在必要时才消耗能量来产生β-半乳糖苷酶、β-半乳糖苷通透酶和硫代半乳糖苷转乙酰酶(也称为半乳糖苷 O-乙酰基转移酶)。它通过lac 阻遏蛋白来实现这一点,lac 阻遏蛋白在没有乳糖的情况下阻止生产,以及分解代谢激活蛋白(CAP),它在没有葡萄糖的情况下协助生产。这种双重控制机制导致葡萄糖和乳糖在两个不同的生长阶段(称为双营养生长)中被依次利用。类似的双营养生长模式已在细菌在其他糖混合物上的生长中观察到,例如葡萄糖和木糖,或葡萄糖和阿拉伯糖等的混合物。这种双营养生长模式背后的遗传控制机制被称为xyl 操纵子和ara 操纵子等。[7] 乳糖操纵子包含三个结构基因,以及一个启动子、一个终止子、一个调节器和一个操纵元。
三个结构基因是:lacZ、lacY 和 lacA。
lacZ 编码β-半乳糖苷酶(LacZ),一种将二糖乳糖裂解成葡萄糖和半乳糖的细胞内酶。
lacY 编码β-半乳糖苷通透酶(LacY),一种将乳糖泵入细胞的膜结合转运蛋白。
lacA 编码β-半乳糖苷转乙酰酶(LacA),一种将乙酰基从乙酰辅酶 A 转移到β-半乳糖苷的酶。
只有 lacZ 和 lacY 似乎是乳糖分解代谢所必需的。
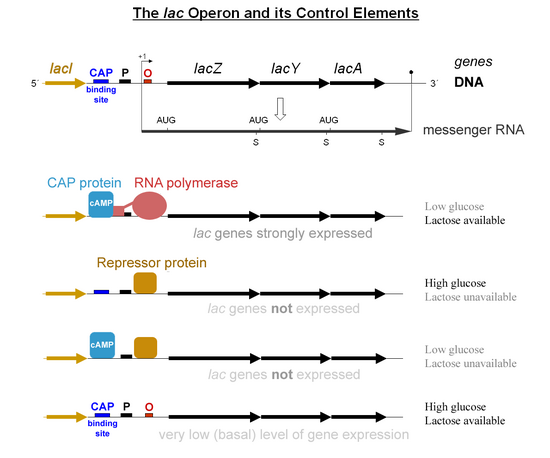
乳糖阻遏蛋白于 1966 年首次由沃尔特·吉尔伯特和本诺·穆勒-希尔分离出来。他们在体外证明了该蛋白质结合了含有乳糖操纵子的 DNA,并在添加 IPTG 后释放了 DNA。(IPTG 是一种异乳糖类似物。)他们还能够通过使用脱氧核糖核酸酶来分离由蛋白质结合的 DNA 部分,脱氧核糖核酸酶会分解 DNA。在处理阻遏蛋白-DNA 复合物后,一些 DNA 保留下来,表明它已被阻遏蛋白掩盖。这后来得到了证实。这些实验很重要,因为它们证实了乳糖操纵子的机制,该机制之前由雅克·莫诺和弗朗索瓦·雅各布提出。乳糖阻遏蛋白的结构包含三个不同的区域
一个核心区域(结合异乳糖)一个四聚化区域(将四个单体连接在一个α-螺旋束中)一个 DNA 结合区域(其中两个 LacI 蛋白结合单个操纵元位点)乳糖阻遏蛋白以四聚体(四个相同的亚基结合在一起)的形式存在。这可以看作是两个二聚体,每个二聚体能够结合到单个乳糖操纵元上。两个亚基分别结合到操纵元中稍微分离的(主沟)区域。启动子被乳糖阻遏蛋白稍微覆盖,因此 RNA 聚合酶无法结合并转录操纵子。DNA 结合区域包含一个螺旋-转角-螺旋结构基序。可在蛋白质百科中找到阻遏蛋白结构(一些与 DNA 结合)的交互式、旋转 3D 视图,包括其如何弯曲 DNA 双螺旋的变形。乳糖阻遏蛋白 (LacI) 通过结合到乳糖操纵子操纵元区域的主沟来发挥作用。这会阻止 RNA 聚合酶结合,从而阻止编码 Lac 蛋白的 mRNA 的转录。当存在乳糖时,异乳糖会结合到乳糖阻遏蛋白上,导致其形状发生变构变化。在这种变化状态下,乳糖阻遏蛋白无法结合到其同源操纵元上。
乳糖基因及其衍生物可以作为报告基因用于多种基于细菌的选择技术,例如双杂交分析,其中必须确定转录激活因子与特定启动子序列的成功结合。在含有 X-gal 的 LB 平板上,菌落颜色从白色变为蓝色对应于大约 20-100 个β-半乳糖苷酶单位,而四氮唑乳糖和麦康凯乳糖培养基的范围为 100-1000 个单位,分别在该范围的高端和低端最敏感。由于麦康凯乳糖和四氮唑乳糖培养基都依赖于乳糖分解的产物,因此它们需要 lacZ 和 lacY 基因的存在。因此,许多仅包含 lacZ 基因的乳糖融合技术适用于 X-gal 平板或 ONPG 液体培养基。[8]
色氨酸操纵子是一个操纵子——一组一起使用或转录的基因——它编码生产色氨酸的成分。色氨酸操纵子存在于许多细菌中,但最初是在大肠杆菌中被表征的。它受到调节,以便当环境中存在色氨酸时,它不会被使用。它是一个重要的实验系统,用于学习基因调控,并且通常用于教授基因调控。
色氨酸操纵子于 1953 年由雅克·莫诺及其同事发现,是第一个被发现的可阻遏操纵子。虽然乳糖操纵子可以被一种化学物质(异乳糖)激活,但色氨酸(Trp)操纵子会被一种化学物质(色氨酸)抑制。该操纵子包含五个结构基因:trp E、trp D、trp C、trp B 和 trp A,它们编码色氨酸合酶。它还包含一个启动子,该启动子与 RNA 聚合酶结合,以及一个操纵元,该操纵元在与阻遏基因(trp R)合成的蛋白质结合时会阻止转录,该蛋白质会结合到操纵元上。在乳糖操纵子中,异乳糖会结合到阻遏蛋白上,允许基因转录,而在色氨酸操纵子中,色氨酸会结合到阻遏蛋白上,有效地阻止基因转录。在这两种情况下,阻遏都是对 RNA 聚合酶转录操纵子中基因的阻遏。与乳糖操纵子不同,色氨酸操纵子还包含一个引导肽和一个衰减子序列,这允许分级调节。
它是基因表达负调控的一个例子。在操纵子的调控序列中,操纵元在存在色氨酸的情况下被阻遏蛋白阻断(从而阻止转录),并在色氨酸不存在的情况下被释放(从而允许转录)。衰减过程补充了这种调控作用。[9]
模式细菌大肠杆菌的L-阿拉伯糖操纵子是分子生物学研究的重点领域已有40多年,并在遗传、生化、生理和生物物理学层面进行了广泛研究。它受双重正负系统控制。有3个结构基因:araB、araA和araD。它们编码分解单糖阿拉伯糖为D-木酮糖-5-磷酸的代谢酶,然后通过戊糖磷酸途径代谢。包含操纵子位点和启动子的起始区称为araI(araI的最后一个字母是大写字母“i”)。在该位点附近是araC基因,它编码一个阻遏蛋白。AraC蛋白结合到起始区araI。
管家基因通常是构成型基因,对于维持基本的细胞功能是必需的,存在于所有的人类细胞中。虽然一些管家基因以相对恒定的水平表达(如HSP90和β-肌动蛋白),但其他管家基因可能会根据实验条件而变化。术语“管家基因”的起源仍然不清楚。1976年的文献使用该术语专门描述tRNA和rRNA。解释基因表达数据可能存在问题,大多数人类基因在每个细胞中注册5-10个拷贝(可能代表误差)。管家基因在每个细胞中至少表达25个拷贝,有时数量达到数千个。
基因表达的调控是指对基因功能产物数量和出现时间的控制。表达控制对于细胞在需要时产生所需的基因产物至关重要;反过来,这使细胞能够灵活地适应可变环境、外部信号、细胞损伤等。基因表达重要的简单例子包括
控制胰岛素表达,使其发出信号以调节血糖
雌性哺乳动物中X染色体的失活,以防止其所含基因的“过量表达”。
细胞周期蛋白表达水平控制真核细胞周期进程
更一般地说,基因调控使细胞能够控制所有结构和功能,是细胞分化、形态发生以及任何生物体多功能性和适应性的基础。基因表达的任何步骤都可以进行调节,从DNA-RNA转录步骤到蛋白质的翻译后修饰。最终基因产物(无论是RNA还是蛋白质)的稳定性也影响基因的表达水平——不稳定的产物会导致低表达水平。一般来说,基因表达是通过改变影响DNA转录和RNA翻译的分子之间相互作用的数量和类型的变化来调节的。根据基因的调节方式,有许多术语用于描述基因类型,这些术语包括:构成型基因是指与兼性基因相比,持续转录的基因,兼性基因仅在需要时转录。管家基因通常是构成型基因,以相对恒定的水平转录。管家基因的产物通常是维持细胞所必需的。一般认为它们的表达不受实验条件的影响。例子包括肌动蛋白、GAPDH和泛素。兼性基因是指与构成型基因相比,仅在需要时转录的基因。诱导型基因是指其表达对环境变化有反应或依赖于细胞周期中位置的基因。[10]
转录调控 转录调控可以细分为三个主要的影响途径;遗传(控制因子与基因的直接相互作用)、调节(控制因子与转录机制的相互作用)和表观遗传(DNA结构的非序列变化,影响转录)。
λ阻遏蛋白转录因子(绿色)以二聚体形式结合到DNA靶标(红色和蓝色)的主要沟,并禁用转录起始。来自PDB 1LMB。与DNA的直接相互作用是蛋白质改变转录水平的最简单、最直接的方法。基因通常在其编码区的周围具有多个蛋白质结合位点,这些位点具有调节转录的特定功能。已知许多类型的调控DNA结合位点,如增强子、绝缘子、阻遏子和沉默子。调节转录的机制多种多样,从阻断RNA聚合酶在DNA上的关键结合位点,到充当激活剂并通过帮助RNA聚合酶结合来促进转录。转录因子的活性还受到细胞内信号的进一步调节,导致蛋白质翻译后修饰,包括磷酸化、乙酰化或糖基化。这些变化会影响转录因子直接或间接结合启动子DNA、招募RNA聚合酶或有利于新合成RNA分子的延伸的能力。真核生物中的核膜允许通过转录因子的核内存在时间来进一步调节转录因子,这种存在时间受其结构可逆变化以及其他蛋白质的结合的调节。环境刺激或内分泌信号可能会引起调控蛋白的修饰,引发细胞内信号级联反应,最终导致基因表达的调控。最近,越来越明显的是,非DNA序列特异性效应对翻译有巨大影响。这些效应被称为表观遗传学,涉及DNA的高级结构、非序列特异性DNA结合蛋白以及DNA的化学修饰。一般来说,表观遗传效应改变了DNA对蛋白质的可及性,因此调节转录。
在真核生物中,DNA以核小体的形式组织。注意DNA(蓝色和绿色)是如何紧紧地包裹在由组蛋白八聚体(带状线圈)构成的蛋白质核心周围的,限制了对DNA的访问。来自PDB 1KX5。DNA甲基化是表观遗传学影响基因表达的普遍机制,存在于细菌和真核生物中,在可遗传的转录沉默和转录调控中发挥作用。在真核生物中,染色质的结构受组蛋白密码控制,调节对DNA的访问,对常染色质和异染色质区域中基因的表达有重大影响。
转录后调控
在真核生物中,由于RNA的转运是翻译的必要条件,因此核转运被认为提供了对基因表达的额外控制。所有进出核的转运都是通过核孔进行的,转运受多种进出口蛋白的控制。编码蛋白质的基因的表达只有在携带密码的信使RNA存活足够长的时间以被翻译的情况下才有可能。在一个典型的细胞中,一个RNA分子只有在被专门保护免受降解的情况下才稳定。RNA降解在真核细胞的表达调控中具有特殊的重要性,在真核细胞中,mRNA必须在被翻译之前长距离移动。在真核生物中,RNA通过某些转录后修饰而稳定,特别是5'帽子和聚腺苷酸化尾。有意降解mRNA不仅用作防御机制,防止外源RNA(通常来自病毒),而且也是mRNA去稳定化的途径。如果一个mRNA分子与一个小的干扰RNA具有互补序列,那么它就会通过RNA干扰途径被靶向降解。
翻译调控
新霉素是减少所有蛋白质基因表达的小分子,它不可避免地导致细胞死亡,因此具有抗生素的作用。
直接调节翻译不如转录或mRNA稳定性的控制普遍,但偶尔也会使用。抑制蛋白质翻译是毒素和抗生素的主要靶标,目的是通过覆盖其正常的基因表达控制来杀死细胞。蛋白质合成抑制剂包括抗生素新霉素和毒素蓖麻毒素。蛋白质降解
蛋白质合成完成后,可以通过蛋白质降解来降低该蛋白质的表达水平。在所有原核生物和真核生物中都存在主要的蛋白质降解途径,其中蛋白酶体是共同的组成部分。不需要或受损的蛋白质通常通过添加泛素来标记降解。
基因工程中使用的质粒被称为载体。质粒是遗传学和生物技术实验室的重要工具,它们通常用于复制(制作特定基因的许多副本)或表达特定基因。许多质粒可用于此类用途。要复制的基因被插入到包含使细胞对特定抗生素具有抗性的基因和多克隆位点(MCS 或多连接体)的质粒副本中,该位点是一个包含几个常用限制性酶切位点的短区域,允许轻松地将 DNA 片段插入此位置。 接下来,质粒通过称为转化的方法插入细菌。然后,细菌暴露于特定的抗生素。只有吸收了质粒副本的细菌才能存活,因为质粒使它们具有抗性。特别是,保护基因被表达(用于制造蛋白质),表达的蛋白质分解抗生素。通过这种方式,抗生素充当过滤器,仅选择修饰过的细菌。现在,这些细菌可以在大量培养、收获和裂解(通常使用碱性裂解法)以分离感兴趣的质粒。质粒的另一个主要用途是制造大量蛋白质。在这种情况下,研究人员培养含有包含感兴趣基因的质粒的细菌。正如细菌产生蛋白质以赋予其抗生素抗性一样,它也可以被诱导产生大量来自插入基因的蛋白质。这是一种廉价且简单的大规模生产基因或其编码的蛋白质的方法,例如胰岛素甚至抗生素。但是,质粒只能包含约 1-10 kbp 的插入片段。为了克隆更长的 DNA 片段,可以使用删除了溶源基因的 λ 噬菌体、柯斯质粒、细菌人工染色体或酵母人工染色体。 [11]
现代载体可能包含除转基因插入片段和骨架之外的其他特征:启动子:所有载体的必要组成部分:用于驱动载体转基因的转录。
遗传标记:病毒载体的遗传标记可以确认载体是否已整合到宿主基因组 DNA 中。
抗生素抗性:具有抗生素抗性开放阅读框的载体允许在含有抗生素的培养基中通过抗生素选择,吸收了载体的细胞存活。
表位:载体包含特定表位的序列,该序列被整合到表达的蛋白质中。允许抗体识别表达目标蛋白质的细胞。
β-半乳糖苷酶:一些载体包含 β-半乳糖苷酶的序列,β-半乳糖苷酶是一种消化半乳糖的酶,其中包含多克隆位点(基因可能被插入的区域)。成功连接到载体的插入片段将破坏 β-半乳糖苷酶基因并使半乳糖消化失效。包含载体插入片段的细胞可以使用蓝白筛选来识别,方法是在含有半乳糖类似物(X-gal)的培养基中培养细胞。表达 β-半乳糖苷酶(因此不包含插入片段)的细胞表现为蓝色菌落。白色菌落将被选择为可能包含插入片段的菌落。其他可能类似于报告基因的功能的蛋白质包括绿色荧光蛋白和荧光素酶。
靶向序列:表达载体可能包含在最终蛋白质中编码靶向序列,该序列将表达的蛋白质引导至细胞中的特定细胞器或特定位置,例如细菌的周质空间。
蛋白质纯化标签:一些表达载体包含蛋白质或肽序列,这些序列允许更容易地纯化表达的蛋白质。示例包括多组氨酸标签、谷胱甘肽-S-转移酶和麦芽糖结合蛋白。这些标签中的一些还可以增加目标蛋白质的溶解度。目标蛋白质与蛋白质标签融合,但在蛋白质和标签之间的多肽连接体区域定位的蛋白酶裂解位点允许后来去除标签。柯斯质粒 柯斯质粒主要是带有细菌 oriV、抗生素选择标记和克隆位点的质粒,但它们带有来自 λ 噬菌体的 1 个或最近 2 个 cos 位点。根据实验的具体目标,可以获得广宿主范围柯斯质粒、穿梭柯斯质粒或“哺乳动物”柯斯质粒(与 SV40 oriV 和哺乳动物选择标记相关)。柯斯质粒的负载能力取决于载体本身的大小,但通常在 40-45 kb 左右。克隆程序涉及生成两个载体臂,然后将其与外源 DNA 连接。针对野生型柯斯质粒 DNA 的选择简单地通过尺寸排阻来完成。然而,柯斯质粒始终形成菌落,而不是噬菌斑。此外,克隆密度要低得多,每 µg 连接的 DNA 约为 105 - 106 个 CFU。在构建重组 λ 或柯斯质粒文库后,将总 DNA 通过称为体外包装的技术转移到合适的 E.coli 宿主中。必要的包装提取物来自 E.coli cI857 溶源体(分别为 red- gam- Sam 和 Dam(头部组装)和 Eam(尾部组装))。这些提取物将识别和体外包装重组分子,产生成熟的噬菌体颗粒(基于 λ 的载体)或包含在噬菌体外壳中的重组质粒(柯斯质粒)。这些差异反映在支持 λ 替代载体的不同感染频率中。这弥补了它们略低的负载能力。噬菌体文库也比柯斯质粒(菌落!)文库更容易储存和筛选。目标 DNA:要克隆的基因组 DNA 必须切割成适当大小范围的限制性片段。这通常通过部分限制性酶切,然后进行尺寸分馏或去磷酸化(使用小肠碱性磷酸酶)来完成,以避免染色体混杂,即物理上没有关联的片段的连接。Fosmids Fosmids 类似于柯斯质粒,但基于细菌 F-质粒。克隆载体受到限制,因为宿主(通常是 E.coli)只能包含一个 fosmid 分子。Fosmids 是 40 kb 的随机基因组 DNA。Fosmid 文库从目标生物的基因组中制备,并克隆到 fosmid 载体中。低拷贝数比同等的高拷贝数柯斯质粒具有更高的稳定性。Fosmid 系统可能有助于构建来自复杂基因组的稳定文库。Fosmid 克隆被用来帮助评估公共人类基因组序列的准确性。
细菌人工染色体 (BAC) 细菌人工染色体 (BAC) 是一种基于功能性生育质粒 (或 F-质粒) 的 DNA 结构,用于细菌(通常是 E.coli)中的转化和克隆。F-质粒发挥着至关重要的作用,因为它们包含促进细菌细胞分裂后质粒均匀分布的分隔基因。细菌人工染色体的通常插入片段大小为 150-350 kbp,但可以大于 700 kbp。一种类似的克隆载体称为 PAC,也已经从细菌 P1-质粒中产生。BAC 通常用于基因组项目中对生物体的基因组进行测序,例如人类基因组项目。生物体的 DNA 的一小段被放大作为插入片段,然后在 BAC 中进行测序。最后,对测序的部分进行计算机模拟,从而得到生物体的基因组序列。
酵母人工染色体 (YAC) 酵母人工染色体 (YAC) 是一种用于克隆大于 100 kb 且高达 3000 kb 的 DNA 片段的载体。YAC 有助于复杂基因组的物理作图和大型基因的克隆。YAC 由 Murray 和 Szostak 于 1983 年首次描述,它是一种人工构建的染色体,包含在酵母细胞中复制和保存所需的端粒、着丝粒和复制起点序列。YAC 是使用一个初始的环状质粒构建的,该质粒通常使用限制性酶切割成两个线性分子;然后使用 DNA 连接酶将感兴趣的序列或基因连接到两个线性分子之间,形成一个大的线性 DNA 片段。[需要引用] 酵母表达载体(如 YAC、YIps(酵母整合质粒)和 YEps(酵母附加型质粒))比细菌人工染色体 (BAC) 具有优势,因为它们可以用于表达需要翻译后修饰的真核蛋白质。然而,已经发现 YAC 比 BAC 稳定性差,会产生嵌合效应。
病毒载体的类型
逆转录病毒
逆转录病毒是当前基因治疗方法的主要支柱之一。重组逆转录病毒(如莫洛尼鼠白血病病毒)能够以稳定的方式整合到宿主基因组中。它们包含一个逆转录酶,该酶允许整合到宿主基因组中。它们已用于许多 FDA 批准的临床试验,例如 SCID-X1 试验。逆转录病毒载体可以是复制competent或复制defective的。复制defective载体是研究中最常见的选择,因为病毒已经具有编码额外轮次病毒体复制和包装所需的基因的编码区,这些编码区被其他基因取代或删除。这些病毒能够感染其目标细胞并递送其病毒载荷,但随后无法继续典型的裂解途径,该途径导致细胞裂解和死亡。相反,复制competent病毒载体包含病毒体合成所需的所有必要基因,并且一旦感染发生就会继续繁殖。由于这些载体的病毒基因组要长得多,因此与复制defective载体的插入片段可能长度相比,实际插入的感兴趣基因的长度受到限制。根据病毒载体的不同,复制defective病毒载体中允许的 DNA 插入片段的典型最大长度通常约为 8-10 kB。虽然这限制了许多基因组序列的引入,但大多数 cDNA 序列仍然可以容纳。使用莫洛尼逆转录病毒等逆转录病毒的主要缺点是需要细胞积极分裂才能进行转导。因此,神经元等细胞对逆转录病毒的感染和转导具有很强的抵抗力。人们担心由于整合到宿主基因组中而引起的插入突变可能导致癌症或白血病。
慢病毒
慢病毒是逆转录病毒的一个亚类。由于它们能够整合到非分裂细胞的基因组中,近年来它们被用作基因传递载体(载体)。这是慢病毒的独特特性,因为其他逆转录病毒只能感染分裂细胞。当病毒进入细胞时,以 RNA 形式存在的病毒基因组被逆转录为 DNA,然后由病毒整合酶在基因组的随机位置插入。该载体现在被称为前病毒,它保留在基因组中,并在细胞分裂时传递给子代细胞。整合位点是不可预测的,这可能是一个问题。前病毒可能干扰细胞基因的功能,并导致癌基因的激活,从而促进癌症的发展,这引起了人们对慢病毒在基因治疗中可能应用的担忧。然而,研究表明,与 γ-逆转录病毒载体相比,慢病毒载体在可能导致癌症的位置整合的可能性更低。更具体地说,一项研究发现,在肿瘤发生率高得多的鼠系中,慢病毒载体既不会导致肿瘤发生率增加,也不会导致肿瘤早期发生。此外,利用慢病毒载体将基因治疗用于治疗 HIV 的临床试验中,没有出现致突变或致癌事件的增加。出于安全原因,慢病毒载体从不携带它们复制所需的基因。为了产生慢病毒,将几种质粒转染到所谓的包装细胞系中,通常是 HEK 293。一个或多个质粒,通常被称为包装质粒,编码病毒蛋白,例如衣壳蛋白和逆转录酶。另一个质粒包含要由载体传递的遗传物质。它被转录产生单链 RNA 病毒基因组,并以 ψ(psi)序列的存在为标志。该序列用于将基因组包装到病毒颗粒中。
腺病毒
与慢病毒相反,腺病毒 DNA 不整合到基因组中,也不在细胞分裂过程中复制。这限制了它们在基础研究中的应用,尽管腺病毒载体偶尔用于体外实验。它们的主要应用是基因治疗和疫苗接种。由于人类通常会接触到腺病毒,腺病毒会导致呼吸道、胃肠道和眼部感染,因此它们会引发快速免疫反应,并可能造成危险后果。为了克服这个问题,科学家们目前正在研究人类没有免疫力的腺病毒。
腺相关病毒
腺相关病毒 (AAV) 是一种感染人类和其他一些灵长类动物的小病毒。目前尚不清楚 AAV 会引起疾病,因此该病毒引起的免疫反应非常轻微。AAV 可以感染分裂和非分裂细胞,并可能将其基因组整合到宿主细胞的基因组中。这些特性使 AAV 成为创建用于基因治疗的病毒载体的非常有吸引力的候选者。

PCR
[edit | edit source]PCR 用于扩增 DNA 链的特定区域(DNA 目标)。大多数 PCR 方法通常扩增不超过 ~10 千碱基对 (kb) 的 DNA 片段,尽管有些技术允许扩增高达 40 kb 的片段。基本的 PCR 设置需要几个组件和试剂。这些组件包括
包含要扩增的 DNA 区域(目标)的 DNA 模板。
两个引物,分别与 DNA 目标的正义链和反义链的 3'(三素)端互补。Taq 聚合酶或其他 DNA 聚合酶,其最佳温度约为 70 °C。脱氧核苷三磷酸 (dNTP),DNA 聚合酶合成新 DNA 链的构建块。缓冲溶液,为 DNA 聚合酶提供最佳活性及稳定性的适当化学环境。二价阳离子,镁或锰离子;通常使用 Mg2+,但 Mn2+ 可用于 PCR 介导的 DNA 诱变,因为更高的 Mn2+ 浓度会提高 DNA 合成过程中的错误率。一价阳离子钾离子。PCR 通常在热循环仪的小反应管(0.2–0.5 ml 体积)中,在 10–200 μl 的反应体积中进行。热循环仪加热和冷却反应管,以在反应的每个步骤中实现所需的温度(见下文)。许多现代热循环仪利用珀耳帖效应,通过简单地反转电流即可实现保持 PCR 管的块的加热和冷却。薄壁反应管允许良好的热传导,从而实现快速热平衡。大多数热循环仪都有加热盖,以防止反应管顶部的冷凝。缺少加热盖的较旧热循环仪需要在反应混合物顶部添加一层油或在管内添加一块蜡。[12]
步骤
图 1:PCR 循环的示意图。 (1) 在 94–96 °C 下变性。 (2) 在 ~65 °C 下退火 (3) 在 72 °C 下延伸。此处显示了四个循环。蓝线代表 DNA 模板,引物(红色箭头)与其退火,并由 DNA 聚合酶(浅绿色圆圈)延伸,得到更短的 DNA 产物(绿色线),这些产物本身在 PCR 进行时用作模板。通常,PCR 包含一系列 20-40 次重复的温度变化,称为循环,每个循环通常包含 2-3 个离散的温度步骤,通常是三个。循环通常以单个温度步骤(称为保持)在高温 (>90 °C) 下进行,并在最后以一个保持结束,用于最终产物延伸或短暂储存。使用的温度以及它们在每个循环中应用的时间长度取决于各种参数。这些包括用于 DNA 合成的酶、反应中二价离子和 dNTP 的浓度以及引物的熔解温度 (Tm)。初始化步骤:此步骤包括将反应加热到 94–96 °C(或如果使用极其耐热聚合酶,则为 98 °C),并保持 1–9 分钟。它仅适用于需要通过热启动 PCR 进行热激活的 DNA 聚合酶。变性步骤:这是第一个常规循环事件,包括将反应加热到 94–98 °C,持续 20–30 秒。它通过破坏互补碱基之间氢键引起 DNA 模板的熔解,从而产生单链 DNA 分子。退火步骤:将反应温度降低到 50–65 °C,持续 20–40 秒,允许引物与单链 DNA 模板退火。通常,退火温度比所用引物的 Tm 低约 3-5 摄氏度。稳定的 DNA-DNA 氢键仅在引物序列与模板序列非常匹配时才会形成。聚合酶与引物-模板杂交体结合,并开始 DNA 合成。延伸/延长步骤:此步骤的温度取决于所使用的 DNA 聚合酶;Taq 聚合酶的最佳活性温度为 75–80 °C,并且通常使用该酶时使用 72 °C 的温度。在此步骤中,DNA 聚合酶通过添加与模板互补的 dNTP 来合成与 DNA 模板链互补的新 DNA 链,以 5' 到 3' 的方向,将 dNTP 的 5'-磷酸基团与新生(延伸)DNA 链末端的 3'-羟基缩合。延长时间取决于所使用的 DNA 聚合酶和要扩增的 DNA 片段的长度。作为一个经验法则,在其最佳温度下,DNA 聚合酶每分钟将聚合 1000 个碱基。在最佳条件下,即如果不存在由限制性底物或试剂造成的限制,在每个延伸步骤中,DNA 目标的数量都会翻倍,导致特定 DNA 片段的指数(几何)扩增。最终延伸:此单个步骤偶尔在最后一个 PCR 循环后在 70–74 °C 的温度下进行 5–15 分钟,以确保任何剩余的单链 DNA 完全延伸。[13]最终保持:此步骤在 4–15 °C 的温度下进行无限时间,可用于短期储存反应。
为了检查 PCR 是否生成了预期的 DNA 片段(有时也称为扩增子或扩增产物),采用琼脂糖凝胶电泳对 PCR 产物进行大小分离。PCR 产物的大小通过与 DNA 梯度(分子量标记)进行比较来确定,DNA 梯度包含已知大小的 DNA 片段,与 PCR 产物一起在凝胶上运行。
限制性内切酶
[edit | edit source]限制性内切酶识别特定的核苷酸序列,并在 DNA 中产生双链切割。虽然识别序列在 4 到 8 个核苷酸之间变化,但其中许多是 回文,对应于从前到后读取相同的氮碱基序列。[14] 理论上,DNA 中可能存在两种类型的回文序列。镜像回文类似于普通文本中发现的回文,其中一个序列在同一 DNA 链(即单链)上前后读取相同,例如 GTAATG。反向重复回文也是一个序列,前后读取相同,但前后序列在互补的 DNA 链(即双链)中找到,例如 GTATAC(注意,GTATAC 与 CATATG 互补)。[15] 反向重复比镜像重复更常见,并且具有比镜像重复更大的生物学意义。
5'-GTATAC-3' :::::: 3'-CATATG-5' |
| 回文识别位点在反向链上的读取方式与在正向链上的读取方式相同 |
EcoRI 酶切产生“粘性”末端,
而 SmaI 限制性内切酶切割产生“平 blunt”末端。
每个限制性内切酶的 DNA 识别序列不同,导致酶切产生的片段长度、序列和链方向(5' 端 或 3' 端)的“粘性末端”悬垂不同。 [16]
识别相同序列的不同限制性内切酶被称为 同源异裂酶。它们通常在序列的不同位置切割;然而,识别和切割相同位置的不同酶被称为 同裂酶。
限制性内切酶分类
限制性内切酶根据其组成和酶辅助因子的需求、目标序列的性质以及其 DNA 切割位点相对于目标序列的位置,被分为三或四类(I 型、II 型和 III 型)。限制性内切酶共有四类:I 型、II 型、III 型和 IV 型。所有类型的酶都识别特定的短 DNA 序列,并进行 DNA 的内切核酸酶切割,产生具有末端 5'-磷酸的特定双链片段。它们在识别序列、亚基组成、切割位置和辅助因子需求方面有所不同,总结如下:
I 型酶(EC 3.1.21.3) 在识别位点以外的位点切割;需要 ATP 和 S-腺苷甲硫氨酸才能发挥作用;具有限制性和甲基化酶(EC 2.1.1.72)两种功能的多功能蛋白。
II 型酶(EC 3.1.21.4) 在识别位点内或距离识别位点很短的距离内切割;大多数需要镁;独立于甲基化酶的单功能(限制性)酶。
III 型酶(EC 3.1.21.5) 在距离识别位点很短的距离内切割;需要 ATP(但不水解 ATP);S-腺苷甲硫氨酸刺激反应但不必需;作为与修饰甲基化酶(EC 2.1.1.72)的复合物的一部分存在。IV 型酶靶向甲基化的 DNA。
限制性内切酶的例子包括:[17]
| 酶 | 来源 | 识别序列 | 切割 |
|---|---|---|---|
| EcoRI | 大肠杆菌 |
5'GAATTC 3'CTTAAG |
5'---G AATTC---3' 3'---CTTAA G---5' |
| EcoRII | 大肠杆菌 |
5'CCWGG 3'GGWCC |
5'--- CCWGG---3' 3'---GGWCC ---5' |
| BamHI | 枯草芽孢杆菌 |
5'GGATCC 3'CCTAGG |
5'---G GATCC---3' 3'---CCTAG G---5' |
| HindIII | 流感嗜血杆菌 |
5'AAGCTT 3'TTCGAA |
5'---A AGCTT---3' 3'---TTCGA A---5' |
| TaqI | 热泉菌 |
5'TCGA 3'AGCT |
5'---T CGA---3' 3'---AGC T---5' |
| NotI | 耳诺卡氏菌 |
5'GCGGCCGC 3'CGCCGGCG |
5'---GC GGCCGC---3' 3'---CGCCGG CG---5' |
| HinfI | 流感嗜血杆菌 |
5'GANTCA 3'CTNAGT |
5'---G ANTC---3' 3'---CTNA G---5' |
| Sau3A | 金黄色葡萄球菌 |
5'GATC 3'CTAG |
5'--- GATC---3' 3'---CTAG ---5' |
| PovII* | 普通变形杆菌 |
5'CAGCTG 3'GTCGAC |
5'---CAG CTG---3' 3'---GTC GAC---5' |
| SmaI* | 粘质沙雷氏菌 |
5'CCCGGG 3'GGGCCC |
5'---CCC GGG---3' 3'---GGG CCC---5' |
| HaeIII* | 埃及嗜血杆菌 |
5'GGCC 3'CCGG |
5'---GG CC---3' 3'---CC GG---5' |
| HgaI[18] | 鸡嗜血杆菌 |
5'GACGC 3'CTGCG |
5'---NN NN---3' 3'---NN NN---5' |
| AluI* | 类芽孢杆菌 |
5'AGCT 3'TCGA |
5'---AG CT---3' 3'---TC GA---5' |
| EcoRV* | 大肠杆菌 |
5'GATATC 3'CTATAG |
5'---GAT ATC---3' 3'---CTA TAG---5' |
| EcoP15I | 大肠杆菌 |
5'CAGCAGN25NN 3'GTCGTCN25NN |
5'---CAGCAGN25NN ---3' 3'---GTCGTCN25 NN---5' |
| KpnI | 肺炎克雷伯菌 |
5'GGTACC 3'CCATGG |
5'---GGTAC C---3' 3'---C CATGG---5' |
| PstI | 斯氏普罗维登斯菌 |
5'CTGCAG 3'GACGTC |
5'---CTGCA G---3' 3'---G ACGTC---5' |
| SacI | 无色链霉菌 |
5'GAGCTC 3'CTCGAG |
5'---GAGCT C---3' 3'---C TCGAG---5' |
| SalI | 白色链霉菌 |
5'GTCGAC 3'CAGCTG |
5'---G TCGAC---3' 3'---CAGCT G---5' |
| ScaI | 丛毛链霉菌 |
5'AGTACT 3'TCATGA |
5'---AGT ACT---3' 3'---TCA TGA---5' |
| SpeI | 球衣菌 |
5'ACTAGT 3'TGATCA |
5'---A CTAGT---3' 3'---TGATC A---5' |
| SphI | 嗜色链霉菌 |
5'GCATGC 3'CGTACG |
5'---GCATG C---3' 3'---C GTACG---5' |
| StuI[19][20] | 结核链霉菌 |
5'AGGCCT 3'TCCGGA |
5'---AGG CCT---3' 3'---TCC GGA---5' |
| XbaI | 黄单胞菌 |
5'TCTAGA 3'AGATCT |
5'---T CTAGA---3' 3'---AGATC T---5' |
关键
* = 平 blunt 末端
N = C 或 G 或 T 或 A
W = A 或 T
基因克隆及其表达
[edit | edit source]在分子生物学中,克隆是指分离一个确定的 DNA 序列并在体外获得其多个拷贝的过程。克隆经常用于扩增包含基因的 DNA 片段,但也可以用于扩增任何 DNA 序列,例如启动子、非编码序列、化学合成的寡核苷酸和随机片段化的 DNA。克隆广泛应用于各种生物学实验和技术应用中,例如大规模蛋白质生产和基因在 HeLa 细胞等细胞系中的表达。
从本质上讲,为了在体内和体外扩增任何 DNA 序列,所讨论的序列必须与能够指导自身和连接序列在所需目标宿主中复制和增殖的主要序列元素连接起来。所需的序列元素根据宿主而不同,但总是包括复制起点和一个可选择标记。然而,在实践中,许多其他特征是需要的,并且存在各种专门的克隆 载体,它们允许蛋白质表达、标记、单链 RNA 和 DNA 的生产,以及在后续应用中很有用的其他操作。
基于重组酶的克隆 一种新的克隆或亚克隆任何 DNA 片段的方法,通过将感兴趣的特殊 DNA 片段插入到目标 DNA 的特殊区域,通过相关 DNA 片段的交换来实现。 [21]
这是一个一步反应:简单、高效,有利于高通量或自动克隆和/或亚克隆。 [22]
限制性/连接克隆 在经典的限制性和连接克隆方案中,克隆任何 DNA 片段基本上涉及四个步骤:使用 限制性内切酶 进行 DNA 片段化,将 DNA 片段与 载体 进行 连接, 转染 和 筛选/选择。尽管这些步骤在克隆过程中是恒定的,但根据具体应用,可以在各个点选择许多替代路线;这些被总结为“克隆策略”。
插入片段的分离 最初,需要分离要克隆的 DNA 片段。克隆 DNA 片段的制备可以通过多种替代方法完成。插入片段的制备通常通过 聚合酶链式反应 来实现,但也可以通过 限制性内切酶 消化、DNA 超声处理 和 琼脂糖凝胶电泳 分离来实现。如果目标序列大小不超过化学合成的限制,也可以使用化学合成的寡核苷酸。插入片段的分离可以使用鸟枪法克隆、c-DNA 克隆、基因机器(人工化学合成)来完成。
转化
连接完成后,连接产物(质粒)被 转化 到细菌中进行增殖。然后将细菌接种到选择性琼脂培养基上,以选择含有感兴趣质粒的细菌。挑取单个菌落并测试其是否含有所需的插入片段。可以进行大提质粒操作以获得大量含有插入基因的质粒。
转染 连接完成后,连接反应的一部分(包括以所需方向插入片段的载体)被 转染 到细胞中。有多种替代技术可用,例如细胞的化学敏感化、电穿孔和基因枪轰击。细胞的化学敏感化经常被采用,因为这不需要专门的设备,并且提供相对较高的转化效率。当需要极高的转化效率时(例如在非常低效的克隆策略中),使用 电穿孔。 基因枪轰击 主要用于植物细胞转化,其中细胞壁是 DNA 被细胞吸收的主要障碍。细菌转化通常通过蓝白筛选来观察。
选择 最后, 转染 的细胞被培养。由于上述过程的效率特别低,因此需要识别含有所需插入片段且方向正确的细胞,并将这些细胞从未成功转化的细胞中分离出来。现代 克隆载体 包括可选择标记(最常见的是 抗生素 抗性标记),这些标记只允许载体(但不是插入片段)被转染的细胞生长。此外,克隆载体可能包含颜色选择标记,这些标记在 X-gal 培养基上提供蓝白筛选(通过 α 因子互补)。然而,这些选择步骤不能完全保证 DNA 插入片段存在于细胞中。需要对所得菌落进行进一步研究以确认克隆是否成功。这可以通过 PCR、限制性片段长度多态性 和/或 DNA 测序来实现。
基因工程
基因工程是一种通过改变生物体的遗传物质,以预定的方式改变其遗传特性的方法。这通常是为了使微生物(如细菌或病毒)能够合成更高产量的化合物,形成全新的化合物,或适应不同的环境。这项技术(也称为重组 DNA 技术)的其他用途包括基因治疗,即向患有遗传疾病或其他疾病(如获得性免疫缺陷综合征 (AIDS) 或癌症)的人提供功能基因,以及整个生物体的克隆。
遗传工程涉及操纵脱氧核糖核酸,即DNA。在这个过程中,重要的工具是限制性内切酶(称为限制性酶),它们是由各种细菌产生的。限制性酶可以识别DNA分子中化学单元链(称为核苷酸碱基)的特定序列,并在该位置切割DNA。这样产生的DNA片段可以使用另一种称为连接酶的酶连接起来。因此,限制性酶和连接酶允许对DNA片段进行特定的切割和重新组装。在DNA操纵中也很重要的是所谓的载体,它们是能够独立于宿主细胞中DNA进行自我复制(产生自身的副本)的DNA片段。载体的例子包括质粒、病毒和人工染色体。载体允许生成特定DNA片段的多个副本,使其成为生成足够数量可用于工作的材料的有用方法。将DNA片段工程化到载体的过程称为“分子克隆”,因为会产生多个相同DNA分子的副本。另一种产生许多相同特定(通常是短的,例如100-3,000个碱基对)DNA片段副本的方法是聚合酶链反应。该方法快速且避免了将DNA克隆到载体的需要。
报告基因
[edit | edit source]在分子生物学中,报告基因是指研究人员将其连接到细胞培养、动物或植物中另一个感兴趣基因的调控序列的基因。某些基因被选作报告基因,因为它们赋予表达它们的生物体的特征易于识别和测量,或者因为它们是选择性标记。报告基因通常被用作特定基因是否被细胞或生物体群体吸收或表达的指示。
为了将报告基因导入生物体,科学家将报告基因和感兴趣基因放在同一个DNA构建体中,将其插入细胞或生物体。对于细菌或培养中的真核细胞,这通常以称为质粒的环状DNA分子的形式出现。重要的是使用在研究的细胞或生物体中未天然表达的报告基因,因为报告基因的表达被用作基因摄取成功的标志。通常用于诱导可视化识别特征的常用报告基因通常涉及荧光和发光蛋白;例如包括编码水母绿色荧光蛋白(GFP)的基因,该基因导致表达它的细胞在蓝光下发出绿色荧光,以及编码萤光素酶的基因,该基因催化与萤光素反应产生光,以及来自基因dsRed的红色荧光蛋白。细菌中另一个常见的报告基因是Lac Z基因,它编码β-半乳糖苷酶蛋白。这种酶导致表达该基因的细菌在含有底物类似物X-gal的培养基上生长时呈蓝色。细菌中选择性标记报告基因的例子是氯霉素乙酰转移酶(CAT)基因,它赋予对氯霉素抗性。
报告基因还可以用来检测感兴趣基因的表达,该基因可能产生对细胞培养或生物体没有明显或直接影响的蛋白质。在这些情况下,报告基因直接连接到感兴趣基因以创建基因融合。这两个基因在相同的启动子元件下,被转录成单个信使RNA分子。然后,mRNA被翻译成蛋白质。在这些情况下,重要的是两种蛋白质能够正确折叠成它们的活性构象并与它们的底物相互作用,尽管它们是融合的。在构建DNA构建体时,通常包含一个编码灵活多肽连接子区域的DNA片段,以便报告基因和基因产物仅在最小程度上相互干扰。[23]
实践中的基因表达和纯化
蛋白质表达在生物化学中至关重要,因为它提供了用于进一步分析所需的底物或酶。在进行大规模蛋白质表达之前,通常会先进行小规模表达检查。BL21感受态大肠杆菌是常用的蛋白质表达感受态细胞。它对某些抗生素(如卡那霉素)具有抗性;它可以进行修饰以表达感兴趣的蛋白质。
在表达检查中,将所需的基因接种并在含有相应抗生素的5ml适当培养基中过夜表达。过夜表达后,将含有所需蛋白质的培养基离心。在去除上清液后,将沉淀物悬浮在适当的裂解缓冲液中并进行超声处理。超声处理后,将样品离心。将可溶性部分和不可溶性部分用于SDS凝胶电泳分析。必须提前知道和计算所需蛋白质的大致大小。如果在SDS凝胶电泳中显示出所需条带,则可以进行大规模蛋白质表达。

在大规模蛋白质表达中,通常使用3升培养瓶进行接种和诱导。在开始时,需要通过在5ml至25ml的灭菌培养基中接种已修饰的蛋白质表达感受态细胞来制备感兴趣基因的起始培养物。常用的培养基是LB,TB等。起始培养物需要在适当的温度(如37摄氏度)下孵育,并伴随充分摇动过夜。在同一天,可以准备升的培养基。在LB培养基的情况下,每升去离子水中需要25克LB。将培养瓶用铝箔封好并进行高压灭菌。接种前,LB需要保持用铝箔覆盖以保持无菌。在接种当天,培养基需要冷却至至少室温。将适当的抗生素添加到培养基中并充分摇动。接种是通过将5-10ml起始培养物添加到每升培养基中进行的。然后将培养基置于37度摇床中。跟踪接种培养物的吸光度至关重要。所需的吸光度为0.6。在大肠杆菌的情况下,这种吸光度大约在接种3小时后获得。大肠杆菌每20分钟复制一次;然而,在抗生素存在的情况下,这种复制时间可能会更长。但3小时的接种时间是安全的。3小时后,应仔细监测培养基的吸光度。如果OD过低,诱导可能不足;而如果OD过高,我们可能会获得不想要的蛋白质,因此所需的OD为0.6。在OD达到0.6后,在诱导之前需要将培养基在冰上冷却。IPTG通常用于诱导BL21感受态细胞。1mM的IPTG足以用于诱导。诱导温度可能与接种温度不同。诱导需要过夜。
第二天,将培养基离心成沉淀物。需要通过法国压机或微流化仪裂解沉淀物,具体取决于可用沉淀物的数量。法国压机通常适用于2升培养物裂解,而微流化仪更适合于任何超过2升的培养物裂解。实际选择还取决于可用设备以及沉淀物在裂解缓冲液中的溶解度。
在将沉淀物悬浮在裂解缓冲液中后,加入溶菌酶、DNase和RNase,并至少孵育10分钟。如果添加任何这些物质的数量不足,沉淀物在裂解过程中会显得粘稠,裂解可能不完全。裂解后,将样品离心以获得含有我们所需蛋白质的可溶性部分。重要的是要保持整个裂解过程处于冷态,因为某些蛋白质可能在室温下沉淀,或者机器变热会导致蛋白质损失。利用含有我们感兴趣蛋白质的可溶性部分,可以进行进一步的纯化。例如盐析、离子交换和亲和层析。进一步的纯化可能涉及FPLC。
适当的裂解缓冲液在蛋白质表达中至关重要。不同的质粒在不同的培养基中表达不同。温度和pH也是需要考虑的重要因素。
参考文献
[edit | edit source]- ↑ 基因表达
- ↑ 转录(遗传学)
- ↑ Beadle GW, Tatum EL (1941年11月15日). “遗传控制着真菌中的生化反应”. PNAS 27 (11): 499–506.
- ↑ 一个基因一个酶假说
- ↑ 操纵子
- ↑ http://en.wikipedia.org/w/index.php?title=Riboswitch&oldid=420979855
- ↑ 乳糖操纵子
- ↑ http://en.wikipedia.org/w/index.php?title=Lac_repressor&oldid=426589639
- ↑ http://en.wikipedia.org/w/index.php?title=Trp_operon&oldid=428240917
- ↑ 基因表达
- ↑ 质粒
- ↑ 聚合酶链反应
- ↑ 聚合酶链反应
- ↑ Pingoud A, Jeltsch A (2001年9月). "II型限制性内切酶的结构和功能". Nucleic Acids Res. 29 (18): 3705–27. doi:10.1093/nar/29.18.3705. PMC 55916. PMID 11557805.
- ↑ David P. Clark 著,分子生物学:理解基因革命,Elsevier Academic Press 出版,2005年。 ISBN 0-12-175551-7.
- ↑ Goodsell DS (2002). "分子视角:限制性内切酶". Stem Cells. 20 (2): 190–1. doi:10.1634/stemcells.20-2-190. PMID 11897876.
- ↑ Roberts RJ (1980年1月). "限制性内切酶和修饰酶及其识别序列". Nucleic Acids Res. 8 (1): r63–r80. doi:10.1093/nar/8.1.197-d. PMC 327257. PMID 6243774.
- ↑ R.J Roberts,1988,Nucl Acids Res. 16(增刊):271 摘自Lodish、Berk、Zipursky、Matsudaira、Baltimore 和 Darnell 编著的《分子细胞生物学》第四版,第 213 页。
- ↑ "来自链霉菌 tubercidicus 的 Stu I". Sigma-Aldrich. 检索于 2008-06-07.
{{cite web}}: 引用中存在空值未知参数:|coauthors=(帮助) - ↑ Shimotsu H, Takahashi H, Saito H (1980年11月). "来自链霉菌 tubercidicus 的一种新的位点特异性内切酶 StuI". Gene. 11 (3–4): 219–25. doi:10.1016/0378-1119(80)90062-1. PMID 6260571.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Copeland NG, Jenkins NA, Court DL (2001). "重组工程:小鼠功能基因组学中的一种强大新工具". Nat. Rev. Genet. 2 (10): 769–79. doi:10.1038/35093556. PMID 11584293.
{{cite journal}}: 未知参数|month=被忽略 (帮助)CS1 maint: multiple names: authors list (link) - ↑ Lu JP, Beatty LK, Pinthus JH. (2008). "基于双表达重组酶(DERB)的单载体系统,用于活细胞中蛋白质相互作用的高通量筛选和验证". Nature Precedings.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ http://en.wikipedia.org/w/index.php?title=Reporter_gene&oldid=422027341

