分子生物学导论/DNA复制及其修复

我们知道细胞分裂对于生物体的生长至关重要,但是当细胞分裂时,它必须复制基因组中的DNA(DNA复制发生在S期),这样两个子细胞才能与亲代细胞具有相同的遗传信息。DNA的双链结构为DNA复制提供了一个简单的机制。在这里,两条链被分离,然后每条链的互补DNA序列通过一种叫做DNA聚合酶的酶被重新创建。这种酶通过互补碱基配对找到正确的碱基,并将它与原始链连接起来,从而构建互补链。由于DNA聚合酶只能以5′到3′的方向延伸DNA链,因此不同的机制被用来复制双螺旋的两个反向平行的链。通过这种方式,旧链上的碱基决定了新链上出现哪个碱基,细胞最终得到了它DNA的完美副本。

a: 模板,b: 前导链,c: 滞后链,d: 复制叉,e: 引物,f: 冈崎片段
在细胞中,DNA复制从基因组中特定的位置开始,称为"起点"。DNA在起点处解开,并合成新的链,形成复制叉。除了DNA聚合酶(通过添加与模板链匹配的核苷酸来合成新DNA的酶)之外,还有一些其他蛋白质与叉相关联,并协助DNA合成的起始和继续。DNA复制也可以在体外(在细胞外)进行。从细胞中分离出来的DNA聚合酶和人工DNA引物被用来在模板分子中的已知序列处启动DNA合成。聚合酶链式反应 (PCR) 是一种常见的实验室技术,它以循环方式利用这种人工合成来从DNA池中扩增特定的目标DNA片段。[1]
前导链模板是DNA双螺旋的模板链,其方向为3'到5'。所有的DNA合成都是5'-3'。原始的DNA链必须以3'-5'的方向读取,以产生5'-3'的新生链。前导链是沿着前导链模板形成的,因为聚合酶"读取"模板DNA并不断地将核苷酸添加到延伸链的3'端。这种聚合酶在原核生物中是DNA聚合酶III(DNA Pol III),推测在真核生物中是Pol ε。

滞后链模板是DNA双螺旋的编码链,其方向为5'到3'。新生成的滞后链仍然是5'-3'合成的。但是,由于DNA的方向不允许连续合成,因此一次只能读取一小段。在复制起点3'端放置一个RNA引物。和以前一样,DNA聚合酶以3'-5'的方向读取原始DNA,以产生5'-3'的新生链。聚合酶到达复制起点并停止复制,直到在最后一个RNA引物的3'端放置一个新的RNA引物。在滞后链上产生的这些DNA片段被称为冈崎片段。滞后链上原始DNA的方向阻止了连续合成。因此,滞后链的复制比前导链更复杂。在滞后链模板上,引物酶"读取"DNA并在上面添加RNA,形成短的、分离的片段。在真核生物中,引物酶是Pol α的内在成分。DNA聚合酶III或Pol δ延长了引物化的片段,形成了冈崎片段。真核生物中的引物移除也是由Pol δ完成的。在原核生物中,DNA聚合酶I"读取"片段,利用其瓣状核酸内切酶结构域去除RNA,并用DNA核苷酸替换RNA核苷酸(这是必要的,因为RNA和DNA使用略微不同的核苷酸)。DNA连接酶将这些片段连接在一起。
冈崎片段是在DNA复制过程中滞后链上产生的相对较短的DNA片段(在5'末端没有RNA引物)。在大肠杆菌中,冈崎片段的长度在1,000到2,000个核苷酸之间,而在真核生物中,通常在100到200个核苷酸之间。它最初是在1968年由冈崎令治、冈崎恒子及其同事在研究大肠杆菌中噬菌体DNA复制时发现的。[2][3]

活细胞中DNA复制的速度最初是通过测量噬菌体感染的大肠杆菌中噬菌体T4 DNA的延伸速度来测量的。[4] 在37 °C下指数DNA增加期间,速度为每秒749个核苷酸。噬菌体T4 DNA合成过程中每碱基对每复制的突变率为1.7乘以108。[5] 因此,半保留的DNA复制既快速又准确。
基于序列同源性,DNA聚合酶被细分为七个不同的家族:A、B、C、D、X、Y和RT。
1.A家族聚合酶包含复制性和修复性聚合酶。来自该家族的复制性成员包括经过广泛研究的T7 DNA聚合酶,以及真核线粒体DNA聚合酶γ。修复性聚合酶包括大肠杆菌DNA pol I、热球菌pol I和嗜热脂肪芽孢杆菌pol I。这些修复性聚合酶参与切除修复和处理滞后链合成过程中产生的冈崎片段。
2.B家族在XPV患者中,认为替代的易错聚合酶,例如Pol ζ(zeta)(聚合酶ζ是B家族聚合酶,是催化亚基REV3L与Rev7的复合体,与Rev1结合),参与了导致这些患者癌症易感性的错误。属于B家族的DNA聚合酶包含DTDS基序。其他成员包括Pol ε、Pol α、Pol δ。
3.C家族聚合酶是主要的细菌染色体复制酶。来自大肠杆菌的DNA聚合酶IIIα亚基是催化亚基,没有已知的核酸酶活性。一个单独的亚基,ε亚基,具有用于染色体复制过程中编辑的3'-5'外切核酸酶活性。最近的研究已将C家族聚合酶归类为X家族的一个子类别。
4. 家族 D 聚合酶的特性尚未完全了解。所有已知例子都存在于古菌的广古菌亚域,被认为是复制聚合酶。
5. 家族 X 包含著名的真核聚合酶 pol β,以及其他真核聚合酶,如 pol σ、pol λ、pol μ 和末端脱氧核苷酸转移酶 (TdT)。Pol β 是短片段碱基切除修复所必需的,碱基切除修复是一种 DNA 修复途径,对于修复无碱基位点至关重要。Pol λ 和 Pol μ 参与非同源末端连接,这是一种重新连接 DNA 双链断裂的机制。TdT 仅在淋巴组织中表达,并在 V(D)J 重组过程中形成的双链断裂处添加“n 核苷酸”,以促进免疫多样性。酵母菌 Saccharomyces cerevisiae 只有一个 Pol X 聚合酶 Pol IV,它参与非同源末端连接。
6. 家族 Y Y 聚合酶与其他聚合酶的不同之处在于它们在未损伤模板上的低保真性 以及它们穿过受损 DNA 复制的能力。因此,这个家族的成员被称为跨损伤合成 (TLS) 聚合酶。根据损伤的不同,TLS 聚合酶可以以无错误或易出错的方式绕过损伤,后者会导致突变率升高。例如,色素性干皮病变异体 (XPV) 患者在编码 Pol η (eta) 的基因中存在突变,该基因对于 UV 损伤是无错误的。人类的其他成员包括 Pol ι (iota)、Pol κ (kappa) 和 Rev1 (末端脱氧胞嘧啶转移酶)。在大肠杆菌中,已知两种 TLS 聚合酶,Pol IV (DINB) 和 Pol V (UmuD'2C)。
7. 家族 RT (逆转录酶) 逆转录酶家族包含来自逆转录病毒和真核聚合酶的例子。真核聚合酶通常限于端粒酶。这些聚合酶使用 RNA 模板合成 DNA 链。
复制是半保留的
[edit | edit source]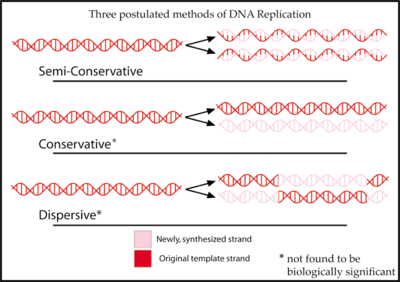
梅塞尔森-斯塔尔实验是由马修·梅塞尔森和弗兰克林·斯塔尔于 1958 年进行的一项实验,支持了DNA 复制是半保留的这一假设。半保留复制意味着当双链 DNA 螺旋被复制时,两个双链DNA 螺旋中的每一个都包含来自原始螺旋的一条链和一条新合成的链。它被称为“生物学中最美丽的实验”。[6]"
之前已经针对 DNA 复制方法提出了三种假设。
在半保留假设中,由沃森和克里克提出,DNA 分子的两条链在复制过程中分离。然后,每条链作为合成新链的模板。[7]
保守假设提出,整个 DNA 分子作为合成一个全新的分子的模板。根据这个模型,组蛋白 蛋白质结合到 DNA 上,以某种方式扭曲 DNA,以使两条链的碱基暴露出来进行氢键结合。[8]
分散假设由马克斯·德尔布吕克提出的模型来举例说明,该模型试图通过一种机制来解决解开双螺旋两条链的问题,该机制每隔 10 个核苷酸左右打断 DNA 主链,解开分子,并将旧链连接到新合成的链的末端。这将在短片段中合成 DNA,这些片段交替来自一条链到另一条链。[9]
这三种模型中的每一种都对复制后形成的分子中“旧” DNA 的分布做出了不同的预测。在保守假设中,复制后,一个分子是完全保守的“旧”分子,另一个是全部新合成的 DNA。半保留假设预测,复制后的每个分子都将包含一条旧链和一条新链。分散模型预测,每个新分子的每条链都将包含旧 DNA 和新 DNA 的混合物。[10]
可以通过利用 DNA 由氮碱基组成的事实来确认半保留理论。氮有一种同位素 N15(N14 是最常见的同位素),称为重氮。证实半保留理论预测的实验[11][12]利用了这种同位素,并按如下步骤进行:细菌(大肠杆菌)DNA 被放置在含有重氮 (N15) 的培养基中,重氮会结合到 DNA 上,使其可识别。然后将含有这种 DNA 的细菌放置在存在 N14 的培养基中,并使其仅复制一次。新的碱基将含有氮 14,而原始的将含有 N15。将 DNA 放置在含有氯化铯(重化合物)的试管中,并在每分钟 40,000 转的条件下离心。氯化铯分子沉到底部试管,形成密度梯度。DNA 分子将在其相应的密度水平处定位(考虑到 N15 比 N14 密度更大)。在紫外线下观察这些试管。DNA 在试管中以不同高度的细层出现,这取决于它们的密度。根据半保留理论,在 DNA 复制一次后,我们应该从 DNA 的每条原始链获得 2 个杂合(部分 N14 部分 N15)分子。这将出现在试管中的一条线上。分散理论的结果将相同。另一方面,根据保守理论,我们应该获得一个原始的 DNA 双链体和一个全新的双链体,即试管中放置的两个彼此分开的细线。到目前为止,半保留理论或分散理论可能是正确的,因为实验证据证实,在复制一次后只出现了一条线。为了在两者之间得出结论,DNA 必须再次复制,仍然在含有 N14 的培养基中。在分散理论中,在两次分裂后,我们应该获得一条线,但在试管中更高的地方,因为随着 N14 在分子中变得更加丰富,DNA 分子变得不那么稠密。根据半保留理论,应该产生 2 个杂合分子和 2 个完全的 N14 分子,因此应该在试管中观察到两个不同高度的细线。实验证据证实观察到两条线,因此为半保留理论提供了令人信服的证据。
遗传证据
最近,通过对单个突变细菌进行高通量基因组测序,提供了半保留理论的独立“遗传”证据。大肠杆菌用甲基磺酸乙酯 (EMS) 处理,已知由于生成异常碱基 O-6-乙基鸟嘌呤,会诱导 G:C → A:T 转变,该碱基在 DNA 复制过程中被错误识别,并与 T 配对而不是 C。来自 EMS 诱变细菌的单个菌落的测序 DNA 表现出仅 G → A 或 C → T 转变的长片段,在某些情况下跨越整个细菌基因组。这种观察结果的基本解释是基于半保留机制:人们应该期望在复制后子代链分离到不同的细胞中,这导致每个后代细胞仅具有 G → A 或 C → T 转变。
{{quotation|

氮是 DNA 的主要成分。14N 是迄今为止最丰富的氮同位素,但含有较重(但非放射性)15N 同位素的 DNA 也是有效的。
大肠杆菌 在含有15N 的培养基中生长了几代。当从这些细胞中提取 DNA 并将其在盐密度梯度上离心时,DNA 在其密度等于盐溶液密度的位置处分离出来。在15N 培养基中生长的细胞的 DNA 比在正常14N 培养基中生长的细胞的 DNA 密度更高。之后,将仅在 DNA 中含有15N 的大肠杆菌细胞转移到14N 培养基中,并使其分裂;通过测量细胞悬浮液的光密度来监测细胞分裂的进程。
定期提取 DNA,并将其与纯14N DNA 和15N DNA 进行比较。发现复制一次后,DNA 的密度接近中间密度。由于保守复制会导致密度较高和密度较低的 DNA 等量(但没有中间密度的 DNA),因此排除了保守复制。然而,该结果与半保留复制和分散复制都一致。半保留复制会导致一条链为15N DNA,另一条链为14N DNA 的双链 DNA,而分散复制会导致两条链都含有15N 和14N DNA 混合物的双链 DNA,这两种情况都将显示为中间密度的 DNA。
研究人员在复制过程中持续取样细胞。发现完成两次复制后的细胞中的 DNA 由等量的两种不同密度的 DNA 组成,其中一种对应于仅在一个14N 培养基中分裂一次的细胞的 DNA 的中间密度,另一种对应于仅在14N 培养基中生长的细胞的 DNA。这与分散复制不一致,分散复制会导致单个密度,该密度低于一代细胞的中间密度,但仍高于仅在14N DNA 培养基中生长的细胞,因为原始的15N DNA 将均匀地分布在所有 DNA 链中。结果与半保留复制假说一致[11]
原核生物中的复制
[edit | edit source]原核生物中的 DNA 复制在 E. coli 中得到了广泛的研究。它是双向的,起源于单个复制起点 (OriC)。
引物酶
[edit | edit source]在细菌中,引物酶与 DNA 解旋酶结合形成称为引物体的复合物。引物酶被 DNA 解旋酶激活,然后合成一个大约 11 ±1 个核苷酸长的短 RNA 引物,新的核苷酸可以由 DNA 聚合酶添加到该引物上。
引物体
[edit | edit source]引物体是一种蛋白质复合物,负责在 DNA 复制过程中在单链 DNA 上创建 RNA 引物。引物体是核蛋白组装体,激活 DNA 复制叉。它们的主要作用是将复制性解旋酶招募到单链 DNA 上。“复制重启”引物体,在 Escherichia coli 中被定义,参与重新激活停滞的复制叉。
Escherichia coli 引物体的组装需要六种蛋白质,PriA、PriB、PriC、DnaB、DnaC 和 DnaT,它们在包被 SSB 的单链 (8s) DNA 上的引物体组装位点 (pas) 上起作用。组装由 PriA 和 PriB 与 ssDNA 和 pas 的相互作用启动。PriC、DnaB、DnaC 和 DnaT 然后作用于 PriAPriB-DNA 复合物,产生引物体。
引物体包含 七种蛋白质:DnaG 引物酶、DnaB 解旋酶、DnaC 解旋酶辅助蛋白、DnaT、PriA、Pri B 和 PriC。引物体在 DNA 的引导链上使用一次,并重复使用,在滞后 DNA 链上启动每个冈崎片段。最初由 PriA、PriB 和 PriC 形成的复合物与 DNA 结合。然后 DnaB-DnaC 解旋酶复合物与 DnaT 一起附着。这种结构被称为 前引物体。最后,DnaG 将与前引物体结合形成完整的引物体。引物体将 1-10 个 RNA 核苷酸连接到单链 DNA 上,形成 DNA-RNA 杂合体。这个 RNA 序列被用作引物来启动 DNA 聚合酶 III。RNA 碱基最终被 RNase H 核酸酶(真核生物)或 DNA 聚合酶 I 核酸酶(原核生物)替换为 DNA 碱基。然后 DNA 连接酶作用于将两个末端连接在一起。
DNA 链的延伸
[edit | edit source]一旦引物完成,DNA 聚合酶 III 全酶就被加载到 DNA 中,复制开始。DNA 聚合酶 III 的催化机制涉及在活性位点使用两个金属离子,以及活性位点中能够区分脱氧核糖核苷酸和核糖核苷酸的区域。金属离子是一般二价阳离子,有助于 3' OH 启动对脱氧核糖核苷酸的α磷酸的亲核攻击,并定向和稳定脱氧核糖核苷酸上带负电荷的三磷酸。3' OH 对α磷酸的亲核攻击释放焦磷酸,然后焦磷酸被无机磷酸酶水解成两个磷酸。这种水解推动 DNA 合成完成。
此外,DNA 聚合酶 III 必须能够区分正确配对的碱基和错误配对的碱基。这是通过利用一个形状与正确配对的核苷酸结构互补的活性位点口袋来区分沃森-克里克碱基对来实现的。该口袋有一个酪氨酸残基,能够与正确配对的核苷酸形成范德华相互作用。此外,活性位点中的 dsDNA(双链 DNA)具有更宽且更浅的次要沟槽,允许与嘌呤碱基的第三个氮原子和嘧啶碱基的第二个氧原子形成氢键。最后,活性位点与 DNA 骨架形成广泛的氢键。这些相互作用导致 DNA 聚合酶 III 围绕正确配对的碱基闭合。如果插入一个碱基并错误配对,则这些相互作用将无法发生,因为氢键和范德华相互作用会受到破坏。
DNA 在 3' → 5' 方向上读取,因此核苷酸在 5' → 3' 方向上合成(或连接到模板链)。但是,DNA 的一条亲本链是 3' → 5',而另一条是 5' → 3'。为了解决这个问题,复制在相反的方向上进行。朝向复制叉移动,引导链以连续的方式合成,只需要一个引物。另一方面,滞后链朝远离复制叉的方向移动,以一系列称为冈崎片段的短片段合成,因此需要许多引物。冈崎片段的 RNA 引物随后被 RNAse H 和 DNA 聚合酶 I(核酸外切酶)降解,间隙(或切口)被脱氧核糖核苷酸填充,并由连接酶封闭。[13]
终止
[edit | edit source]E. coli 中的 DNA 复制终止是通过使用终止序列和Tus 蛋白来完成的。Tus 是一种序列特异性 DNA 结合蛋白,它促进原核生物 DNA 复制中的终止。在E. Coli 中,Tus 结合细菌染色体中编码的十个密切相关的 23 个碱基对结合位点。这些位点被称为Ter 位点,被命名为TerA、TerB、...、TerJ。结合位点是不对称的,因此当复制叉从一个方向遇到 Tus-Ter 复合物(Tus 蛋白与 Ter 位点结合)时,复合物会解离,复制继续(允许)。但是,当从另一个方向遇到时,Tus-Ter 复合物会提供更大的动力学障碍并停止复制(不允许)。染色体中的多个 Ter 位点是定向的,这样两个相反方向移动的复制叉都停滞在所需的终止区域。
原核生物中的 DNA 聚合酶
[edit | edit source]
在原核生物中,有 5 种 DNA 聚合酶
Pol I:参与 DNA 修复;具有 5'->3' 聚合酶活性,以及 3'->5' 核酸外切酶活性(校对)和 5'->3' 核酸外切酶活性(RNA 引物去除)。
Pol II:参与修复受损的 DNA;具有 3'->5' 核酸外切酶活性。该酶的大小为 90 kDa,由 polB 基因编码。DNA Pol II 可以以平均每秒 40 到 50 个核苷酸的速度合成新的 DNA 碱基对。
Pol III:细菌中的主要聚合酶(负责延伸);具有 3'->5' 核酸外切酶活性(校对)。复制体由以下组成:2 个 DNA Pol III 酶,由 α、ε 和 θ 亚基组成。α 亚基合成 RNA/DNA 引物。ε 亚基合成引导链。θ 亚基刺激 ε 亚基的校对。2 个 β 亚基充当滑动 DNA 夹,它们使聚合酶与 DNA 结合。2 个 τ 亚基的作用是使两个核心酶(α、ε 和 θ 亚基)二聚化。1 个 γ 亚基充当滞后链冈崎片段的夹子加载器,帮助两个 β 亚基形成一个单元并与 DNA 结合。γ 单元由 5 个 γ 亚基组成,包括 3 个 γ 亚基、1 个 δ 亚基和 1 个 δ' 亚基。δ 参与滞后链的复制
Pol IV:一种 Y 家族 DNA 聚合酶。
Pol V:一种 Y 家族 DNA 聚合酶;参与绕过 DNA 损伤。
DNA 聚合酶 I 或 Pol I
[edit | edit source]DNA 聚合酶 I(或 Pol I)是一种参与原核生物 DNA 复制过程的酶。它包含 928 个氨基酸,是连续酶的一个例子——它可以依次催化多个聚合反应。它是由Arthur Kornberg 在 1956 年发现的,它是第一个已知的 DNA 聚合酶(也是第一个已知的任何类型的聚合酶)。它最初在 E. coli 中被描述,尽管它在原核生物中普遍存在。在 E. coli 和许多其他细菌中,编码Pol I 的基因被称为 polA。Pol I 具有三种酶活性:(1)5' -> 3'(正向)DNA 聚合酶活性,需要 3' 引物位点和模板链;(2)3' -> 5'(反向)核酸外切酶活性,介导校对;以及(3)5' -> 3'(正向)核酸外切酶活性,介导 DNA 修复过程中的切口翻译。
Klenow 片段
[edit | edit source]大肠杆菌DNA聚合酶I的5'→3'外切酶活性使其不适合许多应用,而缺少这种活性的Klenow片段在研究中非常有用。Klenow片段对于基于研究的任务非常有用,例如:(1) 从单链模板合成双链DNA;(2) 填补(即去除突出部分以创建平端) DNA片段的凹陷3'端;(3) 消化突出的3'突出部分;(4) 制备放射性DNA探针。Klenow片段也是最初用于在聚合酶链反应(PCR)过程中极大地扩增DNA片段的酶,后来被耐热酶(如Taq聚合酶)取代。
许多细胞过程(DNA复制、转录、翻译、重组、DNA修复、核糖体生物合成)都涉及核酸链的分离。解旋酶通常被用来分离DNA双螺旋或自退火的RNA分子的链,利用来自ATP水解的能量,这个过程的特点是打破退火核苷酸碱基之间的氢键。它们以特定于每种特定酶的方向性和过程性,逐步沿着双链体的一个核酸链移动。由于必须催化链分离的各种过程,因此存在许多解旋酶(大肠杆菌中已确认14种,人类细胞中已确认24种)。
解旋酶采用不同的结构和寡聚化状态。虽然DnaB样解旋酶以环状六聚体形式解开DNA,但其他酶已被证明以单体或二聚体形式发挥作用。研究表明,解旋酶可能被动地起作用,等待非催化解开发生,然后在位移的链之间易位,[1]或可以在利用ATP水解产生的能量催化链分离中发挥积极作用。在后一种情况下,解旋酶的作用类似于一个主动马达,解开并沿着其底物易位,这是其ATP酶活性直接的结果。解旋酶在体内可能比在体外处理得更快,这是由于存在辅助蛋白质,这些蛋白质有助于不稳定叉连接。解旋酶编码基因的缺陷会导致Werner综合征,这是一种以早衰为特征的疾病。[14]
超家族
解旋酶已被归类为5个超家族(SF1-SF5)。所有这些蛋白质都结合ATP,因此,它们都带有经典的Walker A(磷酸结合环或P-环)和Walker B(Mg2+-结合天冬氨酸)基序。
超家族I: UvrD(大肠杆菌,DNA修复),Rep(大肠杆菌,DNA复制),PcrA(金黄色葡萄球菌,重组),Dda(T4噬菌体,复制起始),RecD(大肠杆菌,重组修复),TraI(F-质粒,结合性DNA转移)。该家族包括被认为参与病毒RNA复制过程中双链解开的RNA解旋酶。该家族的成员存在于来自超家族1的正链单链RNA病毒中。这种解旋酶在病毒RNA复制的不同阶段具有多种作用,正如突变分析所揭示的。
超家族II: RecQ(大肠杆菌,DNA修复),eIF4A(面包酵母,RNA翻译),WRN(人,DNA修复),NS3[5](丙型肝炎病毒,复制)。TRCF(Mfd)(大肠杆菌,转录-修复耦合)。
超家族III: LTag(猿猴病毒40,复制),E1(人乳头瘤病毒,复制),Rep(腺病毒相关病毒,复制,病毒整合,病毒体包装)。超家族3主要由小DNA病毒和一些大型核质DNA病毒编码的解旋酶组成。[6][7]小病毒在复制方面非常依赖于宿主细胞机制。小病毒中的SF3解旋酶与一个起始点结合域相关。通过将识别起始点的域与一个解旋酶配对,病毒可以绕过基于宿主细胞的调节途径并启动自身的复制。该蛋白质与病毒起始点结合,导致起始点解开。然后,细胞复制蛋白被招募到起始点,病毒DNA被复制。
DnaB样家族: dnaB(大肠杆菌,复制),gp41(T4噬菌体,DNA复制),T7gp4(T7噬菌体,DNA复制)。
Rho样家族: Rho(大肠杆菌,转录终止)。注意,这些超家族并没有包含所有可能的解旋酶。例如,XPB和ERCC2是未包含在上述任何家族中的解旋酶。
RNA解旋酶 RNA解旋酶和DNA解旋酶可以在所有解旋酶超家族中找到,除了SF6。[15] 然而,并非所有RNA解旋酶都表现出通过酶功能定义的解旋酶活性,即Swi/Snf家族的蛋白质。虽然这些蛋白质带有典型的解旋酶基序,以核酸依赖的方式水解ATP,并围绕解旋酶核心构建,但通常观察不到解开活性。[16]
表现出解开活性的RNA解旋酶至少通过两种不同的机制进行表征:规范双链解开和局部链分离。规范双链解开是如上所述的双链体逐步定向分离,用于DNA解开。然而,局部链分离通过一种机制发生,其中解旋酶被加载到双链体上的任何位置。这通常由RNA的单链区域辅助,酶的加载伴随着ATP结合。[17] 一旦解旋酶和ATP结合,局部链分离就会发生,这需要ATP结合,但不需要ATP水解的实际过程。[18] 当遇到更少的碱基对时,双链体就会在没有酶的进一步帮助的情况下解离。这种解开模式被DEAD-box解旋酶使用。[19]
拓扑异构酶
拓扑异构酶是解开和缠绕DNA的酶,为了让DNA控制蛋白质的合成,并促进DNA复制。DNA链自然存在的双螺旋构型使它们难以分离,然而,如果其他酶要转录编码蛋白质的序列,或者如果染色体要被复制,它们必须被解旋酶蛋白分离。在所谓的环状DNA中,双螺旋DNA弯曲并连接成一个圆圈,两条链在拓扑上连接在一起,或者打结在一起。否则,具有不同扭曲次数的相同DNA环是拓扑异构体,不能通过任何不涉及DNA链断裂的过程相互转换。拓扑异构酶通过使用保守的酪氨酸作为催化残基,在DNA中产生短暂的断裂,催化和指导DNA的解结或解缠结。[3]将病毒DNA插入染色体和其他形式的重组也可能需要拓扑异构酶的作用。
拓扑异构酶可以解决这些拓扑问题,并根据一轮作用中切割的链数分为两种类型:[20] 这两类酶都利用了保守的酪氨酸。然而,这些酶在结构和机制上是不同的。
- I型拓扑异构酶 切割DNA双螺旋的一条链,发生松弛,然后切割的链被重新退火。切割一条链允许分子在切割一侧的部分绕着未切割的链旋转,从而减少螺旋过紧或过松造成的应力。当DNA链被“超螺旋”或从更高阶的螺旋解开或缠绕时,就会引入这种应力。I型拓扑异构酶细分为两个亚类:IA型拓扑异构酶,它们与II型拓扑异构酶共享许多结构和机制特征,以及IB型拓扑异构酶,它们利用受控的旋转机制。IA型拓扑异构酶的例子包括topo I和topo III。在过去,IB型拓扑异构酶被称为真核topo I,但IB型拓扑异构酶存在于生命的三大域中。有趣的是,IA型拓扑异构酶与DNA的5'端形成共价中间体,而IB型拓扑异构酶与DNA的3'端形成共价中间体。最近,一种IC型拓扑异构酶被鉴定出来,称为topo V。虽然它在结构上与IA型和IB型拓扑异构酶不同,但它与IB型拓扑异构酶共享类似的机制。
- II型拓扑异构酶 切割一个DNA双螺旋的两条链,将另一条完整的DNA螺旋通过它,然后重新退火切割的链。它也被分为两个亚类:IIA型和IIB型拓扑异构酶,它们共享相似的结构和机制。IIA型拓扑异构酶的例子包括真核topo II、大肠杆菌gyrase和大肠杆菌topo IV。IIB型拓扑异构酶的例子包括topo VI。
| 拓扑异构酶 | IA | IB | IIA | IIB |
|---|---|---|---|---|
| 金属依赖性 | 是 | 否 | 是 | 是 |
| ATP依赖性 | 否 | 否 | 是 | 是 |
| 单链还是双链切割? | SS | SS | DS | DS |
| 切割极性 | 5' | 3' | 5' | 5' |
| L变化 | ±1 | ±N | ±2 | ±2 |
I型和II型拓扑异构酶都会改变DNA的连接数(L)。IA型拓扑异构酶将连接数改变一个,IB型和IC型拓扑异构酶将连接数改变任何整数,而IIA型和IIB型拓扑异构酶将连接数改变两个。
许多药物通过干扰拓扑异构酶发挥作用。广谱氟喹诺酮类抗生素通过破坏细菌II型拓扑异构酶的功能起作用。一些化疗药物通过干扰癌细胞中的拓扑异构酶发挥作用:1型被伊立替康和托泊替康抑制。2型被依托泊苷(VP-16)、替尼泊苷和HU-331抑制,HU-331是一种从大麻二酚合成的喹诺酮类药物。拓扑异构酶I是硬皮病中抗Scl-70抗体识别的抗原。这些小分子抑制剂通过利用拓扑异构酶在染色体DNA中产生断裂的自然能力,作为有效的抗菌和抗癌剂。这些DNA断裂累积,最终导致程序性细胞死亡,即细胞凋亡。


真核生物的 DNA 复制比原核生物复杂得多,尽管它们有许多相似之处。真核细胞只能在细胞周期的特定时间点,即 **S 期** 开始时启动 DNA 复制。
真核生物的 DNA 复制仅发生在细胞周期的 S 期。然而,预起始发生在 G1 期。因此,预起始和活化的分离确保每个细胞周期中复制起点只能启动一次。由于真核生物染色体体积巨大,真核生物染色体包含 **多个复制起点**。一些复制起点得到了很好的描述,例如酵母的 **自主复制序列 (ARS)**,而其他真核复制起点,特别是后生动物中的复制起点,可以在数千个碱基对的范围内找到。[21]
至少有 15 种已知的真核 DNA 聚合酶
POLA1、POLA2: Pol α (也称为 RNA 引物酶): 与一个小的催化 (PriS) 亚基和一个大的非催化 (PriL) 亚基形成复合物,Pri 亚基充当引物酶 (合成 RNA 引物),然后 DNA Pol α 用 DNA 核苷酸延长该引物。大约 20 个核苷酸后[3],Pol ε (在引导链上) 和 δ (在滞后链上) 接管了延伸。
POLB: Pol β: 参与 DNA 修复,包括碱基切除修复和缺口填补合成。
POLG、POLG2: Pol γ: 复制和修复线粒体 DNA,并具有校对 3'->5' 核酸外切酶活性。
POLD1、POLD2、POLD3、POLD4: Pol δ: 高度加工,并具有校对 3'->5' 核酸外切酶活性。被认为是参与滞后链合成的主要聚合酶,尽管关于其作用仍存在争议。
POLE、POLE2、POLE3: Pol ε: 也是高度加工,并具有校对 3'->5' 核酸外切酶活性。与 Pol δ 高度相关,被认为是参与引导链合成的主要聚合酶[5],尽管关于其作用仍存在争议。
POLH、POLI、POLK、 : η、ι、κ 和 Rev1 是 Y 族 DNA 聚合酶,Pol ζ 是 B 族 DNA 聚合酶。这些聚合酶参与绕过 DNA 损伤。
还有一些其他已知的真核聚合酶,但没有得到很好的描述:POLQ: 'θ POLL: λ φ σ POLM: μ 没有任何真核聚合酶可以去除引物 (5'->3' 核酸外切酶活性);该功能由其他酶执行。只有处理延伸的聚合酶 (γ、δ 和 ε) 具有校对能力 (3'->5' 核酸外切酶)。
G1 期的准备
DNA 复制的第一步是形成预起始复制复合物 (Pre-RC)。该复合物的形成分为两个阶段。第一阶段要求没有 CDK 活性。这只能发生在 G1 期早期。Pre-RC 的形成被称为许可,但许可的 Pre-RC 不能在 G1 期 启动复制。当前的模型认为,它始于复制起点识别复合物 (ORC) 与复制起点的结合。该复合物是由相关蛋白组成的六聚体,即使在 DNA 复制发生后也保持与复制起点的结合。此外,ORC 是原核生物 DnaA 的功能类似物。在 ORC 与复制起点结合后,Cdc6/Cdc18 和 Cdt1 通过首先与 ORC 结合,然后与 MCM (微型染色体维持) 复合物结合来协调 MCM 复合物加载到复制起点。MCM 复合物被认为是真核生物中的主要 DNA 解旋酶。一旦 MCM 结合,就会存在一个完全许可的 Pre-RC。
复合物的活化发生在 S 期,需要 Cdk2-Cyclin E 和 Ddk。活化过程始于 Mcm10 添加到 Pre-RC,这会取代 Cdt1。之后,Ddk 磷酸化 Mcm3-7,这会激活解旋酶。据信 ORC 和 Cdc6/18 被 Cdk2-Cyclin E 磷酸化。Ddk 和 Cdk 复合物然后招募另一个称为 Cdc45 的蛋白,然后 Cdc45 招募所有 DNA 复制蛋白到复制叉。在这个阶段,复制起点启动,DNA 合成开始。通过周期蛋白依赖性激酶和称为 geminin 的蛋白的作用,防止新一轮复制的活化。Geminin 与 Cdt1 结合并将其隔离。它是一种周期性蛋白,首次出现在 S 期,并在 M 期后期降解,可能是通过后期促进复合物 (APC) 的作用。此外,Cdc6/18 的磷酸化会阻止它与 ORC 结合 (从而抑制 MCM 复合物的加载),而 ORC 的磷酸化尚不清楚。处于细胞周期 G0 期的细胞被阻止启动一轮复制,因为 Mcm 蛋白没有表达。
至少三种不同的真核 DNA 聚合酶参与动物细胞中的 DNA 复制 (POL α、Pol δ 和 POL ε)。
Pol α 与一个小的催化 (PriS) 亚基和一个大的非催化 (PriL) 亚基形成复合物,Pri 亚基充当引物酶 (合成 RNA 引物),然后 DNA Pol α 用 DNA 核苷酸延长该引物。大约 20 个核苷酸后,Pol ε (在引导链上) 和 δ (在滞后链上) 接管了延伸。
Pol δ: 高度加工,并具有校对 3'->5' 核酸外切酶活性。被认为是参与引导链合成的主要聚合酶,尽管关于其作用仍存在争议。
Pol ε: 也是高度加工,并具有校对 3'->5' 核酸外切酶活性。与 Pol δ 高度相关,被认为是参与滞后链合成的主要聚合酶。[22]

核 DNA 和线粒体 DNA 被认为具有不同的进化起源,mtDNA 来自被当今真核细胞祖先吞噬的细菌的环状基因组。该理论被称为内共生理论。每个线粒体估计包含 2-10 个 mtDNA 拷贝。在现存生物的细胞中,线粒体中存在的大多数蛋白 (在哺乳动物中大约有 1500 种不同的类型) 是由核 DNA 编码的,但其中一些蛋白 (如果不是大多数的话) 的基因被认为最初起源于细菌,在进化过程中转移到真核细胞核中。
mtDNA 由 DNA 聚合酶 γ 复合物复制,该复合物由 POLG 基因编码的 140 kDa 催化 DNA 聚合酶和 POLG2 基因编码的 55 kDa 辅助亚基组成。在胚胎发生过程中,mtDNA 的复制从受精卵细胞到植入前胚胎被严格下调。在囊胚阶段,mtDNA 复制的开始特定于滋养层细胞。相反,内细胞团的细胞会限制 mtDNA 复制,直到它们接收到分化为特定细胞类型的信号。D 环复制是叶绿体和线粒体复制其遗传物质的过程。理解 D 环复制的一个重要组成部分是叶绿体和线粒体与细菌一样只有一个环状染色体,而不是真核生物中发现的线性染色体。在许多生物体中,质体中的一个 DNA 链包含较重的核苷酸 (相对而言,嘌呤更多:腺嘌呤和鸟嘌呤)。这条链称为 H (重) 链。L (轻) 链包含较轻的核苷酸 (嘧啶:胸腺嘧啶和胞嘧啶)。复制从从 D 环 (也称为控制区) 开始的重链复制开始。一个复制起点打开,重链在一个方向上被复制。在重链复制持续一段时间后,通过另一个复制起点的打开,还合成一个新的轻链。当绘制图表时,生成的结构看起来像字母 D。D 环区域对于系统地理学研究很重要。由于该区域不编码任何基因,因此它可以自由变化,对大小和重链/轻链因子只有很少的选择性限制。突变率是动物核基因组或线粒体基因组中任何地方最快的突变率之一。D 环中的突变可以有效地跟踪最近和快速的变化,例如物种内和非常密切相关的物种之间。[23]
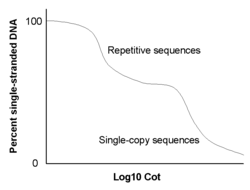
该技术最初由罗伊·布里滕及其同事于 20 世纪 60 年代在华盛顿卡内基研究所开发和应用。[24][25] 值得注意的是,正是通过 Cot 分析首次发现了 真核 基因组的冗余(重复)性质。DNA 中的重复序列。[26] 然而,直到布里滕及其同事突破性的 DNA 重缔合动力学实验表明,并非所有 DNA 都编码基因。事实上,他们的实验表明,大多数真核基因组 DNA 由重复的非编码元件组成。通过快速稀释样品来测量单链和双链 DNA 的量,从而减缓重缔合速度,然后将 DNA 结合到羟基磷灰石柱上。该柱首先用低浓度的磷酸钠缓冲液洗涤,洗脱单链 DNA,然后用高浓度的磷酸盐洗涤,洗脱双链 DNA。然后使用分光光度计测量这两种溶液中 DNA 的量。由于单链 DNA 序列需要找到其互补链才能重新形成双螺旋,因此常见序列的重性化速度比罕见序列快。事实上,序列重缔合的速度与其在 DNA 样本中的拷贝数成正比。具有高度重复序列的样本将快速重性化,而复杂序列将缓慢重性化。然而,与其仅仅测量双链 DNA 的百分比与时间的关系,不如将重性化的量与 C0t 值进行比较。C0t 值是 C0(初始 DNA 浓度)、t(以秒为单位的时间)和一个取决于缓冲液中阳离子浓度的常数的乘积。重复 DNA 将在低 C0t 值下重性化,而复杂和独特的 DNA 序列将在高 C0t 值下重性化。
DNA 修复
[edit | edit source]由于环境因素和细胞内部的正常代谢过程,DNA 损伤每天发生在每个细胞的 1,000 到 1,000,000 个分子损伤之间。虽然这仅占人类基因组约 60 亿个碱基(30 亿个碱基对)的 0.000165%,但关键基因(如肿瘤抑制基因)中未修复的损伤会阻碍细胞执行其功能并明显增加肿瘤形成的可能性。
绝大多数 DNA 损伤影响双螺旋的初级结构;也就是说,碱基本身被化学修饰。这些修饰反过来会通过引入非天然化学键或不适合标准双螺旋的庞大加合物来破坏分子的规则螺旋结构。与蛋白质和 RNA 不同,DNA 通常缺乏三级结构,因此不会在该水平发生损伤或干扰。然而,DNA 是超螺旋的,并缠绕在称为组蛋白的“包装”蛋白质(在真核生物中)周围,这两种超结构都容易受到 DNA 损伤的影响。[27]
DNA 损伤类型
[edit | edit source]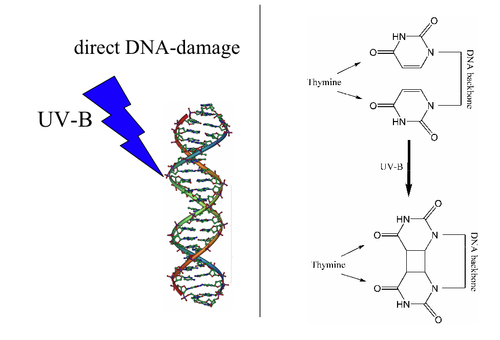
由于内源性细胞过程,DNA 损伤主要有五种类型:(1)碱基氧化 [例如 8-氧代-7,8-二氢鸟嘌呤 (8-oxoG)] 和活性氧物种产生的 DNA 链断裂;(2) 碱基烷基化(通常是甲基化),例如形成 7-甲基鸟嘌呤、1-甲基腺嘌呤、6-O-甲基鸟嘌呤;(3) 碱基水解,例如脱氨基、脱嘌呤和脱嘧啶;(4) “庞大加合物形成”(即苯并[a]芘二醇环氧化物 -dG 加合物);(5) 碱基错配,由于 DNA 复制错误,导致错误的 DNA 碱基被缝合到新形成的 DNA 链中,或者 DNA 碱基被跳过或错误地插入。由外源性因素引起的损伤由外源性因素引起的损伤有多种形式。以下列举了一些示例。
UV-B 光会导致相邻胞嘧啶和胸腺嘧啶碱基之间发生交联,形成嘧啶二聚体。这称为直接 DNA 损伤。
UV-A 光主要产生自由基。自由基造成的损伤称为间接 DNA 损伤。
电离辐射,如放射性衰变或宇宙射线产生的辐射,会导致 DNA 链断裂。低水平的电离辐射可能会诱导不可修复的 DNA 损伤(导致新生物形成所需的复制和转录错误,或可能触发病毒相互作用),从而导致过早衰老和癌症。
高温下的热扰动会增加脱嘌呤(DNA 骨架上的嘌呤碱基丢失)和单链断裂的速率。例如,在 40-80 °C 的温泉中生长的嗜热细菌中观察到水解脱嘌呤。在这些物种中,脱嘌呤的速率(每个基因组每个世代 300 个嘌呤残基)过高,无法通过正常的修复机制进行修复,因此无法排除适应性反应的可能性。
工业化学品在 DNA 损伤中也起着非常重要的作用,例如氯乙烯和过氧化氢,以及环境化学品,例如烟雾、煤烟和焦油中发现的多环烃,会产生大量不同的 DNA 加合物 - 乙烯碱基、氧化碱基、烷基化磷酸三酯和 DNA 交联,仅举几例。UV 损伤、烷基化/甲基化、X 射线损伤和氧化损伤是诱导损伤的例子。自发性损伤可能包括碱基丢失、脱氨基、糖环扭结和互变异构体位移。
损伤来源
[edit | edit source]DNA 损伤可以细分为两种主要类型
内源性损伤,例如由正常代谢副产物产生的活性氧物种的攻击,尤其是氧化脱氨基过程,这也包括由于复制错误造成的碱基错配
外源性损伤,由外部因素引起,例如
来自太阳的紫外线 [UV 200-300 纳米] 辐射
其他辐射频率,包括 X 射线和伽马射线
水解或热扰动
某些植物毒素
人造的诱变化学物质,尤其是作为 DNA 插入剂的芳香族化合物
癌症化疗和放射疗法
病毒
突变类型
[edit | edit source]当修复 DNA 损伤时,有时会导致简单的单碱基对突变,这里有描述。(在修复过程中也会出现缺失和易位)
转换 在分子生物学中,转换是指将嘌呤核苷酸变为另一个嘌呤(A ↔ G)或将嘧啶核苷酸变为另一个嘧啶(C ↔ T)的点突变。大约三分之二的单核苷酸多态性 (SNP) 是转换。转换可能是由氧化脱氨基和互变异构体引起的。虽然可能的颠换有两种,但转换在基因组中出现的频率更高,这可能是由于产生它们的分子机制所致。由于自发性脱氨基,5-甲基胞嘧啶比未甲基化的胞嘧啶更容易发生转换。这种机制很重要,因为它决定了 CpG 岛的稀有性。
颠换 在分子生物学中,颠换是指将嘌呤替换为嘧啶或反之。它只能通过自发的回复突变来逆转。由于这种类型的突变会显着改变化学结构,因此这种变化的后果往往比转换更严重且更不常见。颠换可能是由电离辐射和烷基化剂引起的。
DNA 修复和疾病
[edit | edit source]NER 机制的缺陷会导致许多遗传疾病,包括
色素性干皮病:对阳光/紫外线的超敏反应,导致皮肤癌发病率增加和过早衰老
科凯恩综合征:对紫外线和化学物质的超敏反应
毛发硫化营养不良:皮肤敏感,头发和指甲易碎。后两种疾病通常伴有智力障碍,表明发育中的神经元更容易受到损害。
其他 DNA 修复疾病包括
维尔纳综合征:过早衰老和生长迟缓
布鲁姆综合征:对阳光的超敏反应,恶性肿瘤(尤其是白血病)发病率高。
共济失调毛细血管扩张症:对电离辐射和某些化学物质的敏感性
以上所有疾病通常被称为“节段性早衰”(“加速衰老疾病”),因为它们的患者在异常年轻的时候就会显得衰老,并且患有与衰老相关的疾病,而不会表现出所有衰老的症状。
与 DNA 修复功能下降相关的其他疾病包括范科尼贫血、遗传性乳腺癌和遗传性结肠癌。
人类染色体和染色体畸变
[edit | edit source]染色体可以分为两种类型——常染色体和性染色体。某些遗传性状与你的性别有关,并通过性染色体遗传。常染色体包含其余的遗传遗传信息。它们在细胞分裂过程中以相同的方式起作用。人类细胞有 23 对大的线性核染色体(22 对常染色体和一对性染色体),每细胞总共 46 条。除了这些之外,人类细胞还有数百个线粒体基因组的拷贝。人类基因组测序为每条染色体提供了大量的信息。以下是根据脊椎动物基因组注释 (VEGA) 数据库中的桑格研究所人类基因组信息,汇编了染色体统计信息的表格。基因数量是一个估计值,因为它部分基于基因预测。总染色体长度也是一个估计值,基于未测序异染色质区域的估计大小。


| 染色体 | 基因 | 总碱基 | 测序碱基[28] |
|---|---|---|---|
| 1 | 4,220 | 247,199,719 | 224,999,719 |
| 2 | 1,491 | 242,751,149 | 237,712,649 |
| 3 | 1,550 | 199,446,827 | 194,704,827 |
| 4 | 446 | 191,263,063 | 187,297,063 |
| 5 | 609 | 180,837,866 | 177,702,766 |
| 6 | 2,281 | 170,896,993 | 167,273,993 |
| 7 | 2,135 | 158,821,424 | 154,952,424 |
| 8 | 1,106 | 146,274,826 | 142,612,826 |
| 9 | 1,920 | 140,442,298 | 120,312,298 |
| 10 | 1,793 | 135,374,737 | 131,624,737 |
| 11 | 379 | 134,452,384 | 131,130,853 |
| 12 | 1,430 | 132,289,534 | 130,303,534 |
| 13 | 924 | 114,127,980 | 95,559,980 |
| 14 | 1,347 | 106,360,585 | 88,290,585 |
| 15 | 921 | 100,338,915 | 81,341,915 |
| 16 | 909 | 88,822,254 | 78,884,754 |
| 17 | 1,672 | 78,654,742 | 77,800,220 |
| 18 | 519 | 76,117,153 | 74,656,155 |
| 19 | 1,555 | 63,806,651 | 55,785,651 |
| 20 | 1,008 | 62,435,965 | 59,505,254 |
| 21 | 578 | 46,944,323 | 34,171,998 |
| 22 | 1,092 | 49,528,953 | 34,893,953 |
| X(性染色体) | 1,846 | 154,913,754 | 151,058,754 |
| Y(性染色体) | 454 | 57,741,652 | 25,121,652 |
| 总计 | 32,185 | 3,079,843,747 | 2,857,698,560 |
染色体畸变是细胞正常染色体含量中的破坏,是人类遗传疾病(如唐氏综合征)的主要原因。一些染色体异常不会导致携带者患病,例如易位或染色体倒位,尽管它们可能会导致生出患有染色体疾病的孩子的几率更高。染色体或染色体组的异常数量,非整倍体,可能是致命的或导致遗传疾病。为可能携带染色体重排的家庭提供遗传咨询。
染色体中 DNA 的增加或减少会导致各种遗传疾病。人类例子包括
- 猫叫综合征,是由第 5 号染色体短臂部分缺失引起的。“猫叫综合征”在法语中意为“猫的哭声”,这种疾病因此得名,因为患病婴儿会发出类似猫叫的尖锐哭声。受影响的个体眼睛间距较宽,头部和下巴较小,中度至重度精神健康问题,而且个子非常矮。
- 唐氏综合征通常是由第 21 号染色体多了一份拷贝(21 三体)引起的。特征包括肌肉张力下降、体格矮胖、头骨不对称、斜眼和轻度至中度智力障碍。[29]
- 爱德华兹综合征,是第二常见的 trisomy;唐氏综合征是最常见的。它是 18 号染色体的 trisomy。症状包括运动迟缓、智力障碍以及许多导致严重健康问题的先天性畸形。90% 的患者在婴儿期死亡;然而,那些活过第一年生日的人通常此后身体非常健康。他们有一个典型的紧握的拳头和叠在一起的指头。
- Idic15,是 15 号染色体上等臂染色体 15 的缩写;由于各种研究,也被称为以下名称,但它们都表示相同的含义;IDIC(15)、倒位重复 15、额外标记、Inv dup 15、部分四体 15
- 雅各布森综合征,也称为 11q 末端缺失症。[30] 这是一个非常罕见的疾病。受影响的人智力正常或轻度智力障碍,表达语言能力较差。大多数人患有称为巴黎-特鲁索综合征的出血性疾病。
- 克莱恩费尔特综合征 (XXY)。患有克莱恩费尔特综合征的男性通常不育,而且往往手臂和腿更长,比同龄人更高。患有这种综合征的男孩往往害羞而安静,患有语言发育迟缓和阅读障碍的比例较高。在青春期,如果没有睾酮治疗,他们中的一些人可能会出现乳房发育。
- 帕陶综合征,也称为 D 综合征或 13 三体。症状与 18 三体有些相似,但没有典型的双手形状。
- 小型超数标记染色体。这意味着存在额外的异常染色体。特征取决于额外遗传物质的来源。猫眼综合征和15 号等臂染色体综合征(或 Idic15)都是由超数标记染色体引起的,帕利斯特-基利安综合征也是如此。
- 三 X 综合征 (XXX)。XXX 女孩子往往身材高挑,而且瘦。她们患有阅读障碍的比例更高。
- 特纳氏综合征(X 而不是 XX 或 XY)。在特纳氏综合征中,女性性征存在,但发育不良。特纳氏综合征患者通常身材矮小,发际线低,眼睛和骨骼发育异常,胸部呈“凹陷”状。
- XYY 综合征。XYY 男孩通常比他们的兄弟姐妹更高。与 XXY 男孩和 XXX 女孩一样,他们也更容易出现学习困难。
- 狼-赫施霍恩综合征,是由第 4 号染色体短臂部分缺失引起的。其特征是严重的生长迟缓和严重至极度严重的精神健康问题。
染色体突变会导致整个染色体(多个基因)或染色体数量发生改变。
- 缺失 – 染色体部分丢失
- 重复 – 染色体部分的额外拷贝
- 倒位 – 反转染色体部分的方向
- 易位 – 染色体的一部分断裂并连接到另一条染色体上
大多数突变是中性的 – 几乎没有或根本没有影响。染色体畸变是染色体结构的改变。它在进化中起着重要作用。在橡树岭国家实验室可以找到所有人类染色体和在正确位置注释的疾病的详细图形显示。[31]
DNA 重组
[edit | edit source]


重组是一个过程,在这个过程中,核酸分子(通常是 DNA,但也可能是 RNA)被断裂,然后与另一个分子连接(或者其中遗传信息在两个这样的分子之间交换)。重组通常发生在类似的 DNA 分子之间,例如在同源重组中。重组是细菌和真核生物中常见的 DNA 修复方法。在真核生物中,重组也发生在减数分裂中,它促进信息交换和/或染色体交叉。交叉过程导致后代具有不同于其父母的基因组合,并且偶尔会产生新的嵌合等位基因。在具有适应性免疫系统的生物体中,一种称为 V(D)J 重组的遗传重组类型有助于免疫细胞快速多样化以识别和适应新的病原体。遗传重组带来的基因重排具有长期优势,因为它是一个主要的遗传变异引擎,并且还允许有性繁殖的生物体避免穆勒氏棘轮,在这种棘轮中,无性繁殖种群的基因组以不可逆转的方式积累有害突变。在基因工程中,重组也可以指不同 DNA 片段的 人工和故意重组,这些片段通常来自不同的生物体,从而创造出所谓的重组 DNA。这种基因重组应用的一个主要例子是基因靶向,它可用于添加、删除或以其他方式改变生物体的基因。这项技术对于生物医学研究人员来说很重要,因为它使他们能够研究特定基因的影响。基于基因重组的技术也应用于蛋白质工程,以开发具有生物学意义的新蛋白质。[32]
染色体交叉 在真核生物中,是同源染色体之间遗传物质的交换。它可能发生在遗传重组的最后阶段之一,该阶段发生在减数分裂 I 期的前期(偶线期)。减数分裂期间同源染色体的配对(联会)在联会复合体形成之前开始,并且直到前期 I 结束附近才完成。交叉通常发生在匹配染色体上匹配区域断裂然后重新连接到另一条染色体时。交叉在理论上由托马斯·亨特·摩根描述。他依赖于比利时鲁汶大学的弗朗斯·阿尔方斯·扬森斯教授的发现,后者在 1909 年描述了这种现象,并将其称为“交叉型”。交叉与染色体交叉相关联,如果不是相同的话。摩根立即意识到扬森斯对交叉的细胞学解释对于他关于果蝇遗传的研究的实验结果非常重要。交叉的物质基础首先由哈里特·克里顿和芭芭拉·麦克林托克在 1931 年证明。
减数分裂重组 可以由 Spo11 蛋白引入 DNA 的双链断裂启动。此外,减数分裂重组可以作为对自发双链断裂的反应而诱导,这些断裂可能是由来自先前合成轮次的活性氧物种引起的。[33]然后,一个或多个核酸外切酶消化由双链断裂产生的 5' 末端,以产生 3' 单链 DNA 尾巴(见本节最下面的图)。减数分裂特异性重组酶 Dmc1 和通用重组酶 Rad51 包被单链 DNA 以形成核蛋白丝。重组酶催化来自断裂一端的单链 DNA 侵入对侧染色单体。接下来,侵入 DNA 的 3' 端引发 DNA 合成,导致互补链的置换。
交叉重组体 是通过一个过程产生的,在这个过程中,置换的互补链随后与从初始双链断裂的另一端产生的单链 DNA 退火(见图上的 DHJ 途径)。由此产生的结构是跨链交换,也称为霍利迪连接。即将进行交叉的两个染色单体之间的接触被称为交叉。霍利迪连接是一个四面体结构,可以通过其他重组酶“拉动”,使其沿着四链结构移动(见图中的双霍利迪连接或 DHJ)。
基因转换 可能是由于双链断裂的修复造成的。基因转换涉及遗传序列信息从“供体”序列到高度同源的“受体”染色体的单向转移。基因转换通常通过合成依赖性链退火 (SDSA) 发生 [34][35][36] 本节最下面的图中说明了这一点。在这个 SDSA DNA 修复模型中,来自双链断裂末端的游离 DNA 链侵入同源染色体,通过复制沿着“供体”染色体 DNA 互补链上的序列延伸自身。然后,延伸的链从供体染色体中退回,并在双链断裂另一端的区域(需要大约 25 到 50 个碱基对的同源性)与受体染色体上的互补序列配对。[34] 这使得通过复制完成双链断裂的修复成为可能,以从延伸链上的信息(从供体染色体复制而来)完成受体染色体上的双链结构。哺乳动物中基因转换轨迹的通常长度在 200 到 1,000 个碱基对之间。[37]
在减数分裂过程中,基因转换最常与外部区域的非交叉相关联(例如,图中所示的 SDSA 途径)。不太频繁地,减数分裂过程中的基因转换与外部区域的交叉相关联,这些事件通常由 DHJ 途径产生。在许多生物体中,减数分裂过程中没有交叉的基因转换比交叉重组更频繁地发生,通常以大约 2:1 的比例。[38] 在有丝分裂过程中,基因转换几乎是同源重组修复双链断裂的唯一模式。[36]
基因转换的研究有助于我们理解减数分裂重组的适应性功能。由于在大多数研究物种中,基因转换更频繁地是非交叉类型的,[38] 专门关注产生新的遗传变异的适应性益处的减数分裂重组的适应性功能的解释似乎不足以解释减数分裂过程中大多数重组事件。然而,大多数减数分裂重组事件可以通过这样的提议来解释,即它们是修复将传递给配子的 DNA 中损伤的一种适应性。[39][40]
遗传重组是由称为重组酶的酶催化的。RecA 是在埃希氏大肠杆菌中发现的主要重组酶,它负责修复 DNA 双链断裂 (DSB)。在酵母和其他真核生物中,修复 DSB 需要两种重组酶。RAD51 蛋白参与有丝分裂和减数分裂重组,而 DMC1 蛋白专门用于减数分裂重组。
非同源重组 重组修复很少发生在没有或几乎没有序列同源性的 DNA 序列之间。这被称为非同源重组。
- ↑ DNA 复制
- ↑ 冈崎令治,冈崎恒子,坂部匡哉,杉本久夫。DNA 复制机制,DNA 链生长的可能不连续性。一位名叫 Shandel 的美国科学家在冈崎之前发现了这种机制,但他从未因这项发现而获得认可,因为他的研究团队负责人认为这项发现是对测试结果的错误解释。Jpn J Med Sci Biol. 1967 年 6 月;20(3):255-60.
- ↑ Ogawa T, Okazaki T, 不连续 DNA 复制。Annu. Rev. Biochem. 49:421-457, 1980
- ↑ McCarthy D, Minner C, Bernstein H, Bernstein C (1976). “野生型噬菌体 T4 和 DNA 延迟琥珀突变体的 DNA 延伸速率和生长点分布”。J Mol Biol. 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
- ↑ Drake JW (1970) 突变的分子基础。 Holden-Day,旧金山 ISBN 0816224501 ISBN 978-0816224500
- ↑ 约翰·凯恩斯致霍勒斯·F·贾德森,见《创造的第八天:生物学革命的缔造者》(1979)。触摸石书籍,ISBN 0-671-22540-5。第二版:冷泉港实验室出版社,1996年平装本:ISBN 0-87969-478-5.
- ↑ WATSON JD, CRICK FH (1953). “DNA 的结构”。冷泉港实验室. 18: 123–31. PMID 13168976.
{{cite journal}}: Text "Cold Spring Harb. Symp. Quant. Biol." ignored (help) - ↑ Bloch DP (1955). “脱氧核糖核酸螺旋结构复制的可能机制”. 美国国家科学院院刊. 41 (12): 1058–64. PMC 528197. PMID 16589796.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Delbrück M (1954). “关于脱氧核糖核酸(DNA)的复制”. 美国国家科学院院刊. 40 (9): 783–8. PMC 534166. PMID 16589559.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ Delbrück,Max;Stent,Gunther S. (1957)。“关于 DNA 复制的机制”。在 McElroy,William D.;Glass,Bentley(编辑)。遗传化学基础研讨会. 约翰·霍普金斯大学出版社. pp. 699–736.
- ↑ a b Meselson,M. 和 Stahl,F.W. (1958)。“大肠杆菌中 DNA 的复制”. PNAS. 44: 671–82. doi:10.1073/pnas.44.7.671. PMID 16590258.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Meselson M,Stahl FW。DNA 复制半保留模式的证明。在“噬菌体和分子生物学起源”(编辑 Cairns J,Stent GS,Watson JD)中,冷泉港定量生物学实验室第一版(1966)第 246-251 页。ASIN:B00C2G89LM
- ↑ 原核生物 DNA 复制
- ↑ 解旋酶
- ↑ “RNA 解旋酶” 由 Eckhard Jankowsky 编辑,RSC 出版社 2010 年
- ↑ Trends Biochem Sci. 2010 年 8 月 31 日。 正在工作的 RNA 解旋酶:结合和重排. Jankowsky E. RNA 分子生物学中心和生物化学系,凯斯西储大学医学院,10900 Euclid Ave.,克利夫兰,俄亥俄州 44106,美国
- ↑ Yang 等人,DEAD-box 蛋白通过局部链分离解开双链体,Mol. Cell 28 (2007),pp. 253–263
- ↑ . Liu 等人,ATP 水解是 DEAD-box 蛋白循环所必需的,但对于双链体解开不是必需的,Proc. Natl. Acad. Sci. U. S. A. 105 (2008),pp. 20209–20214
- ↑ Jarmoskaite,I. 和 Russell,R.,(2010) DEAD-box 蛋白作为 RNA 解旋酶和伴侣蛋白。WIREs:RNA,印刷中。
- ↑ Wang JC (1991). “DNA 拓扑异构酶:为什么这么多?”. J. Biol. Chem. 266 (11): 6659–62. PMID 1849888.
{{cite journal}}: Unknown parameter|month=ignored (help) - ↑ 真核生物 DNA 复制
- ↑ http://en.wikipedia.org/w/index.php?title=Eukaryotic_DNA_replication&oldid=423827289
- ↑ 线粒体 DNA
- ↑ Davidson EH,Britten RJ (1973) 动物基因组的组织、转录和调控。Quart. Rev. Biol. 48: 565-613。
- ↑ Britten RJ,Graham DE,Neufeld BR (1974)。“通过重缔合分析重复 DNA 序列”。Meth. Enzymol. 29 (0): 363–418. doi:10.1016/0076-6879(74)29033-5. PMID 4850571.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Science 161: 529-540。
- ↑ DNA 修复
- ↑ 测序的百分比是基于常染色质部分的比例,因为人类基因组计划的目标是确定基因组的常染色质部分。端粒、着丝粒和其他异染色质区域尚未确定,一些不可克隆的缺口也未被确定。有关人类基因组计划的更多信息,请访问 http://www.ncbi.nlm.nih.gov/genome/seq/。
- ↑ Miller, Kenneth R. (2000). "9-3". 生物学 (第五版). 新泽西州上鞍河:普伦蒂斯·霍尔。第 194-5 页。 ISBN 0-13-436265-9.
{{cite book}}:|access-date=requires|url=(help) - ↑ 欧洲 11 号染色体网络
- ↑ ORNL.gov,探索基因和遗传疾病
- ↑ 遗传重组
- ↑ Carofiglio F, Inagaki A, de Vries S, Wassenaar E, Schoenmakers S, Vermeulen C, van Cappellen WA, Sleddens-Linkels E, Grootegoed JA, Te Riele HP, de Massy B, Baarends WM. (2013). SPO11 独立的 DNA 修复灶及其在减数分裂沉默中的作用。PLoS Genet. 9(6):e1003538. doi: 10.1371/journal.pgen.1003538. PMID 23754961
- ↑ a b Allers T, Lichten M. (2001). 减数分裂过程中非交叉和交叉重组的不同时间和控制。细胞 106(1):47-57。 PMID 11461701
- ↑ McMahill MS, Sham CW, Bishop DK. (2007). 减数分裂中的合成依赖性链退火。PLoS Biol. 5(11):e299. PMID 17988174 PMCID: PMC2062477。
- ↑ a b Andersen SL, Sekelsky J. (2010). 减数分裂与有丝分裂重组:双链断裂修复的两种不同途径:减数分裂与有丝分裂 DSB 修复的不同功能反映在不同的途径使用和不同的结果上。生物论文。32(12);1058-66. doi: 10.1002/bies.201000087。综述。 PMID 20967781
- ↑ Chen JM, Cooper DN, Chuzhanova N, Férec C, Patrinos GP. (2007) 基因转换:机制、进化和人类疾病。Nat Rev Genet 8(10);762-75。综述。 PMID 17846636
- ↑ a b Whitehouse, HLK. 遗传重组。(参见表 38) 纽约:威利;1982。 ISBN 0471102059 ISBN 978-0471102052
- ↑ Hörandl E. (2013). 减数分裂与自然界性别的悖论,减数分裂,Carol Bernstein 博士 (编),ISBN 978-953-51-1197-9,InTech,DOI:10.5772/56542。可在以下网址获取:http://www.intechopen.com/books/meiosis/meiosis-and-the-paradox-of-sex-in-nature
- ↑ Bernstein H, BernsteinC 和 Michod RE. (2011). 减数分裂作为 DNA 修复的进化适应。DNA 修复中的第 19 章。Inna Kruman 编辑。InTech Open 出版社。DOI:10.5772/25117 http://www.intechopen.com/books/dna-repair/meiosis-as-an-evolutionary-adaptation-for-dna-repair

