分子生物学导论/RNA 转录及其修饰
真核细胞中mRNA的“生命周期”。RNA 在细胞核中转录;经过处理,它被运输到细胞质中,并被核糖体翻译。在生命结束时,mRNA 被降解。
在转录中,基因的密码子被 RNA 聚合酶复制到信使 RNA 中。简单来说,就是从 DNA 形成 RNA 的过程被称为转录。然后,这种 RNA 拷贝被核糖体解码,核糖体通过将信使 RNA 与携带氨基酸的转移 RNA 配对来读取 RNA 序列。由于有 4 种碱基以 3 个字母的组合存在,因此有 64 种可能的密码子(43 种组合)。这些密码子编码 20 种标准氨基酸,使大多数氨基酸具有不止一种可能的密码子。还有三个“终止”或“无义”密码子表示编码区的结束;它们是 TAA、TGA 和 TAG 密码子。

转录可以简单地用 4 或 5 个步骤解释,每个步骤像波浪一样沿着 DNA 传播。
DNA 解旋/"解开",因为氢键断裂。
RNA 的游离核苷酸与互补的 DNA 碱基配对。
RNA 糖-磷酸骨架形成。(在 RNA 聚合酶的帮助下。)
解开的 RNA+DNA "梯子" 的氢键断裂,释放新的 RNA。
如果细胞有细胞核,RNA 将被进一步处理,然后通过小的核孔进入细胞质。转录过程的主要步骤是
- 前起始复合物的形成
- 转录起始
- 启动子清除
- RNA 的延伸
- 终止
在真核生物中,RNA 聚合酶,以及因此的转录起始,需要 DNA 中存在一个核心启动子序列。启动子是促进转录的 DNA 区域,在真核生物中,它们位于转录起始位点上游 -30、-75 和 -90 个碱基对处。核心启动子是启动子内的序列,对于转录起始至关重要。RNA 聚合酶能够在各种特定转录因子的存在下与核心启动子结合。
真核生物中最常见的核心启动子类型是称为TATA 盒的短 DNA 序列,位于转录起始位点上游 25-30 个碱基对处。TATA 盒作为核心启动子,是称为TATA 结合蛋白 (TBP) 的转录因子的结合位点,而 TBP 本身是另一个称为转录因子 II D (TFIID) 的转录因子的亚基。在 TFIID 通过 TBP 与 TATA 盒结合后,另外五个转录因子和 RNA 聚合酶在一系列阶段结合在 TATA 盒周围,形成前起始复合物。一个转录因子,DNA 解旋酶,具有解旋酶活性,因此参与分离双链 DNA 的相反链,以便访问单链 DNA 模板。然而,只有低或基础的转录率由前起始复合物单独驱动。其他被称为激活剂和阻遏物的蛋白质,以及任何相关的辅激活剂或辅阻遏物,负责调节转录率。
因此,前起始复合物包含
- 核心启动子序列,
- 转录因子,
- DNA 解旋酶,
- RNA 聚合酶,
- 激活剂和阻遏物。
本质上,古菌中的转录前起始与真核生物的转录前起始同源,但复杂程度低得多。[1] 古菌前起始复合物在 TATA 盒结合位点组装;然而,在古菌中,该复合物仅由 RNA 聚合酶 II、TBP 和 TFB(真核转录因子 II B (TFIIB) 的古菌同源物)组成。[2][3]通常,PIC 由六种通用转录因子组成:TFIIA (GTF2A1, GTF2A2)、TFIIB (GTF2B)、B-TFIID (BTAF1, TBP)、TFIID (BTAF1, BTF3, BTF3L4, EDF1, TAF1-15, 共 16 个)、TFIIE、TFIIF 和 TFIIH。此外,在组装过程中的某个时刻,它会与 RNA 聚合酶 II 和全酶的其余组分结合。
- TATA 结合蛋白 (TBP,TFIID 的亚基)、TBPL1 或 TBPL2 可以结合启动子或 TATA 盒。大多数基因缺乏 TATA 盒,而是使用起始元件 (INR) 或下游核心启动子。然而,TBP 始终参与其中,并且被迫在没有序列特异性的情况下结合。当 TATA 盒不存在时,来自 TFIID 的 TAF 也可能参与其中。TFIID TAF 将序列特异性结合,并迫使 TBP 非序列特异性结合,将 TFIID 的其余部分带到启动子。
- TFIIA 与 TFIID 的 TBP 亚基相互作用,并帮助 TBP 结合包含 TATA 盒的启动子 DNA。虽然 TFIIA 本身不识别 DNA,但它与 TBP 的相互作用使它能够稳定并促进 PIC 的形成。
- TFIIB 的 N 端结构域将 DNA 带到适当的位置,以便进入 RNA 聚合酶 II 的活性位点。TFIIB 部分序列特异性结合,对 BRE 有一些偏好。随后,TFIID-TFIIA-TFIIB (DAB)-启动子复合物募集 RNA 聚合酶 II 和 TFIIF。
- TFIIF(两个亚基,RAP30 和 RAP74,与细菌 σ 因子显示出一些相似性)和 Pol II 一起进入复合物。TFIIF 有助于加速聚合过程。
- TFIIE 加入正在生长的复合物,并募集 TFIIH。TFIIE 可能参与启动子处的 DNA 熔化:它包含一个锌带基序,可以结合单链 DNA。TFIIE 有助于打开和关闭 Pol II 的“钳口”状结构,这使得它能够沿着 DNA 链移动。
- DNA 可能会绕前起始复合物包裹一圈,而 TFIIF 有助于保持这种紧密包裹。在此过程中,DNA 上的扭转应力可能有助于启动子处的 DNA 熔化,形成转录气泡。
- TFIIH 和 TFIIJ 一起进入复合物。TFIIH 是一种大型蛋白质复合物,其中包含 CDK7/cyclin H 激酶复合物和 DNA 解旋酶等。TFIIH 具有三种功能:它特异性地结合模板链以确保转录正确的 DNA 链,并通过其解旋酶活性熔化或解开 DNA(依赖于 ATP),以分离两条链。它具有激酶活性,可以在氨基酸丝氨酸处磷酸化 Pol II 的 C 末端结构域 (CTD)。这将 RNA 聚合酶切换为开始产生 RNA。最后,它对于受损 DNA 的核苷酸切除修复 (NER) 至关重要。TFIIH 和 TFIIE 强烈相互作用。TFIIE 影响 TFIIH 的催化活性。如果没有 TFIIE,TFIIH 不会解开启动子。
- TFIIH 帮助形成转录泡,如果 DNA 模板没有被变性或处于超螺旋状态,则可能需要 TFIIH 进行转录。
- 然后,介质蛋白会包裹所有转录因子和 Pol II。它与增强子相互作用,增强子是位于很远位置(上游或下游)的区域,有助于调节转录。
转录前起始复合物 (PIC) 的形成类似于细菌起始中观察到的机制。在细菌中,σ 因子识别并结合到启动子序列。在真核生物中,转录因子执行此功能。
TBP 是真核转录因子 TFIID 的一个亚基。TFIID 是在 RNA 聚合酶 II (RNA Pol II) 的转录前起始复合物形成过程中第一个与 DNA 结合的蛋白。TFIID 与基因启动子区域的 TATA 盒结合,启动了其他因子募集,这些因子需要 RNA Pol II 开始转录。一些被募集的转录因子包括 TFIIA、TFIIB 和 TFIIF。这些转录因子中的每一个都是由许多蛋白亚基相互作用形成的,表明转录是一个受到严格调控的过程。TBP 也是 RNA 聚合酶 I 和 RNA 聚合酶 III 所必需的组成部分,据认为,它是所有三种 RNA 聚合酶所必需的唯一共同亚基。[4]
TATA 结合蛋白 (TBP) 是一种转录因子,特异性地结合到称为 TATA 盒的 DNA 序列。此 DNA 序列位于一些真核基因启动子的转录起始位点上游约 35 个碱基对处。TBP 以及各种与 TBP 相关的因子,构成了 TFIID,TFIID 是一种通用转录因子,它反过来构成了 RNA 聚合酶 II 转录前起始复合物的一部分。作为转录前起始复合物中少数几个以序列特异性方式结合 DNA 的蛋白之一,它有助于将 RNA 聚合酶 II 定位到基因的转录起始位点。然而,据估计,只有 10-20% 的人类启动子具有 TATA 盒。因此,TBP 可能不是定位 RNA 聚合酶 II 的唯一蛋白。
转录因子 II D (TFIID) 是组成 RNA 聚合酶 II 转录前起始复合物的几种通用转录因子之一。在转录开始之前,由 TFIID、TBP 和至少九种其他多肽组成的转录因子 II D (TFIID) 复合物,会与基因核心启动子中的 TATA 盒结合。TFIID 本身是由几个称为 TBP 相关因子 (TAF,有 16 种) 和 TATA 结合蛋白 (TBP) 的亚基组成。在试管中,只有 TBP 对包含 TATA 盒的启动子进行转录是必需的。然而,TAF 会增加启动子选择性,尤其是在没有 TATA 盒序列供 TBP 结合的情况下。TAF 包含在两个不同的复合物中,即 TFIID 和 B-TFIID。TFIID 复合物由 TBP 和八种以上的 TAF 组成。但是,大部分 TBP 存在于 B-TFIID 复合物中,B-TFIID 复合物由 TBP 和 TAFII170 (BTAF1) 以 1:1 的比例组成。
RNAP 是由 Sam Weiss、Audrey Stevens 和 Jerard Hurwitz 在 1960 年独立发现的。[5]
RNA 聚合酶 (RNAP) 是一种产生 RNA 的酶。在细胞中,RNAP 需要从 DNA 基因中构建 RNA 链作为模板,这个过程称为转录。RNA 聚合酶是生命所必需的,存在于所有生物体和许多病毒中。从化学角度来看,RNAP 是一种核苷酸转移酶,它在 RNA 转录本的 3' 端聚合核糖核苷酸。

真核生物有几种类型的 RNAP,以它们合成的 RNA 类型为特征:以下 RNA 聚合酶在真核细胞中非常常见。
1 RNA 聚合酶 I
2 RNA 聚合酶 II
3 RNA 聚合酶 III
4 RNA 聚合酶 IV
5 RNA 聚合酶 V
RNA 聚合酶 I 合成一个前rRNA 45S,它会成熟为 28S、18S 和 5.8S rRNA,这些 rRNA 将构成核糖体的主要 RNA 部分。[6]Pol I 由 8-14 个蛋白亚基 (多肽) 组成。所有 12 个亚基在 PolII 和 Pol III 中都有相同或相关的对应物。rDNA 转录仅限于核仁,核仁中存在数百个 rRNA 基因副本,这些副本排列成串联的头部到尾部重复序列。
Pol I 转录一个大的转录本,反复编码一个 rDNA 基因。该基因编码真核生物核糖体的 18S、5.8S 和 28S RNA 分子。转录本由 snoRNA 裂解。5S 核糖体 RNA 由 Pol III 转录。由于 Pol I 转录的简单性,它是作用最快的聚合酶。当 rRNA 合成受到刺激时,SL1 (选择性因子 1) 将结合到先前沉默的 rDNA 基因的启动子,并募集一个前起始复合物,Pol I 将结合到该复合物并开始 rRNA 转录。rRNA 转录的变化也可以通过转录速率的变化来实现。虽然 Pol I 通过哪种确切的机制提高其转录速率尚不清楚,但有证据表明,rRNA 合成可以在不改变活跃转录的 rDNA 数量的情况下增加或减少。

RNA 聚合酶 II 合成 mRNA 的前体,以及大多数 snRNA 和 microRNA。[7] 这是研究最多的类型,由于转录需要高度控制,因此需要一系列 转录因子 才能使其结合到启动子。RNA 聚合酶 II 是一个 550 kDa 的复合体,包含 12 个亚基。真核生物|真核生物核心 RNA 聚合酶 II 首先使用转录测定法纯化。[8] 纯化的酶通常有 10-12 个亚基(人类和酵母中为 12 个),并且不能识别特定启动子。[9] 许多亚基间的相互作用是已知的。[10] 计算机生成的 POLR2A 基因染色亚基图像:绿色 - RPB1 域 1,蓝色 - RPB1 域 2,沙色 - RPB1 域 3,浅蓝色 - RPB1 域 4,棕色 - RPB1 域 6,以及 洋红色 - RPB1 CTD。DNA 导向的 RNA 聚合酶 II 亚基 RPB1 是一种酶,在人类中由 POLR2A 基因编码。RPB1 是 RNA 聚合酶 II 的最大亚基。它包含一个 C 末端|羧基末端结构域 (CTD),由多达 52 个七肽重复序列 (YSPTSPS) 组成,这些重复序列对于聚合酶活性至关重要。[11] 与其他几个聚合酶亚基结合,它形成了聚合酶的 DNA 结合域,一个 DNA 模板 转录 成 RNA 的凹槽。[12] 它与 RPB8 发生强烈的相互作用。[10] RPB2 (POLR2B) 是第二大亚基,它与至少另外两个聚合酶亚基结合,在聚合酶内形成一个结构,该结构在酶的活性位点维持 DNA 模板和新合成的 RNA 之间的接触。[13] 第三大的亚基 RPB3 (POLR2C) 以与 POLR2J 形成异二聚体的形式存在,形成一个核心亚组装体。RPB3 与 RPB1-5、7、10-12 发生强烈的相互作用。[10] RNA 聚合酶 II 亚基 B4 (RPB4) 由 POLR2D 基因 编码[14],是第四大的亚基,可能具有压力保护作用。在人类中,RPB5 由 POLR2E 基因编码。每个 RNA 聚合酶 II 中存在两个该亚基的分子。[15] RPB5 与 RPB1、RPB3 和 RPB6 发生强烈的相互作用。[10] RPB6 (POLR2F) 与至少另外两个亚基形成一个结构,该结构稳定了 DNA 模板上的转录聚合酶。[16]POLR2G 编码 RPB7,它可能在调节聚合酶功能中发挥作用。[17] RPB7 与 RPB1 和 RPB5 发生强烈的相互作用。[10]RPB8 (POLR2H) 与亚基 RPB1-3、5 和 7 相互作用。[10] DNA 模板被转录成 RNA 的凹槽由 RPB9 (POLR2I) 和 RPB1 组成。RPB10 是基因 POLR2L 的产物。它与 RPB1-3 和 5 相互作用,并且与 RPB3 发生强烈的相互作用。[10] RPB11 亚基本身由人类中的三个亚基组成:POLR2J (RPB11-a)、POLR2J2 (RPB11-b) 和 POLR2J3[18] (RPB11-c)。与 RPB3 相互作用的还有 RPB12 (POLR2K)。[10]
RNA Pol II 的 C 末端结构域 (CTD)
[edit | edit source]
RNAPII 可以存在于两种形式:RNAPII0,具有高度磷酸化的 CTD,以及 RNAPIIA,具有非磷酸化的 CTD。RNA 聚合酶 II 的羧基末端结构域 (CTD) 是聚合酶中参与转录起始、加帽 RNA 转录本 以及与 剪接体 相连以进行 RNA 剪接 的部分。[19] CTD 通常由多达 52 个重复序列 Tyr-Ser-Pro-Thr-Ser-Pro-Ser 组成。[20] 羧基末端重复结构域 (CTD) 对于生命至关重要。仅包含没有重复序列或最多三分之一重复序列的 RNAPII 的细胞是不可存活的。[21]
CTD 是附加到 RNA 聚合酶 II 最大亚基 RPB1 的 C 末端的一个延伸。它作为许多核因子的灵活结合 支架,由 CTD 重复序列上的 磷酸化 模式决定。每个重复序列都包含一个进化上保守且重复的七肽,Tyr1-Ser2-Pro3-Thr4-Ser5-Pro6-Ser7,它在每个转录周期中都经过可逆的磷酸化。[22] 该结构域本质上是非结构化的,但进化上是保守的,并且在 真核生物 中,它包含 25 到 52 个串联的共识重复七肽。[21] 由于 CTD 通常不需要 通用转录因子 (GTF) 介导的起始和 RNA 合成,因此它不构成 RNAPII 的催化本质的一部分,而是执行其他功能。[22][23]
磷酸化主要发生在 CTD 重复序列的 Ser2 和 Ser5 上,尽管这些位置并不等效。磷酸化状态随着 RNAPII 在转录周期中前进而发生变化:起始 RNAPII 为 IIA 形式,延伸酶为 II0 形式。虽然 RNAPII0 确实包含具有超磷酸化的 CTD 的 RNAP,但由于 Ser2 与 Ser5 残基的差异磷酸化和/或 CTD 沿长度的重复序列的差异磷酸化,单个 CTD 上的磷酸化模式可能会有所不同。PCTD(RNAPII0 的磷酸化 CTD)通过将加工因子与延伸的 RNAPII 相连,将前 mRNA 加工与转录物理连接起来,例如,5'-端加帽、3'-端切割和多腺苷酸化。靠近基因 5' 端的 Ser5 磷酸化 (Ser5PO4) 主要取决于 TFIIH 的激酶活性(酵母中的 Kin28;后生动物中的 CDK7)。转录因子 TFIIH 是一种激酶,它会过度磷酸化 RNAP 的 CTD,从而导致 RNAP 复合体从起始位点移开。在 TFIIH 激酶作用之后,Ser2 残基由酵母中的 CTDK-I(后生动物中的 CDK9 激酶)磷酸化。Ctk1 (CDK9) 补充丝氨酸 5 的磷酸化,因此在中后期延伸中观察到。CDK8 和环蛋白 C (CCNC) 是 RNA 聚合酶 II 全酶的组成部分,它们磷酸化羧基末端结构域 (CTD)。CDK8 通过靶向通用转录起始因子 IIH (TFIIH) 的 CDK7/环蛋白 H 亚基来调节转录,从而在介体和基本转录机制之间建立联系。CTDP1 基因编码一种磷酸酶,它与转录起始因子 TFIIF 的羧基末端发生相互作用,TFIIF 是一种调节 RNA 聚合酶 II 延伸和起始的转录因子[24]。
RNA 聚合酶 III
[edit | edit source]RNA 聚合酶 III 合成 tRNA、rRNA 5S 以及其他存在于 细胞核 和 胞质溶胶 中的小 RNA。[25] RNA 聚合酶 III(也称为 Pol III)转录 DNA 以合成核糖体 5S rRNA、tRNA 和其他小 RNA。由 RNA Pol III 转录的基因属于“管家”基因类别,这些基因的表达在所有细胞类型和大多数环境条件下都是必需的。因此,Pol III 转录的调控主要与细胞生长和细胞周期的调控相关,因此需要的调控蛋白比 RNA 聚合酶 II 少。在转录过程中(由任何聚合酶进行),有三个主要阶段:起始;需要在基因的启动子上构建 RNA 聚合酶复合体。延伸;写出 RNA 转录本。终止;完成 RNA 写入并拆卸 RNA 聚合酶复合体。
RNA 聚合酶 IV 在植物中合成 siRNA。[26] 聚合酶 IV 专门针对植物基因组,是合成超过 90% 的所有 siRNA 所必需的。RNA 聚合酶在 siRNA 途径中沉默转座子和重复 DNA。siRNA 在通过 RNA 指导的 DNA 甲基化防御基因组免受入侵病毒和转座因子的攻击中起着重要作用。聚合酶 IV 和 ROS1 脱甲基酶解锁并重新浓缩 5S rDNA 染色质,它存在于种子中,并用于植物中成年特征的发育。聚合酶 IV 参与在植物成熟过程中设置 5S 基因中的甲基化模式。在拟南芥中,聚合酶 IV 与结合蛋白 DCL3 和 RNA 聚合酶 II RDR2 在一个沉默途径中协同工作,其中聚合酶 IV 会产生 RNA,该 RNA 被 RDR2 改变为 dsRNA,然后被 DCL3 转换为 siRNA。
在 线粒体 和 叶绿体 中还有其他类型的 RNA 聚合酶。还有参与 RNA 干扰 的 RNA 依赖性 RNA 聚合酶。[28]

真核生物的转录起始更加复杂。真核生物的 RNA 聚合酶不会直接识别核心启动子序列。相反,一组称为转录因子的蛋白质介导 RNA 聚合酶的结合和转录的起始。只有在某些转录因子附着到启动子后,RNA 聚合酶才会与之结合。转录因子和 RNA 聚合酶的完整组装体与启动子结合,形成转录起始复合体。古细菌域中的转录类似于真核生物中的转录。[29]
在细菌中,转录始于 RNA 聚合酶与 DNA 中启动子的结合。RNA 聚合酶是一个核心酶,由五个亚基组成:2 个 α 亚基、1 个 β 亚基、1 个 β' 亚基和 1 个 ω 亚基。在起始开始时,核心酶与一个σ因子相关联,该σ因子有助于找到启动子序列下游合适的 -35 和 -10 个碱基对。
什么是 σ因子?
σ因子(σ因子)是一种原核生物转录起始因子,它使 RNA 聚合酶能够特异性地与基因启动子结合。不同的σ因子在响应不同的环境条件时被激活。每个 RNA 聚合酶分子都包含一个σ因子亚基,在模式细菌大肠杆菌中,它是下面列出的那些之一。大肠杆菌有七个σ因子;σ因子的数量在细菌物种之间有所不同。σ因子以其特征分子量区分。例如,σ70 指的是分子量为 70 kDa 的σ因子。
转录因子对于基因表达的调节至关重要,因此存在于所有生物体中。生物体中发现的转录因子的数量随着基因组大小的增加而增加,更大的基因组往往每个基因拥有更多的转录因子。人类基因组中大约有 2600 种蛋白质含有 DNA 结合域,其中大多数被认为充当转录因子。因此,基因组中大约 10% 的基因编码转录因子,这使得该家族成为人类蛋白质中最大的单一家族。此外,基因经常被多个用于不同转录因子的结合位点包围,并且这些基因中每一个的有效表达都需要几个不同转录因子的协同作用(例如,参见肝细胞核因子)。因此,大约 2000 种人类转录因子子集的组合使用可以轻松解释人类基因组中每个基因在发育过程中的独特调节。
在分子生物学中,转录因子(有时称为序列特异性 DNA 结合因子)是一种与特定 DNA 序列结合的蛋白质,从而控制遗传信息从 DNA 到 mRNA 的移动(或转录)。转录因子通过促进(作为激活剂)或阻止(作为抑制剂) RNA 聚合酶(执行从 DNA 到 RNA 的遗传信息转录的酶)到特定基因的募集,单独或与其他蛋白质在复合体中执行此功能。
通用转录因子或 GTF 紧密参与基因调控过程,大多数是生命所必需的。TATA 结合蛋白 (TBP) 是一种 GTF,它与 TATAA 盒(T=胸腺嘧啶,A=腺嘌呤)结合,该盒是所有基因编码区域直接上游的核酸基序。TBP 负责招募 RNA Pol II 全酶,这是转录起始的最后一步。这些蛋白质是普遍存在的,并与 DNA 的核心启动子区域相互作用,该区域包含所有 II 类基因的转录起始位点。并非所有 GTF 都在转录起始中发挥作用;有些是转录的第二步(延伸)所必需的。例如,FACT 复合体 (Spt16/Pob3 in S. cerevisiae, SUPT16H/SSRP1 in humans) 的成员促进 RNA Pol II 在基因编码区域的快速移动。这是通过将组蛋白八聚体移出活跃聚合酶的路径并因此解压缩染色质来实现的。
转录因子在结构上是模块化的,包含以下 域
DNA 结合域 (DBD),它附着到 DNA 增强子或启动子的特定序列:所有载体的必要组成部分:用于驱动载体转基因的转录 启动子 序列)与受调控基因相邻。与转录因子结合的 DNA 序列通常被称为 反应元件。
反式激活域 (TAD),它包含其他蛋白质的结合位点,例如 [转录共调节因子。这些结合位点通常被称为 激活功能 (AFs)。[30]
一个可选的 信号感知域 (SSD)(例如,配体结合域),它感知外部信号,并响应这些信号,将这些信号传递到转录复合体的其余部分,导致基因表达的向上或向下调节。此外,DBD 和信号感知域可能存在于转录复合体中关联的单独蛋白质上以调节基因表达。

第一个键合成之后,RNA聚合酶必须从启动子区域脱落。在此期间,RNA转录本容易释放并产生截短的转录本。这种现象称为中止起始,在真核生物和原核生物中都很常见。中止起始持续发生,直到σ因子重新排列,形成转录延伸复合物(形成一个移动的35个碱基对的足迹)。在合成80个核苷酸的mRNA之前,σ因子就会释放。一旦转录本达到大约23个核苷酸,它就不会再滑动,延伸过程就可以开始了。这与大多数转录过程一样,是一个依赖能量的过程,消耗三磷酸腺苷(ATP)。DNA的其中一条链,模板链(或非编码链),被用作RNA合成的模板。随着转录的进行,RNA聚合酶沿着模板链移动,并利用与DNA模板的碱基配对互补性来创建RNA拷贝。虽然RNA聚合酶以3'→5'方向沿着模板链移动,但编码链(非模板链)和新形成的RNA也可以用作参考点,因此转录可以被描述为发生在5'→3'方向。这会产生一个从5'→3'方向的RNA分子,它是编码链的精确复制品(除了胸腺嘧啶被尿嘧啶取代,并且核苷酸由一个核糖(5碳)糖组成,而DNA在其糖磷酸骨架中含有脱氧核糖(少了一个氧原子))。与DNA复制不同,mRNA转录可以涉及单个DNA模板上的多个RNA聚合酶以及多次转录循环(特定mRNA的扩增),因此可以从单个基因拷贝快速产生许多mRNA分子。延伸过程还涉及一个校对机制,可以替换错误掺入的碱基。在真核生物中,这可能对应于转录过程中短暂的暂停,使合适的RNA编辑因子能够结合。这些暂停可能是RNA聚合酶本身固有的,也可能是染色质结构造成的。[31]
转录终止
[edit | edit source]
细菌使用两种不同的策略来终止转录。在ρ非依赖性转录终止中,当新合成的RNA分子形成一个富含G-C的茎环结构,后面跟着一段U的序列时,RNA转录就会停止。当茎环结构形成时,机械应力会破坏弱的rU-dA键,现在填充了DNA-RNA杂合体。这会将多聚U转录本从RNA聚合酶的活性位点中拉出来,从而有效地终止转录。在“ρ依赖性”类型的终止中,一个叫做“ρ”的蛋白质因子会破坏模板和mRNA之间的相互作用,从而将新合成的mRNA从延伸复合物中释放出来。真核生物中的转录终止机制尚未完全了解,但涉及到新转录本的切割,随后在其新的3'端添加模板无关的A,这是一个叫做polyadenylation的过程。
ρ依赖性终止
[edit | edit source]ρ因子作用于RNA底物。ρ的关键功能是其解旋酶活性,其能量由RNA依赖性的ATP水解提供。ρ的初始结合位点是在正在合成的RNA中,位于实际终止序列上游的一个延伸的(约70个核苷酸,有时为80-100个核苷酸)单链区域,该区域富含胞嘧啶,缺乏鸟嘌呤,被称为rho利用位点或rut。已经发现了多个ρ结合序列。这些序列之间没有发现一致性,但不同的序列似乎都是特异性的,因为序列中的微小突变会破坏其功能。ρ结合到RNA上,然后利用其ATP酶活性提供能量沿着RNA移动,直到到达RNA-DNA螺旋区域,在那里它会解开杂合双链结构。RNA聚合酶在终止序列处暂停,这是由于在距离ρ结合位点约100nt处有一个特定位点,被称为ρ敏感暂停位点。因此,即使RNA聚合酶的速度比ρ快约40nt/秒,但这不会对ρ终止机制造成问题,因为RNA聚合酶允许ρ因子赶上。
ρ非依赖性终止
[edit | edit source]ρ非依赖性终止(也称为内在终止)是真核生物和原核生物中的一种机制,导致mRNA转录停止。在这种机制中,mRNA包含一个可以与自身碱基配对形成一个长度为7-20个碱基对的茎环结构的序列,该结构也富含胞嘧啶-鸟嘌呤碱基对。这些碱基彼此之间形成三个氢键,因此特别牢固。在茎环结构之后是一段尿嘧啶残基链。尿嘧啶和腺嘌呤之间的键非常弱。一个与RNA聚合酶结合的蛋白质(nusA)会牢固地结合到茎环结构上,导致聚合酶暂时停滞。这种聚合酶的暂停与多聚尿嘧啶序列的转录相一致。弱的腺嘌呤-尿嘧啶键使RNA-DNA双链体不稳定,导致其解开并从RNA聚合酶上解离。不以多聚尿嘧啶序列结尾的茎环结构会使RNA聚合酶暂停,但它通常会在短暂的时间后继续转录,因为双链体太稳定,无法解开到足以导致终止的程度。ρ非依赖性转录终止是构成顺式作用RNA调节元件(如核糖开关)[32]活性的一种常见机制。
mRNA及其修饰
[edit | edit source]pre-mRNA分子经历三种主要的修饰。这些修饰是在RNA被翻译之前发生在细胞核中的5'端加帽、3'端polyadenylation和RNA剪接。
mRNA的加帽
[edit | edit source]
5'端帽看起来像RNA分子的3'端(帽子的核糖的5'碳原子是连接的,而3'端是未连接的)。这提供了对5'端外切核酸酶的显着抵抗力。pre-mRNA的加帽涉及在5'端添加7-甲基鸟苷(m7G)。为了实现这一点,需要去除末端的5'磷酸,这在磷酸酶的帮助下完成。然后,鸟苷转移酶催化反应,产生二磷酸5'端。二磷酸5'端然后攻击GTP分子中的α磷原子,以便在5'5'三磷酸键中添加鸟嘌呤残基。酶(鸟嘌呤-N7-)甲基转移酶(“帽MTase”)从S-腺苷甲硫氨酸转移一个甲基到鸟嘌呤环上。这种类型的帽子,只有(m7G)在位置上,被称为帽0结构。相邻核苷酸的核糖也可以被甲基化,得到帽1。RNA分子下游核苷酸的甲基化产生帽2、帽3结构等等。在这些情况下,甲基被添加到核糖糖的2'OH基团上。帽子保护了初级RNA转录本的5'端免受对3'5'磷酸二酯键有特异性的核糖核酸酶的攻击。[33]
起点是未改变的RNA分子的5'端。它具有一个末端核苷酸,后面跟着连接到5'碳原子的三个磷酸基团。
一个末端磷酸基团被去除(通过RNA末端磷酸酶),留下两个末端磷酸基团。
GTP被添加到末端磷酸基团上(通过鸟苷转移酶),在此过程中失去了两个磷酸基团(来自GTP)。这导致了5'到5'三磷酸键的形成。
鸟嘌呤的7-氮被甲基化(通过甲基转移酶)。
其他甲基转移酶可选择性地用于进行5'端邻近核苷酸的甲基化。
5'端帽具有4个主要功能
调节核输出。
防止外切核酸酶降解。
促进翻译(参见核糖体和翻译)。
促进5'端邻近内含子的切除。
RNA 的核输出受帽结合复合物 (CBC) 的调节,CBC 专门结合帽化的 RNA。然后,CBC 被核孔复合物识别并输出。在翻译的先锋轮次后到达细胞质后,CBC 被翻译因子 eIF-4E 和 eIF-4G 取代。然后,该复合物被其他翻译起始机制(包括核糖体)识别。帽以两种方式防止 5' 降解。首先,通过在功能上看起来像 3' 末端来防止 5' 外切核酸酶对 mRNA 的降解(如上所述)。其次,CBC 复合物和 eIF-4E/eIF-4G 阻止了脱帽酶对帽的访问。这增加了 mRNA 的半衰期,这在真核生物中至关重要,因为输出过程需要大量时间。mRNA 的脱帽由至少由 Dcp1 和 Dcp2 组成的脱帽复合物催化,该复合物必须与 eIF-4E 竞争才能结合帽。因此,5' 帽是活跃翻译 mRNA 的标志,细胞利用它来调节 mRNA 半衰期,以响应新的刺激。不需要的 mRNA 被发送到 P 体中进行暂时储存或脱帽,其细节仍在解决中。5' 邻近内含子切除促进的机制尚不清楚,但 5' 帽似乎环绕并与剪接体在剪接过程中相互作用,从而促进内含子切除。
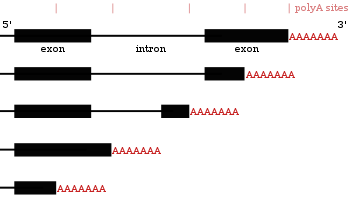
RNA 分子 3' 末端的 pre-mRNA 加工涉及其 3' 末端的裂解,然后添加约 200 个腺嘌呤残基以形成一个 poly(A) 尾巴。如果在 pre-mRNA 分子的 3' 末端附近存在一个 聚腺苷酸化信号序列 (5'- AAUAAA-3'),则发生裂解和腺苷酸化反应,该序列紧随另一个序列,通常为 (5'-CA-3')。第二个信号是裂解位点。一个 富含 GU 的序列 通常也存在于 pre-mRNA 分子的下游。在合成序列元件后,两种称为 蛋白质 的多亚基 裂解和聚腺苷酸化特异性因子 (CPSF) 和 裂解刺激因子 (CStF) 从 RNA 聚合酶 II 转移到 RNA 分子。这两个因子结合到序列元件。形成一个包含其他裂解因子和酶 聚腺苷酸聚合酶 (PAP) 的蛋白质复合物。该复合物在聚腺苷酸化序列和由 (5'-CA-3') 序列标记的裂解位点处的富含 GU 的序列之间裂解 RNA。然后,poly(A) 聚合酶使用 ATP 作为前体,向 RNA 分子的新 3' 末端添加约 200 个腺嘌呤单元。随着 poly(A) 尾巴的合成,它结合了 poly(A) 结合蛋白的多个拷贝,这保护了 3' 末端免受核糖核酸酶的消化。[34]
聚(A) 结合蛋白或“PABP”
聚(A) 结合蛋白(或“PABP”)是一种 RNA 结合蛋白,它结合到 mRNA 的 poly(A) 尾巴。poly(A) 尾巴位于 mRNA 的 3' 末端。核同种型选择性地结合到大约 50 个核苷酸并刺激聚腺苷酸聚合酶的活性。哺乳动物聚(A) 结合蛋白的表达在翻译水平上受反馈机制调节:编码 PABP 的 mRNA 在其 5' UTR 中含有与聚(A) 结合蛋白结合的富含 A 的序列。这导致翻译的抑制。真核生物聚(A) 结合蛋白的胞质同种型通过其 C 末端结构域与起始因子 eIF-4G 结合。EIF-4G 与 eIF-4E 结合,另一个起始因子结合到 mRNA 5' 末端的 5' 帽。这种结合形成了真核蛋白合成的特征性环状结构。胞质溶胶中的聚(A) 结合蛋白相互作用蛋白竞争 eIF-4G 结合位点。聚(A) 结合蛋白还被证明与终止因子 (eRF3) 相互作用。
可变聚腺苷酸化
许多蛋白质编码基因具有不止一个聚腺苷酸化位点,因此一个基因可以编码几种在 3' 末端不同的 mRNA。由于可变聚腺苷酸化改变了 3' 非翻译区的长度,因此它可以改变 3' 非翻译区包含哪些 microRNA 结合位点。microRNA 往往抑制翻译并促进与其结合的 mRNA 的降解,尽管有一些例子表明 microRNA 会稳定转录本。可变聚腺苷酸化还可以缩短编码区,从而使 mRNA 编码不同的蛋白质,但这远不如仅仅缩短 3' 非翻译区常见。聚(A) 位点的选择取决于参与聚腺苷酸化的蛋白质的表达。例如,裂解刺激因子 (CStF) 的一个亚基 CstF-64 的表达在巨噬细胞中响应脂多糖(触发免疫反应的一组细菌化合物)而增加。这导致选择弱聚(A) 位点,从而产生更短的转录本。这去除了防御相关产物(如溶菌酶和 TNF-α)的 mRNA 3' 非翻译区中的调节元件。然后,这些 mRNA 具有更长的半衰期并产生更多这些蛋白质。除了聚腺苷酸化机制中的那些之外,其他 RNA 结合蛋白也可以影响聚腺苷酸化位点的使用,就像靠近聚腺苷酸化信号的 DNA 甲基化一样。[35]。

剪接是将 pre-mRNA 修饰以去除称为内含子的某些非编码序列片段的过程;保留的片段包括蛋白质编码序列,称为外显子。有时 pre-mRNA 信息可以以几种不同的方式剪接,允许单个基因编码多种蛋白质。这个过程称为可变剪接。剪接通常由称为剪接体的 RNA-蛋白质复合物执行,但一些 RNA 分子也能够催化它们自身的剪接。[36]

内含子 对观察到的内含子分布的一个合理的假设是,现代真核生物的古代祖先含有大量的内含子,并且控制快速生长的物种中基因组大小的选择压力可能导致许多古代内含子的消除。使这个问题复杂化的是,发现许多内含子本身是可移动的遗传元件,可以插入基因并从基因中删除。在发现内含子后不久,研究人员提出了相互竞争的理论,这些理论提供了对剪接体内含子的起源和早期进化的替代方案。其他类型的内含子,如自剪接内含子和 tRNA 内含子,没有受到太多争议,但请参阅前者。这些通常被称为“内含子先于” (IE) 和“内含子后于” (IL) 观点。由沃尔特·吉尔伯特倡导的 IE 模型假设内含子非常古老,并且在原核生物和真核生物的最早理论祖先(原生物)中大量存在。在这个模型中,内含子随后从原核生物中丢失,使它们能够获得生长效率。该理论的一个核心预测是,早期的内含子是促进代表蛋白质结构域的外显子重组的媒介。该模型无法解释在相关基因之间观察到的某些内含子位置变异。
IL 模型假设内含子是在真核生物和原核生物分化后,最近插入到最初不含内含子的连续基因中。在这个模型中,内含子可能起源于转座因子。该模型基于观察到的剪接体内含子仅限于真核生物。然而,关于原核生物-真核生物祖先中内含子的存在以及随后的真核生物进化过程中内含子的丢失-获得存在相当大的争议。内含子及其内含子-外显子结构的进化可能在很大程度上独立于编码序列的进化。
内含子最初是在腺病毒的蛋白质编码基因中发现的,但现在已知它们存在于所有生物界中各种各样的基因中。不同基因组中内含子的频率在生物有机体的整个范围内差异很大。例如,内含子在高等脊椎动物(例如人类和小鼠)的核基因组中极为常见,其中蛋白质编码基因几乎总是包含多个内含子,而内含子在一些真核微生物(例如面包酵母(酿酒酵母))的核基因中很少见。相反,脊椎动物的线粒体基因组完全没有内含子,而真核微生物的线粒体基因组可能包含许多内含子。

每个剪接体由五个称为snRNP的小核RNA蛋白(发音为“snurps”)和一系列非snRNP相关蛋白因子组成。构成核剪接体的snRNP分别命名为U1、U2、U4、U5和U6,并参与多个RNA-RNA和RNA-蛋白相互作用。snRNP的RNA成分富含尿嘧啶(尿嘧啶核苷酸的核苷类似物)。剪接体的典型组装在每个hnRNA上重新进行。hnRNA包含特定的序列元件,这些元件在剪接体组装过程中被识别和利用。这些元件包括5'端剪接位点、分支点序列、多嘧啶区和3'端剪接位点。剪接体催化内含子的去除以及侧翼外显子的连接。内含子通常在其5'端剪接位点具有GU核苷酸序列,在其3'端剪接位点具有AG。3'剪接位点可以通过称为多嘧啶区(PPT)的可变长度多嘧啶进一步定义,多嘧啶区具有招募因子到3'剪接位点和可能招募因子到分支点序列(BPS)的双重功能。BPS包含第一个剪接步骤所需的保守腺苷。一组含量较少的snRNP,U11、U12、U4atac和U6atac,连同U5,是所谓的次级剪接体的亚基,该剪接体剪接一类罕见的pre-mRNA内含子,称为U12型。这些snRNP形成U12剪接体,位于细胞质中。从II类内含子的第一个晶体结构中获得的新证据表明,剪接体实际上是一种核酶,它使用双金属离子机制进行催化。
剪接体活性位点形成模型涉及在hnRNA底物上以有序的、逐步的方式组装离散的snRNP颗粒。hnRNA的首次识别涉及U1 snRNP与hnRNA的5'端剪接位点以及其他非snRNP相关因子结合,形成承诺复合物或哺乳动物中的早期(E)复合物。[38][39] 承诺复合物是与hnRNA结合在剪接途径中的ATP无关的复合物。[40] U2 snRNP通过与E复合物成分U2AF(U2 snRNP辅助因子)以及可能与U1 snRNP相互作用而被招募到分支区域。在一个ATP依赖性反应中,U2 snRNP与分支点序列(BPS)紧密结合,形成复合物A。在U2 snRNP和hnRNA分支区域之间形成的双链结构将分支腺苷突出,将其指定为第一个转酯化的亲核试剂。[41]
U2 snRNA中存在假尿嘧啶残基,几乎与分支位点相对,导致U2 snRNP结合后RNA-RNA双链结构的构象发生改变。具体来说,由假尿嘧啶诱导的双链结构的改变将突出腺苷的2'OH置于有利位置,以便进行第一步剪接。[42] U4/U5/U6三聚体snRNP被招募到组装的剪接体中,形成复合物B,并在经过几轮重排后,复合物C(剪接体)被激活以进行催化。[43][44] 尚不清楚三聚体snRNP是如何被招募到复合物A中的,但这个过程可能是通过蛋白-蛋白相互作用和/或U2 snRNA与U6 snRNA之间的碱基配对相互作用来介导的。
U5 snRNP通过U5 snRNA的不变环[45]与5'和3'剪接位点的序列相互作用,而U5蛋白成分与3'剪接位点区域相互作用。[46]
在招募三聚体snRNP后,在第一步催化步骤之前发生了几轮RNA-RNA重排,并且在催化活性剪接体中发生了进一步的重排。许多RNA-RNA相互作用是相互排斥的;然而,尚不清楚是什么触发了这些相互作用,以及这些重排的顺序。第一次重排可能是U1 snRNP从5'剪接位点上移位以及形成U6 snRNA相互作用。众所周知,U1 snRNP只与完全形成的剪接体[47]弱结合,并且U1 snRNP对在包含短5'外显子和5'剪接位点的模型底物寡核苷酸上形成U6-5'剪接位点相互作用具有抑制作用。[48] U2 snRNP与分支点序列(BPS)的结合是RNA-RNA相互作用置换蛋白-RNA相互作用的一个例子。在招募U2 snRNP后,承诺复合物中的分支结合蛋白SF1被置换,因为U2 snRNA和SF1的结合位点是相互排斥的事件。
在U2 snRNA中,存在其他相互排斥的重排,发生在竞争性构象之间。例如,在活性形式中,茎环IIa是被偏爱的;在非活性形式中,环与下游序列之间的相互排斥相互作用占主导地位。[44] 尚不清楚U4是如何从U6 snRNAm上移位的,尽管RNA参与了剪接体的组装,并且可能发挥了解开U4/U6并促进形成U2/U6 snRNA相互作用的作用。U4/U6茎环I和II的相互作用分离,U6的释放的茎环II区域自身折叠形成一个分子内茎环,U4不再需要参与进一步的剪接体组装。释放的U6茎环I区域与U2 snRNA配对,形成U2/U6螺旋I。然而,螺旋I结构与U2 snRNA的内部5'茎环区域的3'半部分是相互排斥的。
snRNP的RNA成分与内含子相互作用,并可能参与催化。已经鉴定出两种类型的剪接体(主要剪接体和次级剪接体),它们包含不同的snRNP。主要剪接体剪接包含5'剪接位点处的GU和3'剪接位点处的AG的内含子。它由U1、U2、U4、U5和U6 snRNP组成,并在细胞核中活跃。此外,还需要一些蛋白质,包括U2AF和SF1,以组装剪接体。
E复合物-U1与5'剪接位点的GU序列结合,以及辅助蛋白/酶ASF/SF2、U2AF(在Py-AG位点结合)、SF1/BBP(BBP=分支结合蛋白);
A复合物-U2与分支位点结合,并水解ATP;
B1复合物-U5/U4/U6三聚体结合,U5在5'位点与外显子结合,U6与U2结合;
B2复合物-U1被释放,U5从外显子转移到内含子,U6在5'剪接位点结合;
C1复合物-U4被释放,U6/U2催化转酯化,使内含子的5'端与内含子上的A连接并形成套索结构,U5与3'剪接位点处的外显子结合,5'位点被切割,导致套索结构的形成;
C2复合物-U2/U5/U6与套索结构保持结合,3'位点被切割,外显子使用ATP水解连接起来。剪接的RNA被释放,套索结构被解开。
这种类型的剪接被称为规范剪接或套索途径,它占剪接的99%以上。相反,当内含子侧翼序列不遵循GU-AG规则时,就会发生非规范剪接(见下文“次级剪接体”)。**次级剪接体**
次级剪接体是一种核糖核蛋白复合物,它催化从植物、昆虫、脊椎动物和一些真菌(米根霉)的真核信使RNA中去除(剪接)一类非典型剪接体内含子(U12型)。这个过程被称为非规范剪接,与U2依赖的规范剪接相反。U12型内含子在人类细胞中占所有内含子的不到1%。然而,它们存在于执行基本细胞功能的基因中。次级剪接体与主要剪接体非常相似,然而,它剪接出具有不同剪接位点序列的罕见内含子。虽然次级剪接体和主要剪接体包含相同的U5 snRNP,但次级剪接体具有不同的但功能类似的snRNP,分别用于U1、U2、U4和U6,它们分别被称为U11、U12、U4atac和U6atac。与主要剪接体一样,它只存在于细胞核中[49]。跨剪接跨剪接是一种剪接形式,它连接两个不在同一RNA转录本中的外显子
自我剪接
[edit | edit source]自我剪接发生在形成核酶的罕见内含子中,通过RNA独自执行剪接体的功能。存在三种类型的自我剪接内含子,I类、II类和III类。I类和II类内含子执行类似于剪接体的剪接,不需要任何蛋白质。这种相似性表明I类和II类内含子可能与剪接体存在进化关系。自我剪接也可能非常古老,并且可能存在于蛋白质出现之前的RNA世界中。两个转酯化反应的特点是I类内含子剪接的机制:游离鸟苷核苷(或位于内含子中)或核苷酸辅因子(GMP、GDP、GTP)的3'OH攻击5'剪接位点处的磷酸盐。5'外显子的3'OH成为亲核试剂,第二个转酯化反应导致两个外显子的连接。II类内含子剪接的机制(与I类内含子类似的两个转酯化反应)如下:内含子中特定腺苷的2'OH攻击5'剪接位点,从而形成套索结构。5'外显子的3'OH触发了3'剪接位点的第二个转酯化反应,从而将外显子连接在一起。
第一类内含子 第一类内含子分布于细菌、低等真核生物和高等植物中。然而,它们在细菌中的出现似乎比在低等真核生物中更零散,并且在高等植物中变得普遍。第一类内含子中断的基因存在显著差异:它们中断细菌基因组中的 rRNA、mRNA 和 tRNA 基因,以及低等真核生物的线粒体和叶绿体基因组,但在低等真核生物的核基因组中只入侵 rRNA 基因。在高等植物中,这些内含子似乎仅限于叶绿体和线粒体的少数 tRNA 和 mRNA 基因。内含子早期理论和内含子晚期理论都在解释第一类内含子的起源方面找到了证据。一些第一类内含子编码同源内切酶 (HEG),它催化内含子的移动。有人提出,HEG 将内含子从一个位置移动到另一个位置,从一个生物体移动到另一个生物体,从而解释了自私的第一类内含子的广泛传播。到目前为止,除了从前体中剪接自身以防止宿主死亡外,尚未确定第一类内含子的任何生物学作用。少数第一类内含子也被发现编码一类称为成熟酶的蛋白质,这些蛋白质促进内含子剪接。
第一类内含子的剪接通过两个连续的酯转移反应进行。外源鸟苷或鸟苷核苷酸 (exoG) 首先停靠在位于 P7 的活性 G 结合位点,其 3'-OH 与位于 P1 的 5' 剪接位点的磷酸二酯键对齐,导致上游外显子产生一个游离的 3'-OH 基团,并且 exoG 连接到内含子的 5' 端。然后,内含子的末端 G (omega G) 交换 exoG 并占据 G 结合位点,以组织第二酯转移反应,P1 中上游外显子的 3'-OH 基团与 P10 中的 3' 剪接位点对齐,导致相邻的上游和下游外显子的连接和催化内含子的释放。人们提出蛋白质聚合酶和磷酸酶中看到的双金属离子机制被第一类和第二类内含子用来处理磷酰基转移反应,这在最近解析的 Azoarcus 第一类内含子高分辨率结构中得到了明确证明。
第二类催化内含子
第二类催化内含子存在于真菌、植物和原生生物的细胞器的 rRNA、tRNA 和 mRNA 中,也存在于细菌的 mRNA 中。它们是大型自剪接核酶,具有 6 个结构域(通常标记为 dI 到 dVI)。此模型和比对仅代表 V 和 VI 结构域。第二类内含子的一个子集还在内含子 ORF 中编码必需的剪接蛋白。因此,这些内含子的长度可以长达 3kb。剪接以与核前 mRNA 剪接几乎相同的方式进行,包含两个转酯化步骤。VI 结构域中凸起腺苷的 2' 羟基攻击 5' 剪接位点,然后上游外显子的 3' OH 对 3' 剪接位点进行亲核攻击。蛋白质机器需要体内剪接,长距离内含子-内含子和内含子-外显子相互作用对于剪接位点定位很重要。第二类内含子进一步细分为 IIA 和 IIB 组,它们在剪接位点共有序列和 VI 结构域中凸起腺苷(形成套索的潜在分支点)与 3' 剪接位点的距离方面存在差异。
第三类内含子
Montandon,P. 和 Stutz,E. (1984) 以及 Hallick,R.B. 等人。(1988 年和 1989 年) 报告了在眼虫叶绿体中发现的一种新型内含子的例子。1989 年,David A.Christopher 和 Richard B.Hallick 提出标题,第三类内含子,以识别这种新类别,其具有以下特征:第三类内含子比其他自剪接内含子类别短得多,在 Christopher 和 Hallick 已知的内含子中,长度在 95 到 110 个核苷酸之间,并且在叶绿体中被发现。另一方面,Christopher 和 Hallick 声明:“相比之下,最小的眼虫叶绿体第二类内含子……是 277 个核苷酸。”它们靠近剪接位点的保守序列与第二类内含子的序列相似,但保守位置更少。它们没有映射到第二类内含子的保守二级结构中。(事实上,Christopher 和 Hallick 无法在第三类内含子中识别任何保守的二级结构元件。)它们通常与参与翻译和转录的基因相关联。它们非常富含 A+T。1994 年,发现了长度为其一个数量级的第三类内含子,这表明长度本身并不是第三类内含子剪接的决定因素(Copertino DW., Hall ET.)。第三类内含子的剪接通过套索和环状 RNA 的形成进行。第三类内含子和核内含子之间的相似性包括保守的 5' 边界序列、套索形成、缺乏内部结构以及使用替代剪接边界的 ability。

动物中观察到的 RNA 编辑系统可能是从单核苷酸脱氨酶进化而来的,这些酶导致了包括 apobec-1 和 adar 基因在内的更大的基因家族。这些基因与参与核苷酸代谢的细菌脱氨酶具有高度相似性。大肠杆菌的腺苷脱氨酶不能脱氨 RNA 中的核苷;该酶的反应袋太小,无法让 RNA 链结合。然而,在相应的 human analog 基因 APOBEC-1 和 ADAR 中,氨基酸变化使该活性位点变宽,从而允许脱氨。在锥虫线粒体中观察到的插入编辑与核苷转化过程无关。其他研究表明,参与其中的酶是从不同来源招募和改造的。但是,通过 gRNA 和 mRNA 之间的相互作用而进行的核苷酸插入的特异性类似于动物和棘阿米巴线粒体中的 tRNA 编辑过程。此外,真核 rRNA 的指导 RNA 分子介导的核糖甲基化可能提供了 RNA 编辑和修饰之间的另一个联系。因此,大量研究表明,RNA 编辑可能在物种形成的特定谱系中进化,因为它们的机制存在细微的差异。数据不支持 RNA 编辑在 RNA 世界中存在,因为其机制与当时可能存在的任何假设过程无关。因此,RNA 编辑似乎是在后期进化而来的,以弥补基因序列的变化并增加变异性。
在来自布氏锥虫线粒体的动力体中发现了通过添加和删除尿嘧啶进行 RNA 编辑。RNA 编辑始于未编辑的初级转录物与引导 RNA (gRNA) 的碱基配对,引导 RNA 包含与插入/删除点周围区域互补的序列。新形成的双链区域然后被包封在编辑体中,编辑体是一个大型多蛋白复合物,催化编辑。编辑体在引导 RNA 和未编辑转录物之间的第一个错配核苷酸处打开转录物并开始插入尿苷。插入的尿苷将与引导 RNA 碱基配对,只要引导 RNA 中存在 A 或 G,插入就会继续,并在遇到 C 或 U 时停止。插入的核苷酸会导致移码,并导致翻译的蛋白质与其基因不同。
编辑体复合物 编辑体机制涉及在引导 RNA 和未编辑转录物之间错配点的内切核酸酶切割。下一步由复合物中的一个酶催化,即末端 U 转移酶,它从 UTP 在 mRNA 的 3' 端添加 Us。打开的末端被复合物中的其他蛋白质固定到位。另一种酶,U 特异性外切核酸酶,去除未配对的 Us。在编辑使 mRNA 与 gRNA 互补后,RNA 连接酶重新连接编辑的 mRNA 转录物的末端。因此,编辑体只能沿初级 RNA 转录物以 3' 到 5' 的方向进行编辑。该复合物一次只能作用于一个引导 RNA。因此,需要广泛编辑的 RNA 转录物将需要多个引导 RNA 和编辑体复合物。
通过脱氨进行编辑
C-U 编辑 编辑涉及胞嘧啶脱氨酶,它将胞嘧啶碱基脱氨成尿嘧啶碱基。C 到 U 编辑的一个例子是人类的载脂蛋白 B 基因。Apo B100 在肝脏中表达,而 Apo B48 在肠道中表达。B100 形式具有 CAA 序列,该序列在肠道中被编辑为 UAA,这是一个终止密码子。它在肝脏中没有被编辑。
A-I 编辑 A 到 I 编辑发生在双链 RNA (dsRNA) 的区域。作用于 RNA 的腺苷脱氨酶 (ADAR) 是参与腺苷到肌苷的亲水性脱氨 (A 到 I 编辑) 的 RNA 编辑酶。A 到 I 编辑可以是特异性的(在 dsRNA 的片段内编辑单个腺苷)或混杂的(最多 50% 的腺苷被编辑)。特异性编辑发生在短双链体中(例如,在 mRNA 中形成的,其中内含子序列与互补的外显子序列配对),而混杂编辑发生在更长的双链体区域中(例如,前 miRNA 或初级 miRNA、由转基因或病毒表达产生的双链体、由配对的重复元件产生的双链体)。A 到 I 编辑有很多影响,源于 I 在翻译和形成二级结构时都表现得像 G。这些影响包括编码能力的改变、改变的 miRNA 或 siRNA 靶标群体、异染色质形成、核隔离、细胞质隔离、Tudor-SN 的内切核酸酶切割、miRNA 和 siRNA 加工的抑制以及改变的剪接[50]。
- ↑ Littlefield, O., Korkhin, Y. 和 Sigler, P.B. (1999)。"TBP/TFB/启动子复合物定向组装的结构基础"。PNAS。96 (24): 13668–13673。doi:10.1073/pnas.96.24.13668。PMC 24122。PMID 10570130.
{{cite journal}}: CS1 maint: 多个名称:作者列表 (link) - ↑ Hausner, W; Thomm, M (2001)。"古细菌转录起始过程中的事件:开放复合物的形成和 DNA-蛋白质相互作用"。细菌学杂志。183 (10): 3025–3031。doi:10.1128/JB.183.10.3025-3031.2001。PMC 95201。PMID 11325929.
{{cite journal}}: |author= 和 |last1= 同时指定了多个参数 (help) - ↑ Qureshi, SA; Bell, SD; Jackson, SP (1997)。"嗜热球菌属 Sulfolobus shibatae 转录所需的因子"。EMBO 杂志。16 (10): 2927–2936。doi:10.1093/emboj/16.10.2927。PMC 1169900。PMID 9184236.
{{cite journal}}: |author= 和 |last1= 同时指定了多个参数 (help) - ↑ TATA 结合蛋白
- ↑ Jerard Hurwitz (2005)。"RNA 聚合酶的发现"。生物化学杂志。280 (52): 42477–85。doi:10.1074/jbc.X500006200。PMID 16230341.
{{cite journal}}: 未知参数 |month= 被忽略 (help) - ↑ Grummt I. (1999)。"RNA 聚合酶 I 调节哺乳动物核糖体基因转录"。核酸研究进展分子生物学。62: 109–54。doi:10.1016/S0079-6603(08)60506-1。PMID 9932453.
- ↑ Lee Y (2004)。"microRNA 基因由 RNA 聚合酶 II 转录"。EMBO J。23 (20): 4051–60。doi:10.1038/sj.emboj.7600385。PMID 15372072.
{{cite journal}}: 未知参数 |coauthors= 被忽略 (建议使用 |author= ) (help); 未知参数 |month= 被忽略 (help) - ↑ Sawadogo M, Sentenac A (1990)。"RNA 聚合酶 B (II) 和通用转录因子"。生物化学年评。59: 711–54。doi:10.1146/annurev.bi.59.070190.003431。PMID 2197989.
{{cite journal}}: 引用中存在空值或未知参数: |month= (help) - ↑ Myer VE, Young RA (1998). "RNA 聚合酶 II 全酶和亚复合物" (PDF). J. Biol. Chem. 273 (43): 27757–60. doi:10.1074/jbc.273.43.27757. PMID 9774381.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ a b c d e f g h Acker J, de Graaff M, Cheynel I, Khazak V, Kedinger C, Vigneron M (1997). "人 RNA 聚合酶 II 亚基之间的相互作用". J Biol Chem. 272 (27): 16815–21. doi:10.1074/jbc.272.27.16815. PMID 9201987.
{{cite journal}}: 未知参数|month=被忽略 (帮助)CS1 维护:多个名字:作者列表 (链接) - ↑ Brickey WJ, Greenleaf AL (1995). "果蝇 RNA 聚合酶 II 的羧基末端重复结构域的体内功能研究". Genetics. 140 (2): 599–613. PMC 1206638. PMID 7498740.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ "Entrez Gene:POLR2A 聚合酶(RNA)II(DNA 指导)多肽 A,220kDa".
- ↑ "Entrez Gene:POLR2B 聚合酶(RNA)II(DNA 指导)多肽 B,140kDa".
- ↑ Khazak V, Estojak J, Cho H, Majors J, Sonoda G, Testa JR, Golemis EA (1998). "新型 RNA 聚合酶 II(pol II)亚基 hsRPB4 与其伴侣 hsRPB7 以及 pol II 相互作用的分析". Mol Cell Biol. 18 (4): 1935–45. PMC 121423. PMID 9528765.
{{cite journal}}: 未知参数|month=被忽略 (帮助)CS1 维护:多个名字:作者列表 (链接) - ↑ "Entrez Gene:POLR2E 聚合酶(RNA)II(DNA 指导)多肽 E,25kDa".
- ↑ "Entrez Gene:POLR2F 聚合酶(RNA)II(DNA 指导)多肽 F".
- ↑ "Entrez Gene:POLR2G 聚合酶(RNA)II(DNA 指导)多肽 G".
- ↑ "POLR2J3 聚合酶(RNA)II(DNA 指导)多肽 J3".
- ↑ Brickey WJ, Greenleaf AL (1995). "果蝇 RNA 聚合酶 II 的羧基末端重复结构域的体内功能研究". Genetics. 140 (2): 599–613. PMC 1206638. PMID 7498740.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ Meinhart A, Cramer P (2004). "RNA 聚合酶 II 羧基末端结构域被 3'-RNA 加工因子识别" (摘要). 自然. 430 (6996): 223–6. doi:10.1038/nature02679. PMID 15241417.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ a b Corden JL (1990). "RNA 聚合酶 II 的尾巴". 生物学趋势科学. 15: 383–7. doi:10.1016/0968-0004(90)90236-5.
{{cite journal}}: 引用为空未知参数:|month=(帮助) - ↑ a b Phatnani HP, Greenleaf AL (2006). "RNA 聚合酶 II CTD 的磷酸化和功能". 基因发育. 20 (1): 2922–36. doi:10.1101/gad.1477006. PMID 17079683.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ http://en.wikipedia.org/wiki/RNA_polymerase_II_holoenzyme
- ↑ http://en.wikipedia.org/wiki/RNA_polymerase_II_holoenzyme
- ↑ Willis IM. (1993). "RNA 聚合酶 III. 基因,因子和转录特异性". 欧洲生物化学杂志. 212 (1): 1–11. doi:10.1111/j.1432-1033.1993.tb17626.x. PMID 8444147.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ Herr AJ, Jensen MB, Dalmay T, Baulcombe DC (2005). "RNA 聚合酶 IV 导向内源 DNA 的沉默". 科学. 308 (5718): 118–20. doi:10.1126/science.1106910. PMID 15692015.
{{cite journal}}: CS1 维护:多个名称:作者列表 (链接) - ↑ Wierzbicki AT, Ream TS, Haag JR, Pikaard CS (2009). "RNA 聚合酶 V 转录引导 ARGONAUTE4 到染色质". Nat. Genet. 41 (5): 630–4. doi:10.1038/ng.365. PMC 2674513. PMID 19377477.
{{cite journal}}: 未知参数|month=被忽略 (帮助)CS1 维护:多个名称:作者列表 (链接) - ↑ Makeyev EV, Bamford DH (2002). "参与转录后基因沉默的细胞 RNA 依赖性 RNA 聚合酶具有两种不同的活性模式". 分子细胞. 10 (6): 1417–27. doi:10.1016/S1097-2765(02)00780-3. PMID 12504016.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ Mohamed Ouhammouch, Robert E. Dewhurst, Winfried Hausner, Michael Thomm, and E. Peter Geiduschek (2003). "通过招募 TATA 结合蛋白来激活古细菌转录". 美国国家科学院院刊. 100 (9): 5097–5102. doi:10.1073/pnas.0837150100. PMC 154304. PMID 12692306.
{{cite journal}}: CS1 维护:多个名称:作者列表 (链接) - ↑ Wärnmark A, Treuter E, Wright AP, Gustafsson J-Å (2003). "核受体的激活功能 1 和 2:转录激活的分子策略". Mol. Endocrinol. 17 (10): 1901–9. doi:10.1210/me.2002-0384. PMID 12893880.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ 转录 (遗传学)
- ↑ http://en.wikipedia.org/w/index.php?title=Intrinsic_termination&oldid=428278551
- ↑ 5' 帽子
- ↑ Hames & Hooper 2006, p. 225
- ↑ http://en.wikipedia.org/w/index.php?title=Polyadenylation&oldid=422375377
- ↑ 信使 RNA
- ↑ 次要剪接体
- ↑ Jamison SF, Crow A, and Garcia-Blanco MA (1992 年 10 月 1 日). "哺乳动物提取物中的剪接体组装途径". 分子与细胞生物学. 12 (10): 4279–87. PMC 360351. PMID 1383687.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Seraphin B. and Rosbash M. (1989). "识别功能性 U1 snRNA 前信使 RNA 复合物,这些复合物致力于剪接体组装和剪接". 细胞. 59 (2): 349–58. doi:10.1016/0092-8674(89)90296-1. PMID 2529976.
- ↑ Legrain P, Seraphin B, Rosbash M (1988 年 9 月 1 日). "酵母前信使 RNA 早期投入剪接体途径". 分子与细胞生物学. 8 (9): 3755–60. PMC 365433. PMID 3065622.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Query, C. C., M. J. Moore, and P. Sharp (1994). "前信使 RNA 剪接中的分支亲核试剂选择:对凸起双链体模型的证据". 基因发育. 8 (5): 587–97. doi:10.1101/gad.8.5.587. PMID 7926752.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Newby M. I. and Greenbaum, N. L. (2002). "通过保守的假尿嘧啶对剪接体分支位点识别基序进行雕刻". 自然结构生物学. 9 (12): 958–65. doi:10.1038/nsb873. PMID 12426583.
- ↑ Burge, C.B.; 等 (1999). "剪接体对 mRNA 的前体进行剪接". 在 Gesteland, R.F., Cech, T.R., Atkins, J.F. (编). RNA 世界. 冷泉港实验室出版社. pp. 525–60. ISBN 0879693800.
{{cite book}}: 在:|author=中明确使用等 (help)CS1 maint: 多个名称:编辑者列表 (link) - ↑ a b Staley JP, Guthrie C (1998). "剪接体的机械装置:电机、时钟、弹簧和其他东西". 细胞. 92 (3): 315–26. doi:10.1016/S0092-8674(00)80925-3. PMID 9476892.
- ↑ Newman AJ,Teigelkamp S 和 Beggs JD (1995)。"酵母剪接体中光活化交联监测 5' 和 3' 剪接位点 snRNA 相互作用"。RNA。1 (9):968–80。 PMC 1369345。 PMID 8548661.
- ↑ Chiara MD,Palandjian L,Feld Kramer R,Reed R (1997)。"证据表明 U5 snRNP 在哺乳动物中识别催化步骤 II 的 3' 剪接位点"。EMBO J。16 (15):4746–59。 doi:10.1093/emboj/16.15.4746。 PMC 1170101。 PMID 9303319.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Moore, M. J. 和 Sharp, P. A. (1993)。"来自前 mRNA 剪接立体化学的剪接体中两个活性位点的证据"。自然。365 (6444):364–8。 doi:10.1038/365364a0。 PMID 8397340.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Konforti BB,Koziolkiewicz MJ,Konarska MM (1993)。"破坏 5' 剪接位点与 U1 snRNA 5' 端之间的碱基配对是剪接体组装所必需的"。细胞。75 (5):863–73。 doi:10.1016/0092-8674(93)90531-T。 PMID 8252623.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Will CL,Lührmann R (2005 年 8 月)。“U12 依赖性剪接体对一类罕见内含子的剪接”。生物化学。386 (8):713–24
- ↑ http://en.wikipedia.org/w/index.php?title=RNA_editing&oldid=424324287
