生命科学方法与概念/PCR

聚合酶链式反应 (PCR) 用于将一段 DNA 扩增几个数量级,产生特定 DNA 序列的数千到数百万个拷贝。
PCR 由 Kary Mullis 于 1983 年开发,现已成为医疗和生物研究实验室中常用的、且经常不可或缺的技术,应用于各种领域。1993 年,Mullis 因其在 PCR 方面的贡献与 Michael Smith 一起获得了诺贝尔化学奖。
该方法依赖于热循环,包括对反应进行重复的加热和冷却循环,以实现 DNA 熔化和 DNA 的酶促复制。包含与目标区域互补序列的引物以及耐热 DNA 聚合酶是实现选择性和重复扩增的关键成分。随着 PCR 的进行,生成的 DNA 本身被用作复制的模板,启动连锁反应,其中 DNA 模板呈指数级扩增。PCR 可以被广泛修改以执行各种遗传操作。


PCR 用于扩增 DNA 链的特定区域(DNA 目标)。大多数 PCR 方法通常扩增 0.1 到 10 千碱基对 (kbp) 的 DNA 片段,尽管一些技术允许扩增高达 40 kbp 的片段。扩增产物的数量由反应中可用的底物决定,随着反应的进行,底物变得有限。
基本的 PCR 设置需要多种成分和试剂。这些成分包括
- 包含待扩增 DNA 区域的 DNA 模板。
- 两个引物,它们与 DNA 目标的正链和反链的 3' 端互补。
- 一种耐热的 DNA 聚合酶
- 脱氧核苷三磷酸 (dNTP)
- 缓冲溶液,为 DNA 聚合酶的最佳活性及稳定性提供合适的化学环境。
PCR 通常在 热循环仪 中的小反应管(0.2-0.5 毫升体积)中,以 10-200 微升的反应体积进行。热循环仪加热和冷却反应管,以实现反应每个步骤所需的温度(见下文)。许多现代热循环仪利用珀耳帖效应,通过简单地反转电流,就可以实现对装有 PCR 管的模块进行加热和冷却。薄壁反应管具有良好的热传导性,可以快速热平衡。大多数热循环仪都有加热盖,以防止反应管顶部出现冷凝。缺少加热盖的较旧热循环仪需要在反应混合物顶部放置一层油或在管内放置一个蜡球。

PCR 通常包括一系列 20-40 次重复的温度变化,每次循环通常包括三个不同的温度步骤。循环通常在高温(>90°C)下进行单次温度步骤,并在最后进行一次保持,以进行最终产物延伸或短暂储存。使用的温度以及它们在每个循环中应用的时间长度取决于多种参数。这些参数包括用于 DNA 合成的聚合酶、反应中二价离子和 dNTP 的浓度以及引物的熔解温度 (Tm)。
- 初始化 (仅适用于需要通过热启动 PCR 进行热活化的 DNA 聚合酶):此步骤包括将反应加热到 94-96°C(或如果使用极耐热的聚合酶,则为 98°C),并保持 1-9 分钟。
- 变性:此步骤是第一个常规循环事件,包括将反应加热到 94-98°C,持续 20-30 秒。它通过破坏互补碱基之间的氢键,使 DNA 模板链分离,产生单链 DNA 分子。
- 退火:反应温度降低到 50-65°C,持续 20-40 秒,允许引物退火到单链 DNA 模板。此温度需要足够低,以允许引物与链杂交,但需要足够高,以确保杂交的特异性,即引物应仅与模板的完全互补部分结合。如果温度过低,引物可能会不完全结合。如果温度过高,引物可能不会结合。通常,退火温度比所用引物的 Tm 低约 3-5°C。只有当引物序列与模板序列非常匹配时,才会形成稳定的 DNA-DNA 氢键。聚合酶与引物-模板杂交体结合,并开始 DNA 形成。
- 延伸:此步骤的温度取决于所用的 DNA 聚合酶;Taq 聚合酶在 75-80°C 时具有最佳活性温度,并且通常在这种酶的情况下使用 72°C 的温度。在此步骤中,DNA 聚合酶通过添加与模板互补的 dNTP,以 5' 到 3' 的方向合成与 DNA 模板链互补的新 DNA 链,将 dNTP 的 5'-磷酸基团与新生 DNA 链末端的 3'-羟基结合。延伸时间既取决于所用的 DNA 聚合酶,也取决于待扩增的 DNA 片段的长度。作为经验法则,在最佳温度下,DNA 聚合酶每分钟会聚合一千个碱基。在最佳条件下,即如果由于底物或试剂限制而没有限制,那么在每个延伸步骤中,DNA 目标的数量都会加倍。延伸后,DNA 会再次变性,新合成的片段可以在下一个循环中用作模板,从而导致特定 DNA 片段的指数级扩增。
- 最终延伸:此单一步骤偶尔在 70-74°C(这是大多数用于 PCR 的聚合酶的最佳活性所需的温度)下进行,持续 5-15 分钟,以确保任何剩余的单链 DNA 完全延伸。
- 最终保持:此步骤在 4-15°C 下进行无限时间,可用于短期储存反应。
好的引物对于成功的 PCR 反应至关重要。设计引物时应考虑几个因素
- 一般来说,引物的长度应该在 18-30 个核苷酸之间。
- 尝试使引物的熔解温度 (Tm) 在 65°C 到 75°C 之间,并且彼此相差 5°C 以内。
- 目标 GC 含量在 40% 到 60% 之间。
- 在每个引物的 3' 端使用 G 或 C(“GC 夹”)以促进结合。但是,3 个或更多个 G 或 C 碱基在该端可能会稳定引物的非特异性退火。
- 引物不应自身互补,也不应与反应中的另一个引物互补,否则它们会形成二聚体。特别是引物的 3' 端至关重要。
- 引物不应形成稳定的二级结构。
- 尝试避免 4 个或更多个相同碱基的连续序列,或二核苷酸重复。
- 应将引物与模板序列进行比较,以检测二级结合位点。
- 当在引物的 5' 端添加限制位点时,添加 3 到 4 个额外的核苷酸以允许有效切割。
以下公式可以用作经验法则来估计引物的熔解温度
由于GC碱基对比AT碱基对更稳定,因此假设每个G或C使熔解温度升高4°C,而A或T仅贡献2°C。有一些工具使用更复杂的算法来计算Tm
- OligoAnalyzer 计算Tm,还可以检查二聚体和发夹结构。
- 寡核苷酸性质计算器 使用不同的算法计算Tm。
- NEB Tm 计算器 考虑了聚合酶。
- AmplifX 评估引物的多种特性。
引物可以通过手动设计或使用软件来设计,这些软件可以根据预先确定的参数为给定的模板序列推荐引物对。
- Primer3 设计引物,并让用户对引物的性质有很大控制权。
- Primer-BLAST 使用Primer3设计引物,然后使用BLAST自动分析其特异性。
这些只是众多用于设计和分析引物的工具中一些常用的例子。有些工具针对特殊应用进行了优化,例如实时PCR。以下网站提供了引物设计工具的概述
实时PCR
[edit | edit source]实时PCR或定量PCR是PCR的一种变体,用于扩增和同时检测或定量DNA分子。该程序遵循聚合酶链式反应的一般原理;其关键特征是在“实时”反应过程中检测到扩增的DNA。与标准PCR相比,这是一种新方法,在标准PCR中,反应产物是在反应结束时检测到的。定量PCR中用于检测产物的两种常用方法是
- 与任何双链DNA插入的非特异性荧光染料。
- 由标记有荧光报告基因的寡核苷酸组成的序列特异性DNA探针。
MIQE指南(定量实时PCR实验出版物的最低信息)建议使用缩写qPCR来表示定量实时PCR,而使用RT-qPCR来表示逆转录-qPCR。缩写“RT-PCR”通常表示逆转录聚合酶链式反应,而不是实时PCR,但并非所有作者都遵循此惯例。
检测方法
[edit | edit source]荧光染料
[edit | edit source]检测和定量PCR产物的最简单方法是使用DNA结合染料,如溴化乙锭或SYBR Green。这些荧光团的荧光强度在与DNA结合时会增加,这意味着PCR过程中DNA的积累与荧光强度的增加相关。测量是在每个循环的延伸结束时进行的。
这种方法的缺点是特异性低。无法区分不同的PCR产物,非特异性产物(如引物二聚体)会干扰目标序列的准确定量。
荧光探针
[edit | edit source]荧光报告探针仅检测包含探针序列的DNA;因此,使用报告探针显着提高了特异性,并且即使在非特异性DNA扩增存在的情况下也能实现定量。荧光探针可用于多重检测——在同一反应中检测多个基因——基于具有不同颜色标记的特异性探针,前提是所有靶基因的扩增效率相似。
这些方法依赖于荧光共振能量转移(FRET)来检测两个荧光团是否在空间上接近。FRET是一种在两个感光分子之间发生能量转移的机制。最初处于电子激发态的供体发色团可以通过非辐射偶极-偶极耦合将能量转移到受体发色团。这种能量转移的效率与供体和受体之间的距离的六次方成反比,使得FRET对距离的微小变化极其敏感。因此,可以利用FRET效率的测量来确定两个荧光团是否彼此之间的距离在一定范围内。
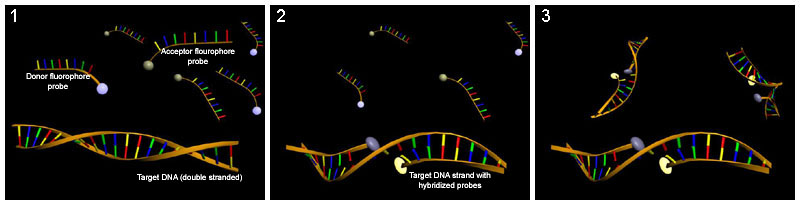
LightCycler探针
[edit | edit source]LightCycler探针由两个寡核苷酸组成,它们互补于靶序列的相邻区域。一个探针携带一个FRET供体,另一个携带一个FRET受体。当两个寡核苷酸都与靶序列退火时,就会发生FRET,并且可以利用荧光强度进行定量。
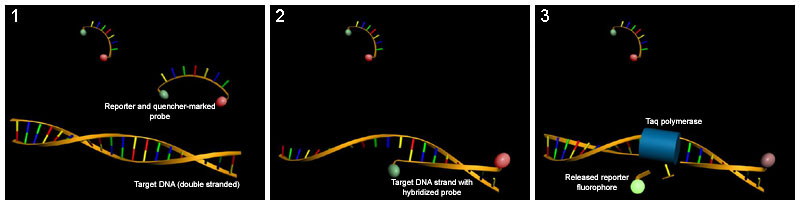
TaqMan探针
[edit | edit source]这种方法依赖于一个寡核苷酸,该寡核苷酸在一端有一个荧光报告基因,在另一端有一个荧光猝灭剂。只要探针完整,报告基因与猝灭剂的紧密接近就会阻止检测其荧光。探针在两个引物之间退火,并在延伸过程中被Taq聚合酶的5'-3'外切核酸酶活性降解。这打破了报告基因-猝灭剂的接近,从而允许未猝灭的荧光发射,这可以在用激光激发后检测到。因此,在每个PCR循环中,由报告探针靶向的产物增加会导致荧光比例增加,这是由于探针分解和报告基因释放所致。
定量
[edit | edit source]
与终点PCR(传统PCR)不同,实时PCR可以通过测量荧光来对扩增过程中的任何点进行目标产物的定量(实际上,测量的是其在给定阈值上的水平)。定量PCR中常用的DNA定量方法依赖于将荧光绘制成循环次数。用于检测基于DNA的荧光的阈值略高于背景。荧光超过阈值的循环次数称为阈值循环(Ct)或根据MIQE指南,定量循环(Cq)。
在指数扩增阶段,目标 DNA 模板(扩增子)的数量每循环翻倍。例如,Cq 比另一个样本早 3 个循环的 DNA 样本,其模板量是后者的 2^3 = 8 倍。然而,不同引物和模板之间的扩增效率往往不同。因此,引物-模板组合的效率是在 DNA 模板的系列稀释实验中评估的,以创建 Cq 随每次稀释而变化的标准曲线。然后使用线性回归的斜率来确定扩增效率,如果 1:2 的稀释导致 Cq 差异为 1,则扩增效率为 100%。循环阈值法对反应机制做了几个假设,并且依赖于扩增曲线低信噪比区域的数据,这会在数据分析过程中引入很大差异。
为了量化基因表达,将目标基因的 RNA 或 DNA 的 Cq 从同一样本中管家基因的 RNA/DNA 的 Cq 中减去,以消除不同样本之间 RNA 量和质量差异的影响。这种归一化程序通常称为ΔCt 法,允许比较不同样本中目标基因的表达。然而,要进行这种比较,需要在所有样本中,正常化参考基因的表达非常相似。因此,选择满足此标准的参考基因非常重要,而且往往具有挑战性,因为只有极少数基因在不同条件或组织中表现出相同的表达水平。虽然循环阈值分析已与许多商业软件系统集成,但在可重复性成为问题的情况下,应该考虑更准确、更可靠的扩增曲线数据分析方法。
熔解曲线
[edit | edit source]
qPCR 允许通过分析其熔解温度来鉴定特定的扩增 DNA 片段。这通常使用双链 DNA 结合染料作为报告分子(例如 SYBR Green)来完成。DNA 熔解温度对扩增片段具有特异性。该技术的实验结果通过比较分析的 DNA 样品的解离曲线获得。
与传统的 PCR 不同,这种方法避免了之前使用电泳技术来展示所有样本的结果。这是因为,尽管是一种动力学技术,但定量 PCR 通常在不同的终点进行评估。因此,该技术通常提供更快的结果和/或使用比电泳更少的试剂。如果需要后续电泳,则只需测试那些实时 PCR 显示结果存疑的样本,或者对那些对特定决定因素检测呈阳性的样本进行结果验证。
PCR 的其他变体
[edit | edit source]- 组装 PCR(也称为聚合酶循环组装或 PCA)是通过对一组具有短重叠片段的长寡核苷酸进行 PCR 来合成长的 DNA 结构,将两个或多个 DNA 片段组装成一个片段。在聚合酶循环过程中,寡核苷酸与互补片段退火,然后被聚合酶填补。因此,每个循环都随机增加了各种片段的长度,具体取决于哪些寡核苷酸相互找到。至关重要的是,所有片段之间都存在某种程度的互补性,否则将不会产生最终的完整序列。在这一初始构建阶段之后,添加了涵盖两端的额外引物以执行常规的 PCR 反应,从而将目标序列从所有较短的不完整片段中扩增出来。然后可以使用凝胶纯化来识别和分离完整的序列。这种技术可以替代基于连接的组装。
- 在菌落 PCR中,细菌菌落通过 PCR 直接筛选,无需过夜培养和质粒制备。菌落 PCR 可以用作一种高通量方法,用于检查细胞中的质粒是否包含插入片段。使用合适的引物,还可以确定插入片段的方向。使用无菌移液器吸头对菌落进行取样,将少量细胞转移到 PCR 混合物中。在最初的变性步骤中,质粒从细胞中释放出来,可以作为模板。延长变性步骤和使用裂解缓冲液可以促进细胞裂解。
- 热启动 PCR是一种手动操作的技术,在添加聚合酶之前,将反应成分加热到 DNA 熔解温度(例如 95 °C)。这样可以防止在较低温度下发生非特异性扩增。或者,特种试剂在环境温度下抑制聚合酶的活性,要么通过抗体的结合,要么通过共价结合的抑制剂的存在,这些抑制剂仅在高温活化步骤后解离。'热启动/冷终止 PCR' 是使用新的杂交聚合酶实现的,这些聚合酶在环境温度下不活跃,仅在升高的温度下被激活。
- 多重 PCR使用几对引物退火到不同的目标序列。这允许在单个样本中同时分析多个目标。例如,在检测基因突变时,可以组合六个或更多个扩增。在 DNA 指纹图谱的标准方案中,检测的目标通常以 3 或 4 组进行扩增。
- 嵌套 PCR用于提高 DNA 扩增的特异性。在两个连续的反应中使用两组引物。在第一次 PCR 中,使用一对引物产生 DNA 产物,这些产物可能包含从非目标区域扩增的产物。然后,第一次 PCR 的产物用作第二次 PCR 的模板,使用一个(半嵌套)或两个不同的引物,其结合位点位于(嵌套)第一组内,从而提高了特异性。与传统 PCR 相比,嵌套 PCR 在特异性地扩增长 DNA 产物方面往往更成功,但它需要更详细地了解目标序列。


- 重叠延伸聚合酶链反应(OE-PCR)也称为重叠延伸/重叠悬垂延伸(SOE)PCR。它用于在序列的特定位置插入特定的突变,或者将较小的 DNA 片段拼接成较大的多核苷酸。
与大多数 PCR 反应一样,每个序列使用两个引物 - 一个用于每个末端。为了拼接两个 DNA 分子,在要连接的末端使用特殊的引物。对于每个分子,在要连接的末端处的引物被构建为使得它具有与另一个分子末端互补的 5' 悬垂。在第一步中,这些延伸片段被扩增。之后,将两个 DNA 分子混合,并仅使用远端引物进行 PCR。引入的重叠互补序列将充当引物,这两个序列将被融合。这种方法相对于其他基因拼接技术具有一个优势,即不需要限制性位点。为了获得更高的产量,一些引物像不对称 PCR 中一样过量使用。
为了将突变插入 DNA 序列,设计了一个特定的引物。该引物可能包含单个替换,或者在其 5' 末端包含一个新序列。如果需要删除,则添加一个位于删除位置 5' 侧的序列,因为引物的 3' 末端必须与模板链具有互补性,以便引物能够充分退火到模板 DNA。
- cDNA 末端快速扩增(RACE)用于获得 RNA 转录本的全长序列。RACE 导致产生感兴趣的 RNA 序列的 cDNA 拷贝,该拷贝通过逆转录产生,然后通过 cDNA 拷贝的 PCR 扩增。RACE 可以提供从转录本中已知的小序列到 RNA 的 5' 端(5' RACE-PCR)或 3' 端(3' RACE-PCR)的 RNA 转录本的序列。这种技术有时被称为单侧 PCR 或锚定 PCR。
- 在降落 PCR中,退火温度在后续循环中逐渐降低。早期循环中的退火温度通常比所用引物的标准 Tm 高 3-5 °C,而在后期循环中,它比 Tm 低类似的量。初始较高的退火温度导致引物结合的特异性更高,而较低的温度允许在反应结束时更有效地扩增。
参考文献
[edit | edit source]- Bustin, S.A., 2004. 定量 PCR 的 A-Z. 电泳 1–15。
- Higuchi, R., Dollinger, G., Walsh, P.S., Griffith, R., 1992. 特定 DNA 序列的同时扩增和检测。生物技术。 (纽约)。10, 413–417. doi:10.1038/nbt0492-413
- Kubista, M., Andrade, J.M., Bengtsson, M., Forootan, A., Jonák, J., Lind, K., Sindelka, R., Sjöback, R., Sjögreen, B., Strömbom, L., Ståhlberg, A., Zoric, N., 2006. 实时聚合酶链反应。分子医学的各个方面。
- Mullis, K.B., 1990. 聚合酶链反应的非凡起源。科学美国人。
- Powledge, T.M., 2004. 聚合酶链反应。生理学教育进展。28, 44–50。
- Saiki, R.., Scharf, S., Faloona, F., Mullis, K.B., Horn, G.T., 1985. β-珠蛋白基因组序列的酶促扩增和限制性位点分析用于诊断镰状细胞贫血。科学 (80-. )。239, 1350–54. doi:10.1126/science.2999980

