下一代测序 (NGS)/预处理
FASTQ 文件通过包含序列质量信息来扩展 FASTA 文件中的序列信息。FASTQ 文件通常包含四行。
- 以 @ 开头的行,包含序列标识符
- 实际序列
- 以 + 开头的行,后跟可选序列标识符
- 用 ASCII 空间编码的质量值行
因此,第二行和第四行必须具有相同的长度。下面给出一个示例,显示一个序列“ATGTCT”...
@HWI-ST999:102:D1N6AACXX:1:1101:1235:1936 1:N:0: ATGTCTCCTGGACCCCTCTGTGCCCAAGCTCCTCATGCATCCTCCTCAGCAACTTGTCCTGTAGCTGAGGCTCACTGACTACCAGCTGCAG + 1:DAADDDF<B<AGF=FGIEHCCD9DG=1E9?D>CF@HHG??B<GEBGHCG;;CDB8==C@@>>GII@@5?A?@B>CEDCFCC:;?CCCAC
这里将 -5 到 41 范围内的质量值添加到偏移量,然后从 ASCII 表中获取结果字符。因此,整个数据可以表示为文本。虽然 Illumina 对质量格式进行了多次更改,最终几乎恢复到 Sanger 编码,但最重要的是偏移量是 33(如 Sanger 和 Illumina v1.8 及更高版本)还是 64(如以前的 Illumina(和 Solexa)格式)。从图表中可以看到,如果你发现以下任何字符:! "# $ % & ' ( ) * + , - . / 0 1 2 3 4 5 6 7 8 9 :,你的偏移量必须是 33,而以下任何字符 KLMNOPQRSTUVWXYZ[\]^_`abcdefgh 指向偏移量 64。上面的示例因此以 33 为基准偏移,因为我们发现第一个字符是 1。还要记住 @ 和 + 符号是质量的有效字符,因此即使一行以 @ 或 + 开头,这也可能只是质量字符串的开头。
请参阅下面的质量图表,该图表是根据 维基百科 文章修改的。
SSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSSS..................................................... ..........................XXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXXX...................... ...............................IIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIIII...................... .................................JJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJJ...................... LLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLLL.................................................... !"#$%&'()*+,-./0123456789:;<=>?@ABCDEFGHIJKLMNOPQRSTUVWXYZ[\]^_`abcdefghijklmnopqrstuvwxyz{|}~ | | | | | | 33 59 64 73 104 126 0........................26...31.......40 -5....0........9.............................40 0........9.............................40 3.....9.............................40 0........................26...31........41
S - Sanger and Illumina 1.8+, Offset 33, Quality range (0, 40) (!,I) X - Solexa, Offset 64, Quality range (-5, 40) (;,h) I - Illumina 1.3+ Offset 64, Quality range (0, 40) (@,h) J - Illumina 1.5+ Offset 64, Quality range (3, 40) (B,h) L - Illumina 1.8+ Offset 33, Quality range (0, 41) (!,J)
当进行质量控制时,需要考虑各种指标。
最简单的方法是显然是上面 FASTQ 文件中介绍的质量评分。因此,它已经给出了关于碱基调用质量的有效想法。由于读段的质量通常在序列过程中逐渐下降,因此通常的做法是通过对文件中所有读段的质量进行平均来确定第一个、第二个、第三个、...第 n 个碱基的平均质量。此外,为了了解一些关于分布的信息,通常会给出显示分位数的条形图。这将让我们了解数据需要进行哪种修剪和质量过滤。
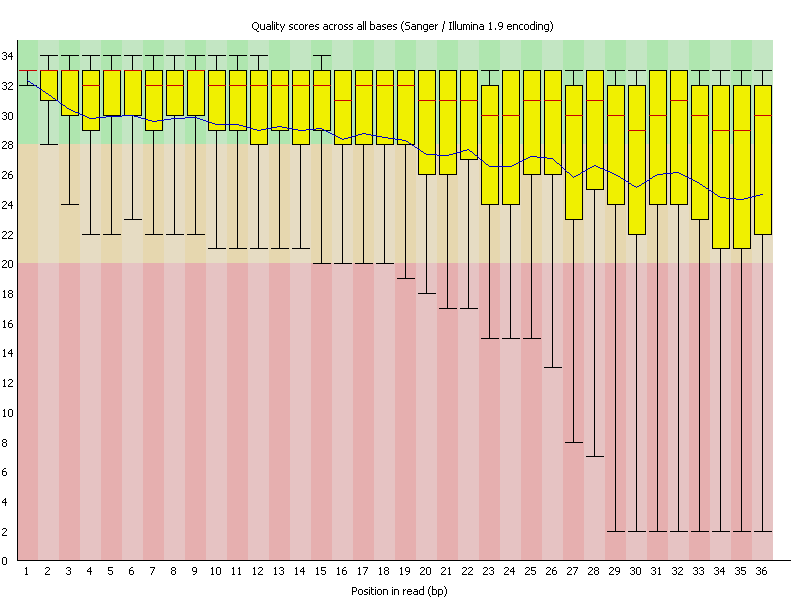
例如,这里使用 FastQC 对序列数据进行了质量调查。可以看到,序列读段长 36 个碱基,平均序列质量(用蓝色线表示)稳步下降。在许多新的 Illumina 试剂盒中,序列质量会在稳步下降之前先上升一点。
但是,除了检查每个碱基之外,还可以对每个读段的质量进行平均,并显示这些序列质量的累积图。
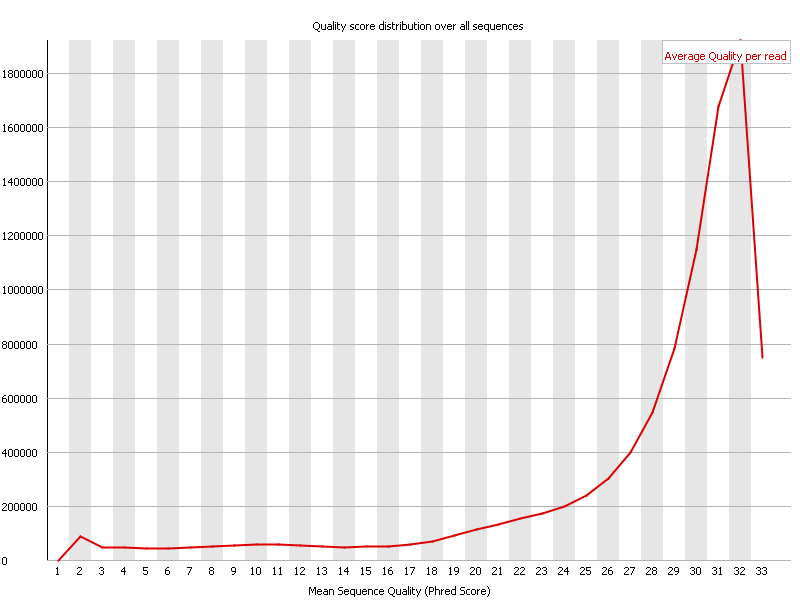
在上面的屏幕截图中,可以看到大多数读段的平均质量为 32。这通常被认为是非常好的,但是鉴于这些读段有点短,它可能充其量只是中等水平的结果。
另一个重要的指标是查看每个位置的碱基内容。假设数据是从序列空间中随机采样的,则在每个位置,贡献应该是相同的。因此,需要看到直线。实际上,第一个碱基有时会表现出一些不规则的行为,这可能是由于引物不完全随机造成的。但在所示示例中,读段完全偏离了。可以看到,在整个读段中,每个碱基的偏差都很大。事实上,这种偏差非常强烈,你甚至可以读出读段中过度表示的碱基。

例如,如果你查看最后几个碱基,你可以将它们读为 CTTGAAA - 序列结束。
如果我们现在将注意力转向 FastQC 中过度表示的序列,我们可以立即找出原因
| 序列 | 计数 | 百分比 | 可能来源 |
|---|---|---|---|
| GATCGGAAGAGCTCGTATGCCGTCTTCTGCTTGAAA | 1870684 | 19.446980406066654 | Illumina 单端适配器 1(33bp 上的 100%) |
| GAAGAGCTCGTATGCCGTCTTCTGCTTGAAAAAAAA | 95290 | 0.9906017065918623 | Illumina 单端适配器 1(28bp 上的 100%) |
如上所述,碱基调用器分配一个质量评分,该评分随后可用于每个碱基。这提供了对该碱基可靠性的估计。请注意,取决于你的测序平台,典型的错误类型不同。Illumina 最常见的错误类型是核苷酸交换,而 454、Ion Torrent/Proton 和其他类似平台在同聚物(例如 AAAAAA)方面存在重大问题,其中正确数量的 A 往往无法完全确定。
为了处理较低的质量数据,通常会去除低质量碱基。通常,会使用滑动窗口方法从例如 3' 端去除低质量碱基,因为每个碱基的质量逐渐下降。
除了去除低质量碱基数据之外,还会去除适配器、PCR 引物和其他伪影。在实践中,会将适配器剪切与质量修剪方法结合起来。
使用kmer方法纠正碱基错误有不同的方法,因为有些错误无法简单地剪切掉,即使质量值很高,也不能保证读段没有错误。这是在组装之前的一个重要步骤,但对于比对来说可能不太重要。
一个基本的想法是基于读段字符串中的kmers。最初的想法可以追溯到至少2001年(Pevzner 2001),首先生成一个kmer谱,然后将超过一定阈值的kmers(称为“solid”)和低于此阈值的kmers视为潜在的错误。
如果一个读段被分成多个kmers,一个测序错误会导致几个重叠的kmers从强变弱。错误校正步骤现在可以尝试找到使读段中所有kmers变强的最小更改次数。
像Quake这样的变体也考虑了碱基质量,以便更好地区分低拷贝真实kmers和高拷贝错误kmers。
数字归一化和分区
[edit | edit source]特别是考虑到RNA测序,众所周知,使用分子生物学技术(实验室归一化)对RNA进行归一化可以帮助提供更好的拼接结果,并且可以获得更好的总体代表性。这是因为使用实验室归一化会耗尽常见或高表达的序列。因此,在进行序列空间采样时,在实验室归一化后,找到以前非常常见的序列的可能性较小,而找到以前表达不足的序列的可能性更大。除了找到表达不足的序列的可能性更高外,还有一个额外的好处,即由于过度采样,现在不太可能在两个或多个独立读段中多次找到相同的测序错误。后者使得组装软件不太可能错误地从一个真正的mRNA中创建多个拼接结果,这是由于这些相关的SNP造成的。也就是说,实验室归一化既不容易,如果外包出去,成本很高。因此,可以使用数字归一化来替代。基本思想是下采样具有大量丰富kmers的读段。此外,这还有助于减少需要处理的读段数量,因此组装转录组可能更加可行(和更快)。一种实现方法是使用Titus Brown的工具集:http://ged.msu.edu/papers/2012-diginorm/
配对末端合并
[edit | edit source]许多工具将使用Illumina双端测序数据,并在它们之间检测到重叠时合并读段,通过在不一致的位置采用更高质量的碱基调用来纠正错误。这可以通过减少数据复杂性来提高组装器的性能,并且还可以通过去除错误数据和改善重复序列的组装来提高拼接结果。完成此操作的工具包括COPE 和FLASH.
去除其他不需要的序列
[edit | edit source]根据实验的设计,在组装之前可能还需要从读段中去除或屏蔽其他不需要的序列。例如,如果对BAC或cosmid DNA池进行测序,可能需要去除大部分甚至所有载体骨架。类似地,大肠杆菌序列会污染BAC或cosmid DNA制备物,可以在预先去除。也可以在组装后去除这些序列。PhiX控制病毒DNA是Illumina测序数据中常见的污染物。SMALT等快速搜索工具可以用来将读段映射到参考基因组,以便识别需要去除的读段。
练习
[edit | edit source]QC工作流程(机器数据 -> 修剪前质量图 -> 修剪 -> 修剪后质量图)
[edit | edit source]因此,一个典型的工作流程可能是先从机器上获取数据,然后评估上一节中所示的典型质量图。这提供了对读段质量的有效且重要的见解,并可能引起人们对可能发生的文库制备问题的注意。在识别和记录这些问题后,可以使用Trimmomatic等修剪工具去除一些错误,从序列末端去除低质量碱基,(可能更重要的是)从读段中去除剩余的接头等。在处理完读段后,再次评估质量,以了解剩余的质量问题。例如,即使在从序列中去除已知接头(如上述情况)后,仍然可能观察到每个碱基的序列偏差,并且可能希望去除这种偏差或至少将其牢记在心。我们将在此讨论一个示例工作流程。
数据集
[edit | edit source]从SRA下载SRR074262(这是上面的示例)。
Fastq输出分析
[edit | edit source]下载FastQC(或在RobiNA中分析您的数据以获得类似的图)。FastQC比较容易理解。打开刚刚下载的FastQ文件。FastQC将运行您的数据集,您只需点击左侧的各个类别,即可生成介绍中所示的图。
仅去除接头
[edit | edit source]我们将使用Trimmomatic简单地去除接头。java -jar trimmomatic-0.30.jar SE -phred33 SRR074262.fastq aclipped.fq.gz ILLUMINACLIP:TruSeq2-SE.fa:2:30:10 MINLEN:25 这告诉trimmomatic质量编码是phred 33(现代Illumina),并将结果存储在压缩文件adapter_clipped.fq.gz中。最后,它将使用trimmomatic提供的TruSeq3接头。
Trimmomatic报告
输入读段:9619406 生存:7443332 (77.38%) 丢弃:2176074 (22.62%) TrimmomaticSE:成功完成
FastQC会自动识别gz文件,因此压缩文件是可以的。实际上,当我们在FASTQC中打开aclipped.fq.gz文件时,接头已被去除。
| 序列 | 计数 | 百分比 | 可能来源 |
|---|---|---|---|
| GGGGAGGAGAGAGCCATTGTTGAGGCGGCCATTGAG | 46910 | 0.630228505190954 | 无匹配 |
| AGGGGAGGAGAGAGCCATTGTTGAGGCGGCCATTGA | 21029 | 0.28252132244000405 | 无匹配 |
去除接头和质量剪切
[edit | edit source]我们将使用Trimmomatic去除接头,并使用滑动窗口方法去除末端的低质量碱基。
java -jar trimmomatic-0.30.jar SE -phred33 SRR074262.fastq aclippedtrim.fq.gz ILLUMINACLIP:TruSeq2-SE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:25
输入读段:9619406 生存:6513310 (67.71%) 丢弃:3106096 (32.29%) TrimmomaticSE:成功完成
