三维电子显微镜/二维粒子对齐

对齐 是将粒子旋转和移动以将其放置在相似方向的过程。为了获得粒子适当的平均二维图像,使用对齐过程 [1]。大多数对齐方法要求粒子居中。在二维粒子对齐方面有多种方法。主要类型的分类涉及对参考图像的可访问性,将方法分为基于参考的对齐和无参考对齐。
基于参考的对齐涉及将每个粒子与其位置和方向与参考图像对齐的过程。在这种方法中,粒子可能会翻转,需要应用镜像方向。接下来,评估互相关系数,以确定粒子与参考图像之间的相似度。基于参考的对齐有许多优点。例如,该技术速度快,易于执行,并且由于参考是已知的,因此它允许对感兴趣粒子的准确方向进行可视化验证。另一方面,基于参考的对齐也有一些缺点。粒子对齐可能会出现偏差。这是因为假设粒子需要看起来像可用的参考图像。将粒子与有缺陷的参考图像对齐会导致噪声数据,这将看起来像参考图像。这使得在评估相似度时变得困难。需要区分相似度是真实存在还是噪声造成的。
当数据集中没有明显的组装并且参考图像不可用时,通常使用无参考对齐。这种特定技术会查找整个数据集中所有具有最佳比较方向的可能对。通过应用互相关函数来计算它。该方法利用了全局平均值的随机近似算法,并迭代直到达到收敛。无参考对齐有一些优点。该方法中使用的算法允许对一系列图像进行位移和旋转对齐。它也很快,只需要粒子的半径。这种方法的主要缺点之一是难以对具有多种形状、低信噪比或非常小的数据集的粒子进行对齐。从技术上讲,所有无参考方法都创建了一个参考来处理数据集。
经典二维对齐方法是一种基于参考的技术。它涉及在 x 和 y 坐标上的位移,然后是在平面上的图像旋转,同时与参考图像进行比较。每个旋转都伴随着图像与参考图像之间的互相关计算。最高的互相关值对应于最佳对齐。
蜘蛛方法,也称为基于环的相关,利用环。它获取像素的不同环,将其切成小圆圈,将其平铺,并堆叠这些环以进行互相关。它不进行 x, y 位移,而是通过角度来表示最佳相关值。这种方法的特点是存在大量噪声,从而导致假阳性。

拉东变换 在模式识别和图像处理等技术中非常有用。它涉及图像的横截面扫描。它在围绕其中心旋转后,对每个旋转后的图像进行平均以生成一个切片。切片堆叠是图像的最终拉东变换。该过程的下一步涉及角度近似,该角度可以通过拉东变换的 y 位移来计算,这对应于参考图像的角度。这也可以通过使用互相关来实现,其中最高峰将给出最佳角度。最后一步是将图像旋转到其参考图像。除了平均过程之外,这种方法还计算二维对齐技术中使用的误差度量和分辨率。拉东变换遵循三维重建过程,其中二维和三维的平移和旋转对齐可以完成 [2]。
最大似然 或 ML 是一种无参考方法,它涉及找到最有可能的模型来拟合获得的数据。在这种方法中,测量观察值的概率。这些是基于原始模型,并且它是互相关技术的替代方法,该技术基于整个数据集的差异做出选择。ML 方法评估粒子的相对方向,这些方向被处理为隐变量,这些变量被纳入似然计算。这种 ML 细化方法还考虑了误差模型,这反过来又减少了大型数据集中的偏差 [3]。它是最慢的方法之一,因为它涉及大型数据集。

分类是三维重建步骤之前的关键步骤。重建所使用的单个图像需要满足严格的要求。这些是研究中同一个生物样本的不同视图。它允许从同质种群中分离出粒子的不同视图。就像 Gro-El 分子一样,分类应用于分离顶部视图和侧面视图 (Pascual-Montano 233-245)。
主成分分析 用于图像识别和压缩技术。它被称为一种数学方法,用于将事物分组在一起,然后降低其维度,同时保留最接近原始数据集的数据。当记录的变量之间存在强相关性时,此属性效果很好,因为它沿特征向量投影数据。首先,它将图像转换为大型矩阵数据。接下来,它分析不同视图的关键特征,并创建分配了特征值的特征图像。该方法的主要功能是减少冗余、提取一般特征以及对图像和数据压缩进行预测。这种方法通常用于人脸识别过程中,它可以找到将图像类别区分开来的关键特征[4]。
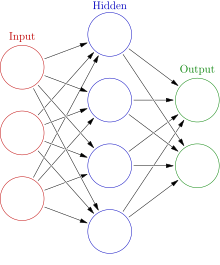
自组织映射 是一种出色的数据映射和分类方法,当数据集和类别数量很大时,它非常有用。如果类别之间的边界不清晰,这将特别有用。SOM 也被称为 Kohonen 自组织映射,因为这些是在 1997 年由 Kohenen 首次发现的[5]。SOM 是将高维数据非线性投影到低维空间。神经网络的结构由 3 层组成。分别是输入层、中间隐藏层和输出层。SOM 模拟了人脑在输入数据呈现给它时进行的自组织过程。
- ↑ "二维粒子对齐方法。"沃兹沃斯中心,[在线]。于 2013 年 11 月 16 日访问。可从以下网址获取:http://spider.wadsworth.org/spider_doc/spider/docs/align.html
- ↑ Radermacher. M.,(1997) 用于随机投影对齐和三维重建的 Radon 变换技术。扫描显微镜,第 11 卷,(171-177)
- ↑ Scheres.S.H.W.,Valle.M.,Nunez.R.,Sorzano.C.O.S.,Marabini.R.,Herman.G.T.,Carazao.J.M.,(2005) 用于电子显微镜图像的极大似然多参考细化。J. Mol. Biol. 348, (139–149)
- ↑ Kyungnam.K.,“使用主成分分析的人脸识别”马里兰大学,[在线]。于 2013 年 11 月 16 日访问。可从以下网址获取:http://www.umiacs.umd.edu/~knkim/KG_VISA/PCA/FaceRecog_PCA_Kim.pdf
- ↑ Pascual-Montano A.,Donate L.E.,Valle M.,Barcena M.,Pascual-Marqui R.D.,Carazo J.M.,(2001) 用于分析和分类 EM 单粒子图像的新型神经网络技术。结构生物学杂志 133,233-245
