分子生物学导论/蛋白质的功能和结构
蛋白质最早由荷兰化学家Gerhardus Johannes Mulder于1838年描述,并由瑞典化学家Jöns Jakob Berzelius命名。早期的营养学家,如德国的Carl von Voit,认为蛋白质是维持身体结构最重要的营养素,因为人们普遍认为“肉长肉”。
多肽链中的氨基酸通过肽键连接。一旦在蛋白质链中连接,单个氨基酸被称为残基,连接的碳、氮和氧原子系列被称为主链或蛋白质骨架。肽键具有两种共振形式,它们贡献了一些双键特征并抑制了其轴线周围的旋转,因此α碳大致共面。肽键中的另外两个二面角决定了蛋白质骨架所假设的局部形状。具有游离羧基的一端称为C端或羧基端,而具有游离氨基的一端称为N端或氨基端。
蛋白质、多肽和肽这些词语有些模糊,含义可能重叠。蛋白质通常用于指代处于稳定构象的完整生物分子,而肽通常保留用于指代短的氨基酸寡聚体,这些寡聚体通常缺乏稳定的三维结构。然而,两者之间的界限并不明确,通常在20-30个残基附近。多肽可以指任何单个线性氨基酸链,通常与长度无关,但通常意味着缺乏定义的精氨酸构象。[1]


有22种标准氨基酸,但只有21种存在于真核生物中。在这22种中,20种直接由通用遗传密码编码。人类可以从彼此或从其他中间代谢分子中合成这20种氨基酸中的11种。另外9种必须从饮食中摄取,因此被称为必需氨基酸;它们是组氨酸、异亮氨酸、亮氨酸、赖氨酸、蛋氨酸、苯丙氨酸、苏氨酸、色氨酸和缬氨酸。另外两种,硒代半胱氨酸和吡咯赖氨酸,通过独特的合成机制整合到蛋白质中。
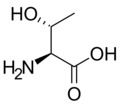
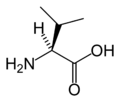
每个α-氨基酸都由一个所有氨基酸类型中都存在的骨架部分和一个特定于每种残基类型的侧链组成。脯氨酸是这条规则的例外,其中氢原子被与侧链的键取代。由于碳原子与四个不同的基团结合,因此它是手性的,但生物蛋白质中只存在一种异构体。然而,甘氨酸不是手性的,因为它的侧链是一个氢原子。一个简单的助记符来记忆正确的L-形式是“CORN”:当以H在前的视角观察Cα原子时,残基以“CO-R-N”的顺时针方向排列。[2]
异构体
标准α-氨基酸,除甘氨酸外,都可以存在于两种光学异构体中,称为L或D氨基酸,它们是彼此的镜像。虽然L-氨基酸代表了在核糖体翻译过程中发现的所有蛋白质中的氨基酸,但D-氨基酸存在于一些由酶翻译后修饰后产生的蛋白质中,这些修饰发生在翻译后并易位到内质网,例如在锥形蜗牛等奇异的海生生物中。它们也是细菌肽聚糖细胞壁的丰富成分,并且D-丝氨酸可能在大脑中充当神经递质。氨基酸构型的L和D约定不指氨基酸本身的光学活性,而是指理论上可以合成该氨基酸的甘油醛异构体的光学活性(D-甘油醛是右旋的;L-甘油醛是左旋的)。或者,(S)和(R)指定用于指示绝对立体化学。蛋白质中几乎所有氨基酸在α碳处都是(S),其中半胱氨酸是(R),甘氨酸是非手性的。半胱氨酸很特殊,因为它的侧链中第二个位置有一个硫原子,它的原子质量比连接到第一个碳的基团更大,而第一个碳连接到其他标准氨基酸的α碳,因此是(R)而不是(S)。[3]
两性离子
氨基酸中发现的胺和羧酸官能团使其具有两性性质。在被称为等电点的特定pH值下,氨基酸没有总电荷,因为质子化的氨基数量(正电荷)和去质子化的羧酸根数量(负电荷)相等。所有氨基酸都有不同的等电点。在等电点产生的离子同时具有正负电荷,被称为两性离子,这个词来自德语单词Zwitter,意思是“雌雄同体”或“混合体”。氨基酸可以在固体和水等极性溶液中以两性离子的形式存在,但在气相中不存在。两性离子在其等电点处具有最小的溶解度,可以通过调节pH值使其达到特定的等电点来从水中沉淀出来,从而分离氨基酸。[4]
20种天然存在的氨基酸具有不同的物理和化学性质,包括它们的静电电荷、pKa、疏水性、尺寸和特定官能团。这些性质在塑造蛋白质结构中起着重要作用。下表描述了氨基酸的主要特征。
| 氨基酸 | 缩写 | 备注 | |
|---|---|---|---|
丙氨酸 |
A | Ala | 非常丰富,用途广泛。比甘氨酸更硬,但足够小,只对蛋白质构象构成微小的空间限制。它表现得相当中性,可以位于蛋白质外部的亲水区域和内部的疏水区域。 |
| 天冬酰胺或天冬氨酸 | B | Asx | 当两种氨基酸都可能占据一个位置时的占位符。 |
半胱氨酸 |
C | Cys | 硫原子易与重金属离子结合。在氧化条件下,两个半胱氨酸可以连接在一起形成二硫键,形成氨基酸胱氨酸。当胱氨酸是蛋白质的一部分时,例如胰岛素,其三级结构会得到稳定,这使得蛋白质更能抵抗变性;因此,二硫键在必须在恶劣环境中发挥作用的蛋白质中很常见,包括消化酶(例如,胃蛋白酶和胰凝乳蛋白酶)和结构蛋白(例如,角蛋白)。二硫键也存在于本身无法保持稳定形状的肽中(例如胰岛素)。 |
天冬氨酸 |
D | Asp | 行为类似于谷氨酸。带有一个带强负电荷的亲水性酸性基团。通常位于蛋白质的外表面,使其可溶于水。与带正电荷的分子和离子结合,常用于酶中固定金属离子。当位于蛋白质内部时,天冬氨酸和谷氨酸通常与精氨酸和赖氨酸配对。 |
谷氨酸 |
E | Glu | 行为类似于天冬氨酸。具有更长、更灵活的侧链。 |
苯丙氨酸 |
F | Phe | 对人类至关重要。苯丙氨酸、酪氨酸和色氨酸在侧链上含有大的刚性芳香基团。这些是最大的氨基酸。与异亮氨酸、亮氨酸和缬氨酸一样,它们是疏水的,并且倾向于朝向折叠的蛋白质分子的内部定向。苯丙氨酸可以转化为酪氨酸。 |
甘氨酸 |
G | Gly | 由于α碳上的两个氢原子,甘氨酸没有旋光性。它是最小的氨基酸,易于旋转,为蛋白质链添加了灵活性。它能够适应最紧密的间隙,例如胶原蛋白的三螺旋。由于过多的灵活性通常不可取,因此作为结构组分,它不如丙氨酸常见。 |
组氨酸 |
H | His | 即使在弱酸性条件下,氮的质子化也会发生,改变组氨酸和整个多肽的性质。它被许多蛋白质用作调节机制,在酸性区域(例如晚期内体或溶酶体)中改变多肽的构象和行为,强制酶发生构象变化。然而,这只需要很少的组氨酸,因此它相对稀少。 |
异亮氨酸 |
I | Ile | 对人类至关重要。异亮氨酸、亮氨酸和缬氨酸具有大的脂肪族疏水侧链。它们的分子是刚性的,它们的相互疏水相互作用对于蛋白质的正确折叠至关重要,因为这些链往往位于蛋白质分子的内部。 |
| 亮氨酸或异亮氨酸 | J | Xle | 当任一氨基酸可能占据一个位置时的占位符 |
赖氨酸 |
K | Lys | 对人类至关重要。行为类似于精氨酸。包含一个长而灵活的侧链,其末端带正电荷。链的灵活性使得赖氨酸和精氨酸适合与表面上具有多个负电荷的分子结合。例如,DNA结合蛋白的活性区域富含精氨酸和赖氨酸。强电荷使得这两种氨基酸容易位于蛋白质的外部亲水表面;当它们位于内部时,它们通常与相应的带负电荷的氨基酸配对,例如天冬氨酸或谷氨酸。 |
亮氨酸 |
L | Leu | 对人类至关重要。行为类似于异亮氨酸和缬氨酸。参见异亮氨酸。 |
蛋氨酸 |
M | Met | 对人类至关重要。始终是第一个被整合到蛋白质中的氨基酸;有时在翻译后被移除。与半胱氨酸一样,含有硫,但带有甲基而不是氢。该甲基可以被激活,并用于许多反应中,在这些反应中,新的碳原子被添加到另一个分子中。 |
天冬酰胺 |
N | Asn | 类似于天冬氨酸。Asn 包含一个酰胺基团,而 Asp 具有一个羧基。 |
| 吡咯赖氨酸 | O | Pyl | 类似于赖氨酸,带有吡咯烷环。 |
脯氨酸 |
P | Pro | 对 N 末端胺基含有不寻常的环,这迫使 CO-NH 酰胺序列处于固定构象。可以破坏蛋白质折叠结构,例如α 螺旋或β 折叠,迫使蛋白质链产生所需的弯曲。在胶原蛋白中很常见,在那里它经常发生翻译后修饰形成羟脯氨酸。 |
谷氨酰胺 |
Q | Gln | 类似于谷氨酸。Gln 包含一个酰胺基团,而 Glu 具有一个羧基。用于蛋白质中,并作为氨的储存。体内最丰富的氨基酸。 |
精氨酸 |
R | Arg | 在功能上类似于赖氨酸。 |
丝氨酸 |
S | Ser | 丝氨酸和苏氨酸有一个短的基团,其末端是羟基基团。它的氢很容易被移除,因此丝氨酸和苏氨酸经常在酶中充当氢供体。两者都非常亲水,因此可溶性蛋白质的外区域往往富含它们。 |
苏氨酸 |
T | Thr | 对人类至关重要。行为类似于丝氨酸。 |
| 硒代半胱氨酸 | U | Sec | 硒化形式的半胱氨酸,它取代了硫。 |
缬氨酸 |
V | Val | 对人类至关重要。行为类似于异亮氨酸和亮氨酸。参见异亮氨酸。 |
色氨酸 |
W | Trp | 对人类至关重要。行为类似于苯丙氨酸和酪氨酸(参见苯丙氨酸)。血清素的前体。天然荧光。 |
| 未知 | X | Xaa | 当氨基酸未知或不重要时的占位符。 |
酪氨酸 |
Y | Tyr | 行为类似于苯丙氨酸(酪氨酸的前体)和色氨酸(参见苯丙氨酸)。黑色素、肾上腺素和甲状腺激素的前体。天然荧光,尽管荧光通常会被能量转移到色氨酸而猝灭。 |
| 谷氨酸或谷氨酰胺 | Z | Glx | 当两种氨基酸都可能占据一个位置时的占位符。 |
-
L-丙氨酸
(Ala / A) -
L-精氨酸
(Arg / R) -
L-天冬酰胺
(Asn / N) -
L-天冬氨酸
(Asp / D) -
L-半胱氨酸
(Cys / C) -
L-谷氨酸
(Glu / E) -
L-谷氨酰胺
(Gln / Q) -
甘氨酸
(Gly / G) -
L-组氨酸
(His / H) -
L-异亮氨酸
(Ile / I) -
L-亮氨酸
(Leu / L) -
L-赖氨酸
(Lys / K) -
L-蛋氨酸
(Met / M) -
L-苯丙氨酸
(Phe / F) -
L-脯氨酸
(Pro / P) -
L-丝氨酸
(Ser / S) -
L-苏氨酸
(Thr / T) -
L-色氨酸
(Trp / W) -
L-酪氨酸
(Tyr / Y) -
L-缬氨酸
(Val / V) -
L-硒代半胱氨酸
(Sec / U) -
L-吡咯赖氨酸
(Pyl / O)
氨基酸分类
[edit | edit source]由遗传密码直接编码的 20 种氨基酸可以根据它们的性质分为几组。重要的因素包括电荷、亲水性或疏水性、大小和官能团。氨基酸通常根据其侧链的性质分为四组。侧链可以使氨基酸成为弱酸或弱碱,以及如果侧链是极性的亲水性物质,或者如果侧链是非极性的疏水性物质。

蛋白质氨基酸通过缩合反应组合成单个多肽链。这种反应是由催化的核糖体在称为翻译的过程中进行的。
| 必需 | 非必需 |
|---|---|
| 异亮氨酸 | 丙氨酸 |
| 亮氨酸 | 天冬酰胺 |
| 赖氨酸 | 天冬氨酸 |
| 蛋氨酸 | 半胱氨酸* |
| 苯丙氨酸 | 谷氨酸 |
| 苏氨酸 | 谷氨酰胺* |
| 色氨酸 | 甘氨酸* |
| 缬氨酸 | 脯氨酸* |
| 硒代半胱氨酸* | |
| 丝氨酸* | |
| 酪氨酸* | |
| 精氨酸* | |
| 组氨酸* | |
| 鸟氨酸* | |
| 牛磺酸* |
极性和非极性氨基酸及其单字母和三字母代码
| 氨基酸 | 三字母代码 | 单字母代码 | 侧链极性 | 侧链电荷 (pH 7.4) | 疏水性指数 | 吸光度 λmax(nm) | ε 在 λmax (x10−3 M−1 cm−1) |
|---|---|---|---|---|---|---|---|
| 丙氨酸 | Ala | A | 非极性 | 中性 | 1.8 | ||
| 精氨酸 | Arg | R | 极性 | 正 | −4.5 | ||
| 天冬酰胺 | Asn | N | 极性 | 中性 | −3.5 | ||
| 天冬氨酸 | Asp | D | 极性 | 负 | −3.5 | ||
| 半胱氨酸 | Cys | C | 非极性 | 中性 | 2.5 | 250 | 0.3 |
| 谷氨酸 | Glu | E | 极性 | 负 | −3.5 | ||
| 谷氨酰胺 | Gln | Q | 极性 | 中性 | −3.5 | ||
| 甘氨酸 | Gly | G | 非极性 | 中性 | −0.4 | ||
| 组氨酸 | His | H | 极性 | 正(10%) 中性(90%) |
−3.2 | 211 | 5.9 |
| 异亮氨酸 | Ile | I | 非极性 | 中性 | 4.5 | ||
| 亮氨酸 | Leu | L | 非极性 | 中性 | 3.8 | ||
| 赖氨酸 | Lys | K | 极性 | 正 | −3.9 | ||
| 蛋氨酸 | Met | M | 非极性 | 中性 | 1.9 | ||
| 苯丙氨酸 | Phe | F | 非极性 | 中性 | 2.8 | 257, 206, 188 | 0.2, 9.3, 60.0 |
| 脯氨酸 | Pro | P | 非极性 | 中性 | −1.6 | ||
| 丝氨酸 | Ser | S | 极性 | 中性 | −0.8 | ||
| 苏氨酸 | Thr | T | 极性 | 中性 | −0.7 | ||
| 色氨酸 | Trp | W | 非极性 | 中性 | −0.9 | 280, 219 | 5.6, 47.0 |
| 酪氨酸 | Tyr | Y | 极性 | 中性 | −1.3 | 274, 222, 193 | 1.4, 8.0, 48.0 |
| 缬氨酸 | Val | V | 非极性 | 中性 | 4.2 |
此外,还有两种额外的氨基酸通过覆盖终止密码子来整合
| 第 21 和 22 个氨基酸 | 3 字母 | 1 字母 |
|---|---|---|
| 硒代半胱氨酸 | Sec | U |
| 吡咯赖氨酸 | Pyl | O |
除了特定的氨基酸代码外,当化学或晶体学分析无法确定肽或蛋白质中残基的身份时,会使用占位符。
| 模棱两可的氨基酸 | 3 字母 | 1 字母 |
|---|---|---|
| 天冬酰胺或天冬氨酸 | Asx | B |
| 谷氨酰胺或谷氨酸 | Glx | Z |
| 亮氨酸或异亮氨酸 | Xle | J |
| 未指定或未知的氨基酸 | Xaa | X |
Unk有时用作Xaa的替代,但不如Xaa标准。
此外,许多非标准氨基酸有特定的代码。例如,一些肽类药物,如硼替佐米或MG132是人工合成的,并保留其保护基团,这些基团有特定的代码。硼替佐米是Pyz-Phe-boroLeu,而MG132是Z-Leu-Leu-Leu-al。此外,为了帮助分析蛋白质结构,光交联氨基酸类似物也可用。这些包括光亮氨酸 (pLeu) 和光甲硫氨酸 (pMet)。[5]
肽键
[edit | edit source]
肽键(酰胺键)是两个分子之间形成的共价化学键,当一个分子的羧基与另一个分子的氨基反应时形成,释放一个水分子 (H2O)。这是一个脱水合成反应(也称为缩合反应),通常发生在氨基酸之间。形成的 C(O)NH 键称为肽键,形成的分子为酰胺。四原子官能团 -C(=O)NH- 称为肽键。多肽和蛋白质是通过肽键连接在一起的氨基酸链,就像 PNA 的主链一样。
肽键可以通过酰胺水解(加水)断裂。蛋白质中的肽键是亚稳的,这意味着在水存在的情况下,它们会自发断裂,释放 2-4 kcal/mol 的自由能,但这个过程非常缓慢。在生物体中,这个过程是由酶加速的。生物体也利用酶形成肽键;这个过程需要自由能。肽键的吸收波长为 190-230 nm。
由于双键电子的离域,肽键倾向于平面。刚性的肽二面角 ω(C1 和 N 之间的键)始终接近 180 度。二面角 phi φ(N 和 Cα 之间的键)和 psi ψ(Cα 和 C1 之间的键)可以具有一定的可能值范围。这些角度是蛋白质的内部自由度,它们控制着蛋白质的构象。它们受几何形状限制,限制在特定二级结构元素的允许范围内,并在拉马钱德兰图中表示。下表给出了一些重要的键长。[6]
| 肽键 | 平均长度 | 单键 | 平均长度 | 氢键 | 平均值(±30) |
| Ca -C | 153 pm | C - C | 154 pm | O-H --- O-H | 280 pm |
| C - N | 133 pm | C - N | 148 pm | N-H --- O=C | 290 pm |
| N - Ca | 146 pm | C - O | 143 pm | O-H --- O=C | 280 pm |
β-肽
[edit | edit source]在 α 氨基酸(左侧的分子)中,羧酸基团(红色)和氨基基团(蓝色)都连接到同一个碳中心,称为 α 碳 (),因为它距离羧酸基团一个原子。在 β 氨基酸中,氨基连接到 β 碳 (),它存在于 20 种标准氨基酸中的大多数。只有甘氨酸没有 β 碳,这意味着 β-甘氨酸是不可能的。
β 氨基酸的化学合成可能具有挑战性,特别是考虑到连接到 β 碳的官能团的多样性以及维持手性的必要性。在所示的丙氨酸分子中,β 碳是非手性的;然而,大多数较大的氨基酸都有手性 原子。已经引入了许多合成机制来有效地形成 β 氨基酸及其衍生物[7][8],特别是那些基于阿恩特-艾斯特合成的。
存在两种主要的 β-肽类型:有机残基 (R) 靠近胺的称为 β3-肽,而位置靠近羰基的称为 β2-肽。[9]
酶
[edit | edit source]酶通常是球状蛋白质,大小从 4-草酰巴豆酸互变异构酶单体的 62 个氨基酸残基到动物脂肪酸合酶的 2500 多个残基不等。少数基于 RNA 的生物催化剂存在,最常见的是核糖体;这些被称为 RNA 酶或核酶。酶的活性由其三维结构决定。然而,尽管结构决定功能,但仅仅从结构预测一种新的酶的活性是一个非常困难的问题,目前还没有解决。
大多数酶比它们作用的底物大得多,只有酶的一小部分(大约 3-4 个氨基酸)直接参与催化。包含这些催化残基、结合底物并进行反应的区域被称为活性位点。酶也可以包含结合辅因子的位点,辅因子对于催化是必需的。一些酶还具有结合小分子的结合位点,这些小分子通常是催化反应的直接或间接产物或底物。这种结合可以提高或降低酶的活性,提供了一种反馈调节的手段。与所有蛋白质一样,酶是折叠成三维产物的长线性氨基酸链。每个独特的氨基酸序列都会产生特定的结构,该结构具有独特的性质。单个蛋白质链有时可以聚集成一起形成蛋白质复合物。大多数酶可以被热或化学变性剂变性,即展开和失活,这会破坏蛋白质的三维结构。根据酶的不同,变性可能是可逆的或不可逆的。使用时间分辨晶体学方法可以获得酶与底物或底物类似物在反应过程中形成的复合物的结构。[10]
酶的分类
[edit | edit source]酶的名称通常源于其底物或其催化的化学反应,其词尾为 -ase。例如乳糖酶、醇脱氢酶和 DNA 聚合酶。这可能导致具有相同功能的不同酶,称为同工酶,具有相同的名称。同工酶具有不同的氨基酸序列,可以通过其最佳 pH 值、动力学性质或免疫学来区分。同工酶和同工酶是同源蛋白。此外,酶催化的正常生理反应可能与人工条件下不同。这可能导致同一酶被赋予两个不同的名称。例如,工业上用于将葡萄糖转化为甜味剂果糖的葡萄糖异构酶,在体内是木糖异构酶。
国际生物化学与分子生物学联盟制定了酶的命名法,即 EC 编号。酶委员会编号 (EC 编号) 是基于酶催化的化学反应对酶进行的数值分类方案。作为酶命名的系统,每个 EC 编号都与相应酶的推荐名称相关联。每种酶都由一个以“EC”开头的四位数序列来描述。第一个数字根据其机制将酶广泛分类。严格地说,EC 编号不指定酶,而是指定酶催化的反应。如果不同的酶(例如来自不同生物体的酶)催化相同的反应,那么它们将获得相同的 EC 编号。相比之下,UniProt 标识符通过其氨基酸序列唯一地指定蛋白质。[11]
EC 1 氧化还原酶:催化氧化/还原反应
EC 2 转移酶:转移一个官能团(例如甲基或磷酸基团)
EC 3 水解酶:催化各种键的水解
EC 4 裂解酶:通过除水解和氧化以外的其他方式断裂各种键
EC 5 异构酶:催化单个分子内的异构化变化
EC 6 连接酶:通过共价键连接两个分子。
| 组 | 催化的反应 | 典型的反应 | 具有俗名的酶示例 |
|---|---|---|---|
| EC 1 氧化还原酶 |
催化氧化/还原反应;将 H 和 O 原子或电子从一种物质转移到另一种物质 | AH + B → A + BH (还原) A + O → AO (氧化) |
脱氢酶,氧化酶 |
| EC 2 转移酶 |
将一个官能团从一种物质转移到另一种物质。该基团可以是甲基、酰基、氨基或磷酸基团 | AB + C → A + BC | 转氨酶,激酶 |
| EC 3 水解酶 |
通过水解从底物形成两种产物 | AB + H2O → AOH + BH | 脂肪酶,淀粉酶,肽酶 |
| EC 4 裂解酶 |
底物非水解添加或去除基团。C-C、C-N、C-O 或 C-S 键可能会断裂 | RCOCOOH → RCOH + CO2 或 [x-A-B-Y] → [A=B + X-Y] | 脱羧酶 |
| EC 5 异构酶 |
分子内重排,即单个分子内的异构化变化 | AB → BA | 异构酶,变位酶 |
| EC 6 连接酶 |
通过合成新的 C-O、C-S、C-N 或 C-C键将两个分子连接在一起,同时分解ATP | X + Y+ ATP → XY + ADP + Pi | 合成酶 |
氧化还原酶
[edit | edit source]在分子生物学和生物化学中,氧化还原酶是一种催化电子从一个分子(还原剂,也称为氢或电子供体)转移到另一个分子(氧化剂,也称为氢或电子受体)的酶。这组酶通常利用 NADP 或 NAD 作为辅因子。一般来说,多肽是无支链的聚合物,因此它们的初级结构通常可以通过沿其主链的氨基酸序列来指定。然而,蛋白质可以发生交联,最常见的是通过二硫键,并且初级结构还需要指定交联原子,例如,指定蛋白质中参与二硫键的半胱氨酸。其他交联包括脱氢脯氨酸……多肽链的手性中心可能会发生外消旋化。特别是,蛋白质中通常发现的 L-氨基酸可以在 Cα 原子上自发异构化形成 D-氨基酸,D-氨基酸不能被大多数蛋白酶裂解。[13]
蛋白质的结构
[edit | edit source]
蛋白质的初级结构
[edit | edit source]
蛋白质是 α-氨基酸的线性链的提议几乎同时由两位科学家在 1902 年的同一会议上提出,即第 74 届德国科学家和医师学会会议,在卡尔斯巴德举行。弗朗茨·霍夫迈斯特在上午提出了这个提议,基于他对蛋白质中双缩脲反应的观察。几个小时后,霍夫迈斯特被埃米尔·费歇尔接替,费歇尔积累了大量支持肽键模型的化学细节。为了完整起见,蛋白质含有酰胺键的提议早在 1882 年就由法国化学家E. Grimaux提出。[15]
尽管有这些数据以及后来的证据表明蛋白水解消化的蛋白质仅产生寡肽,但蛋白质是氨基酸的线性、无支链聚合物的观点并没有立即被接受。一些德高望重的科学家,如威廉·阿斯特伯里,怀疑共价键是否足够强以将如此长的分子结合在一起;他们担心热振动会将如此长的分子震碎。赫尔曼·施陶丁格在 1920 年代面临着类似的偏见,当时他认为橡胶是由大分子组成的。因此,出现了几个替代假设。胶体蛋白假说指出蛋白质是较小分子的胶体集合。这个假设在 1920 年代被特奥多尔·斯维德伯格的超速离心测量所驳斥,超速离心测量表明蛋白质具有明确的、可重复的分子量,以及阿恩·蒂塞利乌斯进行的电泳测量,表明蛋白质是单个分子。
第二个假设是环醇假说,由多萝西·温奇提出,该假说提出线性多肽经历了一种化学环醇重排 C=O + HN C(OH)-N,其交联了其主链酰胺基团,形成了二维织物。各种研究人员提出了蛋白质的其他初级结构,例如埃米尔·阿贝德哈尔登的二酮哌嗪模型和 1942 年特罗恩斯高德的吡咯/哌啶模型。尽管从未得到太多重视,但当弗雷德里克·桑格成功地对胰岛素进行测序以及麦克斯·佩鲁茨和约翰·肯德鲁通过晶体学测定肌红蛋白和血红蛋白时,这些替代模型最终被否定了。
肽和蛋白质的初级结构是指其氨基酸结构单元的线性序列。“初级结构”一词最初由林德斯特罗姆-朗在 1951 年提出。按照惯例,蛋白质的初级结构从氨基末端 (N) 端到羧基末端 (C) 端报告。蛋白质的翻译后修饰,如二硫键形成、磷酸化和糖基化,通常也被认为是初级结构的一部分,无法从基因中读取。
蛋白质的二级结构
[edit | edit source]
二级结构是指高度规则的局部亚结构。两种主要的二级结构,α螺旋 和 β折叠,是由莱纳斯·鲍林及其同事在 1951 年提出的。[16] 这些二级结构由主链肽基团之间的氢键模式定义。它们具有规则的几何形状,被限制在拉氏图上的二面角 ψ 和 φ 的特定值。α 螺旋和 β 折叠都代表了一种使肽骨架中所有氢键供体和受体饱和的方式。蛋白质中的一些部分是有序的,但没有形成任何规则的结构。它们不应该与无规卷曲混淆,无规卷曲是指缺乏任何固定三维结构的未折叠多肽链。几个连续的二级结构可以形成一个 "超二级结构单元"。[17]

氨基酸在形成各种二级结构元素的能力上有所不同。脯氨酸和甘氨酸有时被称为“螺旋破坏者”,因为它们会破坏 α 螺旋主链构象的规律性;然而,它们两者都具有不同寻常的构象能力,并且通常存在于转角中。倾向于在蛋白质中采用螺旋构象的氨基酸包括蛋氨酸、丙氨酸、亮氨酸、谷氨酸和赖氨酸(在氨基酸单字母代码中为“MALEK”);相反,大的芳香族残基(色氨酸、酪氨酸和苯丙氨酸)和 Cβ 支链氨基酸(异亮氨酸、缬氨酸和苏氨酸)倾向于采用 β 折叠构象。然而,这些偏好不足以产生一种可靠的方法,仅从序列中预测二级结构。蛋白质中的二级结构由氢键介导的局部残基间相互作用组成,或不组成。最常见的二级结构是 α 螺旋和 β 折叠。其他螺旋,如 310 螺旋和 π 螺旋,被计算出具有能量上有利的氢键模式,但在天然蛋白质中很少观察到,除非在 α 螺旋的末端,因为螺旋中心的骨架堆积不利。
拉氏图(也称为拉氏图或拉氏图或 [φ,ψ] 图),由 Gopalasamudram Narayana Ramachandran 和 Viswanathan Sasisekharan 开发,是一种可视化蛋白质结构中氨基酸残基的二面角 ψ 相对于 φ 的方法。[18] 它显示了多肽的 ψ 和 φ 角的可能构象。
在数学上,拉氏图是函数 的可视化。该函数的定义域是环面。因此,传统的拉氏图是环面在平面上的投影,导致失真视图和不连续性的出现。可以预期,较大的侧链会导致更多的限制,因此拉氏图中允许的区域更小。在实践中,情况似乎并非如此;只有 α 位置的亚甲基基团有影响。甘氨酸在 α 位置有一个氢原子,其范德华半径小于甲基基团。因此,它的限制最少,这在甘氨酸的拉氏图中很明显,对于甘氨酸,允许的区域要大得多。相反,脯氨酸的拉氏图仅显示了 ψ 和 φ 的非常有限的可能组合。拉氏图是在第一个原子分辨率的蛋白质结构确定之前计算出来的。40 年后,通过 X 射线晶体学确定了数万个高分辨率蛋白质结构,并沉积到蛋白质数据库 (PDB) 中。从 1000 个不同的蛋白质链中,绘制了超过 200 000 个氨基酸的拉氏图,显示了一些显著的差异,特别是对于甘氨酸(Hovmöller 等人,2002)。发现左上角区域被分成两部分;一个在左边包含 β 折叠中的氨基酸,另一个在右边包含这种构象的无规卷曲中的氨基酸。也可以用这种方式绘制多糖和其他聚合物的二面角。对于前两个蛋白质侧链二面角,类似的图是 Janin 图。
α 螺旋
α 螺旋中的氨基酸排列成右手螺旋结构,其中每个氨基酸残基对应于螺旋的 100° 转弯(即螺旋每转 3.6 个残基),以及沿螺旋轴的 1.5 Å(0.15 nm)平移。(短段左手螺旋有时会出现在大量非手性甘氨酸氨基酸中,但对其他正常的生物 L-氨基酸来说是不利的。)α 螺旋的螺距(螺旋的一个连续转弯之间的垂直距离)为 5.4 Å(0.54 nm),它是 1.5 和 3.6 的乘积。最重要的是,一个氨基酸的 N-H 基团与前面四个残基的 C=O 基团形成氢键;这种重复的氢键是 α 螺旋最突出的特征。官方国际命名法指定了两种定义 α 螺旋的方法,规则 6.2 是根据重复的 φ、ψ 扭转角,规则 6.3 是根据螺距和氢键的组合模式。不同的氨基酸序列具有形成 α 螺旋结构的不同倾向。蛋氨酸、丙氨酸、亮氨酸、未带电荷的谷氨酸和赖氨酸(在氨基酸单字母代码中为“MALEK”)都具有特别高的螺旋形成倾向,而脯氨酸和甘氨酸则具有较差的螺旋形成倾向。脯氨酸要么破坏螺旋,要么使螺旋弯曲,这两种情况都是因为脯氨酸不能捐赠酰胺氢键(因为没有酰胺氢),而且它的侧链会与前一个螺旋的骨架发生空间位阻。在一个螺旋中,这会导致螺旋轴弯曲约 30°。[19] 然而,脯氨酸通常被视为螺旋的第一个残基,这可能是由于其结构刚性。在另一个极端,甘氨酸也往往会破坏螺旋,因为它很高的构象灵活性使其在熵上采用相对受限的 α 螺旋结构很昂贵。

β 折叠 第一个 β 折叠结构是由威廉·阿斯特伯里在 1930 年代提出的。他提出了平行或反平行延伸 β 折叠之间形成氢键的想法。然而,阿斯特伯里没有关于氨基酸键几何形状的必要数据来构建准确的模型,特别是他当时不知道肽键是平面的。莱纳斯·鲍林和罗伯特·科里在 1951 年提出了一个改进的版本。
β 折叠(也称为 β 折叠片)是蛋白质中第二种规则的二级结构,只是比 α 螺旋略不常见。β 折叠由至少两个或三个主链氢键横向连接的 β 折叠组成,形成一个通常扭曲的、褶皱的片。β 折叠(也称为 β 折叠)是多肽链的一段,通常长 3 到 10 个氨基酸,主链处于几乎完全伸展的构象中。
涉及 β 折叠的非常简单的结构模体 是β 折叠发夹,其中两个反平行折叠通过一个 2 到 5 个残基的短环连接起来,其中一个通常是甘氨酸或脯氨酸,它们两者都可以假设紧凑转角所需的非寻常二面角构象。然而,单个折叠也可以以更复杂的方式连接起来,形成长环,这些长环可能包含α 螺旋,甚至整个蛋白质结构域。
希腊键模体 希腊键模体由四个相邻的反平行折叠及其连接环组成。它由三个由发夹连接的反平行折叠组成,而第四个折叠与第一个折叠相邻,并通过更长的环连接到第三个折叠。这种类型的结构在蛋白质折叠过程中很容易形成。[20][21] 它以希腊装饰艺术中常见的图案命名(见回纹)。
β-α-β 基序 由于其组成氨基酸的手性,所有链在大多数高级 β 折叠结构中都表现出“右手”扭曲。特别是,两个平行链之间的连接环几乎总是具有右手交叉手性,这受到片材固有扭曲的强烈偏爱。此连接环通常包含一个螺旋区域,在这种情况下,它被称为 β-α-β 基序。一个密切相关的基序被称为 β-α-β-α 基序,它构成了最常见观察到的蛋白质 三级结构,即 TIM 桶 的基本组成部分。
β-弯曲基序 一个简单的 超二级 蛋白质拓扑结构,由 2 个或多个连续的反平行 β 链组成,通过 发夹 环连接在一起。 [22] [23] 此基序在 β 折叠中很常见,可以在几种结构体系中找到,包括 β 桶 和 β 推进器。

Psi 环基序 Psi 环,Ψ 环,基序由两个反平行链组成,一个链介于两者之间,通过氢键连接到两者。 [24] 如 Hutchinson 等人 (1990) 所述,单一 Ψ 环有四种可能的链拓扑结构。此基序很少见,因为导致其形成的过程似乎不太可能在蛋白质折叠过程中发生。Ψ 环首先在 天冬氨酸蛋白酶 家族中被发现。 [25]
卷曲螺旋
Francis Crick 在 1952 年提出了 α 角蛋白卷曲螺旋的可能性,以及确定其结构的数学方法。值得注意的是,这紧随 1951 年 Linus Pauling 及其同事提出的 α 螺旋结构。
卷曲螺旋通常包含一个重复的模式,hxxhcxc,疏水性 (h) 和带电荷 (c) 氨基酸残基,被称为七肽重复。七肽重复中的位置通常标记为 abcdefg,其中 a 和 d 是疏水位置,通常被异亮氨酸、亮氨酸或缬氨酸占据。将具有此重复模式的序列折叠成 α 螺旋二级结构会导致疏水残基呈“条纹”状,以左手方式绕螺旋轻轻盘绕,形成两亲结构。在细胞质充满水的环境中,两种这种螺旋排列的最有利方式是将疏水链包裹在一起,夹在亲水氨基酸之间。因此,疏水表面的埋藏提供了寡聚化的热力学驱动力。卷曲螺旋界面中的堆积非常紧密,在 a 和 d 残基的侧链之间几乎完全存在范德华接触。这种紧密的堆积最初是由 **弗朗西斯·克里克在 1952 年预测**的,被称为“突起进入孔洞”堆积。α 螺旋可以是平行的或反平行的,并且通常采用左手超螺旋。尽管不受青睐,但一些右手卷曲螺旋也在自然界和设计的蛋白质中被观察到。 [26]
| 几何属性 | α-螺旋 | 310 螺旋 | π-螺旋 |
|---|---|---|---|
| 每圈残基数 | 3.6 | 3.0 | 4.4 |
| 每残基平移 | 1.5Å | 2.0Å | 1.1Å |
| 螺旋半径 | 2.3Å | 1.9Å | 2.8Å |
| 螺距 | 5.4Å | 6.0Å | 4.8Å |

蛋白质的三级结构
[edit | edit source]三级结构被认为很大程度上由蛋白质的一级结构决定——它所组成的氨基酸序列。从一级结构预测三级结构的努力通常被称为蛋白质结构预测。然而,蛋白质合成和允许折叠的环境是其最终形状的重要决定因素,通常不会被当前的预测方法直接考虑。
在球状蛋白质中,三级相互作用经常通过疏水氨基酸残基在蛋白质核心中的隔离而稳定,从该核心排除水,并因此在蛋白质的水暴露表面富集带电荷或亲水残基。在没有在细胞质中停留的分泌蛋白中,半胱氨酸残基之间的二硫键有助于维持蛋白质的三级结构。许多在功能和进化上无关的蛋白质中出现各种常见且稳定的三级结构——例如,许多蛋白质的形状像 TIM 桶,以酶磷酸三糖异构酶命名。另一种常见结构是高度稳定的二聚体卷曲螺旋结构,由 2-7 个 α 螺旋组成。
迄今为止已知的蛋白质结构大多数是用 X 射线晶体学实验技术解决的,该技术通常提供高分辨率数据,但没有提供关于蛋白质构象灵活性的时间依赖性信息。解决蛋白质结构的第二种常见方法是使用核磁共振,该方法通常提供较低分辨率的数据,并且仅限于相对较小的蛋白质,但可以提供关于蛋白质在溶液中运动的时间依赖性信息。双偏振干涉法是一种时间分辨分析方法,用于确定表面捕获蛋白质的整体构象和构象变化,为这些高分辨率方法提供补充信息。关于可溶性球状蛋白质的三级结构特征比关于膜蛋白的了解更多,因为后者类别使用这些方法极其难以研究。 [28]
蛋白质的四级结构
[edit | edit source]一些蛋白质实际上是多个 **多肽链** 的集合,在更大集合的背景下被称为蛋白质亚基。除了亚基的三级结构之外,多亚基蛋白质还具有四级结构,即亚基组装成的排列。由具有不同功能的亚基组成的酶有时被称为全酶,其中一些部分被称为调节亚基,而功能核心被称为催化亚基。具有四级结构的蛋白质的例子包括血红蛋白、DNA 聚合酶和离子通道。其他被称为多蛋白复合物的集合也具有四级结构。例如 **核小体和微管**。
四级结构的变化可以通过单个亚基内的构象变化或亚基相对于彼此的重新定位来发生。正是通过这种变化,它构成“多聚”酶中的协同性和变构作用的基础,许多蛋白质经历调节并执行其生理功能。以上定义遵循生物化学的经典方法,该方法是在蛋白质和功能性蛋白质单元之间的区别难以阐明的时候建立的。最近,人们在讨论蛋白质的四级结构时提到蛋白质-蛋白质相互作用,并将所有蛋白质的集合视为蛋白质复合物。 [29]
蛋白质结构测定
[edit | edit source]
蛋白质数据库中大约 90% 的蛋白质结构是通过 X 射线晶体学确定的。这种方法使人们能够测量蛋白质中电子的 3D 密度分布(在晶体状态下),从而推断所有原子的 3D 坐标,以便以一定分辨率确定它们。大约 9% 的已知蛋白质结构是通过核磁共振技术获得的。可以通过圆二色性或双偏振干涉法确定二级结构组成。冷冻电子显微镜最近已成为一种以高分辨率(小于 5 埃或 0.5 纳米)确定蛋白质结构的方法,预计在未来十年将成为高分辨率工作工具的效力将会提高。对于使用非常大的蛋白质复合物(如病毒衣壳蛋白和淀粉样蛋白纤维)进行研究的研究人员来说,这种技术仍然是一项宝贵的资源。
X 射线晶体学
[edit | edit source]生物分子的X射线晶体学随着多萝西·克劳福特·霍奇金的出现而蓬勃发展,她解开了胆固醇(1937年)、维生素B12(1945年)和青霉素(1954年)的结构,并因此于1964年获得了诺贝尔化学奖。1969年,她成功地解开了胰岛素的结构,为此她研究了30多年。[30]
X射线晶体学是一种确定晶体中原子排列的方法,其中一束X射线照射到晶体上,并衍射到许多特定方向。蛋白质的晶体结构(不规则且比胆固醇大数百倍)从1950年代后期开始被解开,首先是抹香鲸肌红蛋白的结构,由马克斯·佩鲁茨和约翰·肯德鲁爵士解开,他们因此于1962年获得了诺贝尔化学奖。[31] 自那次成功以来,已确定了超过61840种蛋白质、核酸和其他生物分子的X射线晶体结构。[32] 相比之下,在分析结构方面,最接近的竞争方法是核磁共振(NMR)波谱法,它已经解析了8759种化学结构。[33] 此外,晶体学可以解决任意大小的分子结构,而溶液态NMR则局限于相对较小的分子(小于70 kDa)。X射线晶体学现在被科学家们常规地用来确定药物如何与蛋白质靶标相互作用,以及哪些改变可以改进药物。[34] 然而,内在膜蛋白的结晶仍然具有挑战性,因为它们需要去垢剂或其他方法才能在分离状态下溶解,而这些去垢剂通常会干扰结晶。这种膜蛋白是基因组的重要组成部分,包括许多具有重要生理意义的蛋白质,例如离子通道和受体。[35][36]
核磁共振波谱法或NMR
[edit | edit source]蛋白质核磁共振波谱法(通常缩写为蛋白质NMR)是结构生物学的一个领域,其中使用NMR波谱法来获取有关蛋白质结构和动力学的的信息。该领域由理查德·R·恩斯特和库尔特·武特里希等人在内开创[1]。蛋白质NMR技术不断地在学术界和生物技术行业中使用和改进。通过NMR波谱法进行结构测定通常包括以下几个阶段,每个阶段都使用一组高度专业化的技术。样品制备、共振分配、约束生成以及结构计算和验证。
如何测序蛋白质?
[edit | edit source]蛋白质测序是一种确定蛋白质氨基酸序列的技术,以及蛋白质采用的构象,以及它与任何非肽分子复合的程度。发现生物体中蛋白质的结构和功能是理解细胞过程的重要工具,并使针对特定代谢途径的药物更容易发明。蛋白质测序的两种主要直接方法是**质谱法**和**艾德曼降解反应**。如果已知编码蛋白质的DNA或mRNA序列,也可以从该序列生成氨基酸序列。然而,还有许多其他反应可以用来获得关于蛋白质序列的更多有限信息,这些信息可以用作上述测序方法的预备步骤,或用来克服其中特定方面的不足。[37]
艾德曼降解
[edit | edit source]
艾德曼降解是蛋白质测序中非常重要的反应,因为它可以确定蛋白质的有序氨基酸组成。自动艾德曼测序仪现在被广泛使用,能够对长度约为50个氨基酸的肽进行测序。通过艾德曼降解对蛋白质进行测序的反应方案如下 - 其中一些步骤将在后面详细说明。用氧化剂如**甲酸**或还原剂如**2-巯基乙醇**破坏蛋白质中的任何二硫键。可能需要使用如碘乙酸的保护基团来防止键重新形成。如果存在多个蛋白复合物链,则分离并纯化单个链。
确定每条链的氨基酸组成。
确定每条链的末端氨基酸。
将每条链断裂成长度小于50个氨基酸的片段。
分离并纯化片段。
确定每个片段的序列。
以不同的断裂模式重复。
构建整个蛋白质的序列。
消化成肽片段 长度超过约50-70个氨基酸的肽不能通过艾德曼降解可靠地测序。因此,需要将长蛋白链分解成小的片段,然后可以单独测序。消化是通过内肽酶(如胰蛋白酶或胃蛋白酶)或化学试剂(如氰化溴)进行的。不同的酶产生不同的断裂模式,并且片段之间的重叠可以用来构建一个完整的序列。
苯异硫氰酸酯在弱碱性条件下与未带电的末端氨基反应,形成循环的苯硫代氨基甲酰衍生物。然后,在酸性条件下,末端氨基酸的这种衍生物被裂解为噻唑啉酮衍生物。噻唑啉酮氨基酸然后被选择性地提取到有机溶剂中,并用酸处理以形成更稳定的苯硫代腙(PTH)-氨基酸衍生物,该衍生物可以使用色谱法或电泳法进行鉴定。然后可以重复此过程以鉴定下一个氨基酸。这项技术的一个主要缺点是,以这种方式测序的肽不能超过50到60个残基(实际上,小于30个)。肽的长度受循环衍生化并非总是完成的限制。衍生化问题可以通过在进行反应之前将大肽裂解成小肽来解决。它能够准确地测序高达30个氨基酸,现代机器能够在每个氨基酸上达到99%以上的效率。艾德曼降解的一个优点是,它只需要10-100皮摩尔的肽进行测序。艾德曼降解反应已经自动化,以加快这一过程。[38][39]
N末端氨基酸分析
[edit | edit source]确定哪个氨基酸构成肽链的N端有两个原因:有助于将单个肽片段的序列排列成完整的链,以及因为第一轮Edman降解通常会被杂质污染,因此无法准确确定N端氨基酸。以下是N端氨基酸分析的通用方法:用能选择性标记末端氨基酸的试剂与肽反应。水解蛋白质。通过色谱法和与标准品的比较确定氨基酸。有很多不同的试剂可用于标记末端氨基酸。它们都与胺基反应,因此也会与赖氨酸等氨基酸侧链中的胺基结合 - 因此,在解释色谱图时必须小心,以确保选择了正确的点。两种比较常用的试剂是桑格试剂(1-氟-2,4-二硝基苯)和丹磺酰衍生物,如丹磺酰氯。用于Edman降解的试剂苯异硫氰酸酯也可以使用。这里与氨基酸组成测定中提出的问题相同,唯一的例外是无需染色,因为试剂会产生有色衍生物,并且只需要定性分析,因此氨基酸无需从色谱柱中洗脱,只需与标准品进行比较即可。另一个需要考虑的因素是,由于任何胺基都会与标记试剂反应,因此不能使用离子交换色谱,而应使用薄层色谱或高效液相色谱。 [40]
C端氨基酸分析
[edit | edit source]用于C端氨基酸分析的方法数量远远少于可用于N端分析的方法数量。最常用的方法是在蛋白质溶液中添加羧肽酶,定期取样,并通过分析氨基酸浓度随时间的变化图来确定末端氨基酸。
质谱法
[edit | edit source]当今的研究人员正在使用质谱法作为表征蛋白质的重要工具。蛋白质质谱法是指将质谱法应用于蛋白质的研究。两种主要的完整蛋白质电离方法是电喷雾电离(ESI)和基质辅助激光解吸/电离(MALDI)。为了符合现有质谱仪的性能和质量范围,使用两种方法来表征蛋白质。首先,通过上述两种技术之一电离完整的蛋白质,然后将其导入质量分析仪。这种方法被称为蛋白质分析的“自上而下”策略。其次,使用胰蛋白酶等蛋白酶将蛋白质酶解成更小的肽段。随后,这些肽段被导入质谱仪,并通过肽质量指纹图谱或串联质谱法进行鉴定。因此,这种后一种方法(也称为“自下而上”蛋白质组学)使用肽水平上的鉴定来推断蛋白质的存在。
完整蛋白质质量分析主要使用飞行时间(TOF)质谱仪或傅里叶变换离子回旋共振(FT-ICR)。这两种类型的仪器在这里更受欢迎,因为它们具有较宽的质量范围,并且在FT-ICR的情况下,具有较高的质量精度。蛋白水解肽的质量分析是一种更流行的蛋白质表征方法,因为可以更便宜的仪器设计用于表征。此外,一旦完整蛋白质被消化成更小的肽片段,样品制备就变得更容易。用于肽质量分析最广泛使用的仪器是MALDI飞行时间仪器,因为它们允许快速获取肽质量指纹图谱(PMF)(大约10秒内可以分析一个PMF)。多级四极杆飞行时间和四极杆离子阱也在这项应用中得到应用。
蛋白质类型
[edit | edit source]结合蛋白
[edit | edit source]结合蛋白是指与通过共价键或弱相互作用连接的其他化学基团相互作用的蛋白质。许多蛋白质只包含氨基酸,没有其他化学基团,被称为简单蛋白质。然而,其他类型的蛋白质在水解时,除了氨基酸外,还会产生其他化学成分,这些蛋白质被称为结合蛋白。结合蛋白的非氨基部分通常被称为其辅基。大多数辅基是由维生素形成的。结合蛋白根据其辅基的化学性质进行分类。一些结合蛋白的例子是
脂蛋白
[edit | edit source]脂蛋白是包含蛋白质和脂质的生化组合体,脂质与蛋白质结合在一起。许多酶、转运蛋白、结构蛋白、抗原、粘附素和毒素都是脂蛋白。例如,高密度 (HDL) 和低密度 (LDL) 脂蛋白能够将脂肪带到血液中,线粒体和叶绿体的跨膜蛋白以及细菌脂蛋白。
糖蛋白
[edit | edit source]糖蛋白是包含寡糖链(糖基)的蛋白质,寡糖链通过共价键连接到多肽侧链。碳水化合物在蛋白质的共翻译或翻译后修饰中连接到蛋白质。这个过程被称为糖基化。在具有胞外延伸段的蛋白质中,胞外延伸段通常被糖基化。糖蛋白通常是重要的整合膜蛋白,在细胞间相互作用中发挥作用。糖蛋白也存在于胞质溶胶中,但它们的功能以及在这种隔室中产生这些修饰的途径尚未完全了解。糖蛋白通常是结合蛋白中最大且最丰富的蛋白质群。它们从构成糖萼的细胞表面膜糖蛋白,到白细胞产生的重要抗体。
磷蛋白
[edit | edit source]磷蛋白是指与含有磷酸的物质化学结合的蛋白质(有关更多信息,请参阅磷酸化)。包括 Fc 受体、Ulks、钙调神经磷酸酶、K 芯片和尿皮质素的类别。
金属蛋白
[edit | edit source]包含金属离子作为辅因子的蛋白质被称为金属蛋白。金属蛋白在细胞中具有许多不同的功能,例如酶、转运和储存蛋白以及信号转导蛋白。事实上,大约四分之一到三分之一的所有蛋白质都需要金属才能发挥其功能。金属离子通常由属于多肽链中氨基酸的氮、氧或硫原子以及掺入蛋白质中的大环配体进行配位。金属离子的存在使金属酶能够执行氧化还原反应等功能,而这些功能不能轻易由氨基酸中发现的有限的功能基团集来执行。

| 金属离子 | 含有该离子的酶的例子 |
|---|---|
| 镁 | 葡萄糖-6-磷酸酶 己糖激酶 DNA 聚合酶 |
| 钒 | 钒结合蛋白 |
| 锰 | 精氨酸酶 |
| 铁 | 过氧化氢酶 氢化酶 IRE-BP 顺乌头酸酶 |
| 镍[41] | 脲酶 氢化酶 |
| 铜 | 细胞色素氧化酶 漆酶 |
| 锌 | 醇脱氢酶 羧肽酶 氨基肽酶 β淀粉样蛋白 |
| 钼 | 硝酸还原酶 |
| 硒 | 谷胱甘肽过氧化物酶 |
| 各种 | 金属硫蛋白 磷酸酶 |
血红蛋白
[edit | edit source]血红蛋白(或血红素蛋白、血红素蛋白),或血红素蛋白,是一种含血红素辅基的金属蛋白,共价或非共价结合到蛋白质本身。血红素中的铁能够进行氧化和还原(通常为 +2 和 +3,尽管在过氧化物酶中已知稳定的 Fe+4 甚至 Fe+5 物种)。血红蛋白可能起源于一种原始策略,允许将血红素的原卟啉 IX 环中所含的铁 (Fe) 原子结合到蛋白质中。这种策略在进化过程中一直保持着,因为它使血红蛋白对能够结合二价铁 (Fe) 的分子做出反应。这些分子包括,但可能不限于,气体分子,例如氧气 (O2)、一氧化氮 (NO)、一氧化碳 (CO) 和硫化氢 (H2S)。一旦与血红蛋白的辅基血红素基团结合,这些气体分子可以以一种据说可以提供信号转导的方式调节这些血红蛋白的活性/功能。因此,当在生物系统(细胞)中产生时,这些气体分子被称为气体递质。血红蛋白含有含铁的辅基,即血红素。正是血红素基团通过氧分子与血红素基团中发现的铁离子 (Fe2+) 的结合,将氧分子输送到全身。[42]
血红蛋白 血红蛋白(也拼写为血红蛋白,缩写为 Hb 或 Hgb)是所有脊椎动物[1](除鱼类家族 Channichthyidae 外)红血球和一些无脊椎动物组织中的一种含铁的氧转运金属蛋白。血液中的血红蛋白将氧气从肺部或鳃部输送到身体其他部位(即组织),在那里释放氧气供细胞使用,并收集二氧化碳将其带回肺部。在哺乳动物中,这种蛋白质约占红血球干物质的 97%,约占总含量的 35%(包括水)[需要引用]。血红蛋白的氧气结合能力为每克血红蛋白 1.34 ml O2,这使血液的总氧气容量增加了 70 倍。血红蛋白参与其他气体的运输:它以氨基甲酸血红蛋白的形式携带一些人体呼吸二氧化碳(约占总量的 10%),其中 CO2 与珠蛋白结合。该分子还携带重要的调节分子一氧化氮,其结合到珠蛋白蛋白硫醇基团,在氧气释放的同时释放它。血红蛋白也存在于红血球及其祖系之外。其他含有血红蛋白的细胞包括黑质中的 A9 多巴胺能神经元、巨噬细胞、肺泡细胞和肾脏的系膜细胞。在这些组织中,血红蛋白具有非氧气转运功能,作为抗氧化剂和铁代谢调节剂。血红蛋白和血红蛋白样分子也存在于许多无脊椎动物、真菌和植物中。在这些生物体中,血红蛋白可以携带氧气,或者可以作为二氧化碳、一氧化氮、硫化氢和硫化物的运输和调节剂。这种分子的一个变体,称为豆血红蛋白,用于清除氧气,以防止其毒害厌氧系统,如豆科植物的固氮结瘤。光敏色素,
细胞色素
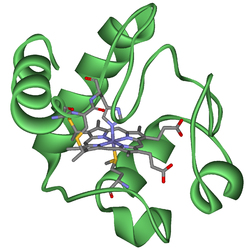
一般来说,细胞色素是膜结合血红蛋白,含有血红素基团并进行电子传递。它们以单体蛋白(例如,细胞色素 c)或更大酶复合物的亚基形式存在,这些复合物催化氧化还原反应。它们存在于真核生物的线粒体内膜和内质网中,存在于植物的叶绿体中,存在于光合微生物和细菌中。
| 细胞色素 | 组合 |
| *a* 和 *a3* | 细胞色素 c 氧化酶(“复合物 IV”),电子通过可溶性细胞色素 c传递到复合物(因此得名) |
| *b* 和 c1 | 辅酶 Q - 细胞色素 c 还原酶(“复合物 III”) |
| *b6* 和 f | 质体醌醇 - 质体蓝蛋白还原酶 |

| 类型 | 辅基 |
| 细胞色素 a | 血红素 a |
| 细胞色素 b | 血红素 b |
| 细胞色素 d | 四吡咯螯合物的铁 |
视蛋白
[edit | edit source]视蛋白是一组对光敏感的 35-55 kDa 膜结合 G 蛋白偶联受体,属于视黄醛蛋白家族,存在于视网膜的光感受器细胞中。五组经典的视蛋白参与视觉,介导光子转化为电化学信号,这是视觉转导级联反应的第一步。另一种存在于哺乳动物视网膜中的视蛋白,黑视蛋白,参与昼夜节律和瞳孔反射,但不参与成像。
黄素蛋白
[edit | edit source]黄素蛋白是含有核黄素的核酸衍生物的蛋白质:黄素腺嘌呤二核苷酸 (FAD) 或黄素单核苷酸 (FMN)。黄素蛋白参与各种生物过程,包括但不限于生物发光、去除导致氧化应激的自由基、光合作用、DNA 修复和细胞凋亡。黄素辅因子的光谱特性使其成为活性位点内发生变化的天然报告器;这使得黄素蛋白成为研究最多的酶家族之一。
简单蛋白质
[edit | edit source]水解后仅产生氨基酸的蛋白质称为简单蛋白质。
白蛋白
[edit | edit source]白蛋白(拉丁语:albus,白色)一般指任何水溶性蛋白质,在浓盐溶液中中等溶解,并发生热变性。它们通常存在于血浆中,并且在其他血浆蛋白中独一无二,因为它们没有糖基化。含有白蛋白的物质,例如蛋清,被称为白蛋白类。
球蛋白
[edit | edit source]球蛋白是三种血清蛋白类型之一,另外两种是白蛋白和纤维蛋白原。一些球蛋白在肝脏中产生,而另一些则由免疫系统产生。球蛋白一词包含一组异质蛋白,其典型的分子量较高,溶解度和电泳迁移率均低于白蛋白。
组蛋白
[edit | edit source]在生物学中,组蛋白是存在于真核细胞核中的高度碱性蛋白质,它们将 DNA 包装并排列成称为核小体的结构单元。它们是染色质的主要蛋白质成分,充当 DNA 缠绕的线轴,并在基因调控中发挥作用。
衍生蛋白
[edit | edit source]胨
[edit | edit source]胨是从动物乳汁或肉类中通过蛋白水解消化而得的。除了含有小肽外,所得的喷雾干燥材料还包括脂肪、金属、盐、维生素和许多其他生物化合物。胨用于培养细菌和真菌的营养培养基
蛋白酶
[edit | edit source]蛋白酶自然存在于所有生物体中。这些酶参与许多生理反应,从简单的食物蛋白质消化到高度调节的级联反应(例如,血液凝固级联反应、补体系统、细胞凋亡途径和无脊椎动物前苯酚氧化酶激活级联反应)。蛋白酶可以根据蛋白质的氨基酸序列,要么断裂特定的肽键(有限蛋白水解),要么将完整的肽分解为氨基酸(无限蛋白水解)。该活性可以是破坏性变化,消除蛋白质的功能或将其消化成主要成分;它可以是功能的激活,或者可以是信号通路中的信号。
蛋白质数据库或 PDB
[edit | edit source]蛋白质数据库 (PDB) 的诞生源于两股力量的汇聚,如同燃料和火焰:1) 一种规模虽小但不断增长的蛋白质结构数据集,这些结构是通过 X 射线衍射法确定的;2) 1968 年问世的分子图形显示系统,布鲁克海文光栅显示器 (BRAD),用于三维观察这些结构。1969 年,在布鲁克海文国家实验室的沃尔特·汉密尔顿博士的赞助下,埃德加·迈耶博士 (德克萨斯 A&M 大学) 开始编写软件,以通用格式存储原子坐标文件,以便进行几何和图形评估。到 1971 年,程序 SEARCH 已远程运行,用于提取和检查结构数据,并因此成为网络启动的关键因素,标志着 PDB 的功能性起点。
汉密尔顿博士于 1973 年逝世,托马斯·科兹尔博士接手 PDB 的领导工作,并在随后的 20 年间一直担任此职。1994 年 1 月,以色列魏茨曼科学研究所的约尔·萨斯曼博士被任命为 PDB 主任。1998 年 10 月,[43] PDB 转移至结构生物信息学研究合作中心 (RCSB);转移于 1999 年 6 月完成。新任主任是来自罗格斯大学 (RCSB 成员机构之一) 的海伦·M·伯曼博士。[44] 2003 年,随着 wwPDB 的成立,PDB 成为一个国际组织。创始成员包括 PDBe(欧洲)、RCSB(美国)和 PDBj(日本)。BMRB 于 2006 年加入。wwPDB 的四个成员都可以作为 PDB 数据的沉积、数据处理和分发中心。数据处理是指 wwPDB 工作人员审查和注释每个提交的条目。然后自动检查数据的合理性(该验证软件的源代码已免费公开提供)。
蛋白质数据库 (PDB)[45] 是大型生物分子(如蛋白质和核酸)的三维结构数据的存储库。(另请参阅晶体学数据库)。这些数据通常通过 X 射线晶体学或核磁共振波谱获得,由来自世界各地的生物学家和生物化学家提交,并通过其成员组织(PDBe、PDBj 和 RCSB)的网站在互联网上免费获取。PDB 由一个名为全球蛋白质数据库 (wwPDB) 的组织监督。
胰岛素
[edit | edit source]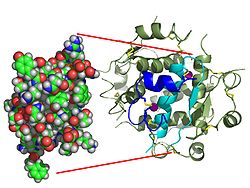

在脊椎动物体内,胰岛素的氨基酸序列非常保守。牛胰岛素与人胰岛素仅在三个氨基酸残基上有所不同,而猪胰岛素与人胰岛素仅在单个氨基酸残基上有所不同。甚至一些鱼类的胰岛素也与人胰岛素足够相似,可以在临床上对人有效。一些无脊椎动物的胰岛素在序列上也与人胰岛素非常相似,并具有相似的生理作用。在不同物种的胰岛素序列中观察到的高度同源性表明,它在动物进化史上大部分时间里都得到了保守。然而,胰岛素原的 C 肽在不同物种间差异更大;它也是一种激素,但是一种次要激素。
胰岛素在体内以六聚体(由六个胰岛素分子组成)的形式生成并储存,而活性形式是单体。六聚体是长期稳定的非活性形式,可以保护高反应性的胰岛素,并使其随时可用。六聚体与单体的转化是胰岛素注射制剂的核心方面之一。六聚体比单体稳定得多,这对实际应用来说是可取的,但单体是一种反应速度更快的药物,因为扩散速率与粒径成反比。反应速度快的药物意味着胰岛素注射不必在饭前数小时进行,从而使糖尿病患者在日常生活中更加灵活。胰岛素可以聚集并形成纤维状交错的 β 片。这会导致注射淀粉样变,并会阻止胰岛素长时间储存。[46]
1869 年,柏林的一名医学生保罗·朗格汉斯在显微镜下研究胰腺结构时,发现了一些以前未发现的组织团块散布在胰腺的大部分区域。这些“小堆细胞”的功能尚不清楚,后来被命名为朗格汉斯岛,但爱德华·拉盖斯随后提出,它们可能产生分泌物,在消化过程中发挥调节作用。保罗·朗格汉斯的儿子阿奇博尔德也帮助理解了这种调节作用。“胰岛素”一词源于拉丁语中的“insula”,意为小岛或岛屿。1889 年,波兰裔德国医生奥斯卡·明可夫斯基与约瑟夫·冯·梅林合作,从一只健康的狗身上切除了胰腺,以测试其在消化中的假定作用。在狗的胰腺被切除后的几天里,明可夫斯基的动物管理员注意到一群苍蝇在狗的尿液上觅食。他们对尿液进行检测后发现,狗的尿液中含有糖,从而首次确定了胰腺与糖尿病之间的关系。1901 年,尤金·奥皮取得了又一个重大进展,他明确地建立了朗格汉斯岛与糖尿病之间的联系:糖尿病……是由朗格汉斯岛的破坏引起的,只有当这些岛体部分或全部被破坏时才会发生。在他进行这项工作之前,胰腺与糖尿病之间的联系是明确的,但朗格汉斯岛的确切作用尚不清楚。
1923 年,诺贝尔奖委员会将胰岛素的实际提取归功于多伦多大学的一个团队,并将诺贝尔奖颁给了两位男性;弗雷德里克斯·班坦和 J.J.R. 麦克莱昂。他们在 1923 年因发现胰岛素而获得诺贝尔生理学或医学奖。班坦对贝斯特没有被提及感到不满,他与贝斯特分享了他的奖项,而麦克莱昂也立即与詹姆斯·科利普分享了他的奖项。胰岛素的专利以半美元的价格卖给了多伦多大学。
胰岛素的一级结构由英国分子生物学家弗雷德里克·桑格确定。它是第一个被确定序列的蛋白质。他因这项工作获得了 1958 年诺贝尔化学奖。1969 年,经过数十年的努力,多萝西·克劳福特·霍奇金通过 X 射线衍射研究确定了该分子的空间构象,即所谓的三级结构。她因发展晶体学获得了 1964 年诺贝尔化学奖。罗莎琳·萨斯曼·亚洛因开发了用于胰岛素的放射免疫分析法,获得了 1977 年诺贝尔医学奖。[47]
参考文献
[edit | edit source]- ↑ http://en.wikipedia.org/w/index.php?title=Protein&oldid=425576197
- ↑ http://en.wikipedia.org/w/index.php?title=Proteinogenic_amino_acid&oldid=420804587
- ↑ http://en.wikipedia.org/w/index.php?title=Amino_acid&oldid=425389108
- ↑ http://en.wikipedia.org/w/index.php?title=Amino_acid&oldid=425389108
- ↑ 光亮亮氨酸和光亮亮氨酸允许在活细胞中识别蛋白质-蛋白质相互作用。自然方法:4,261-7,2005
- ↑ http://en.wikipedia.org/w/index.php?title=Peptide_bond&oldid=417601014
- ↑ Basler B, Schuster O, Bach T (2005). “通过四氢呋喃酸酰胺的分子内 [2 + 2] 光环加成反应和随后的内酯环开环制备构象受限的 β-氨基酸衍生物”. J. Org. Chem. 70 (24): 9798–808. doi:10.1021/jo0515226. PMID 16292808.
{{cite journal}}: 未知参数|month=被忽略 (帮助)CS1 maint: 多个名称: 作者列表 (链接) - ↑ Murray JK, Farooqi B, Sadowsky JD; 等 (2005). “使用微波照射高效合成 β-肽组合库”. J. Am. Chem. Soc. 127 (38): 13271–80. doi:10.1021/ja052733v. PMID 16173757.
{{cite journal}}: 在|author=中显式使用“等” (帮助); 未知参数|month=被忽略 (帮助)CS1 maint: 多个名称: 作者列表 (链接) - ↑ Seebach D, Matthews JL (1997). “β-肽:每一次都有惊喜”. Chem. Commun. (21): 2015–22. doi:10.1039/a704933a.
- ↑ http://en.wikipedia.org/w/index.php?title=Enzyme&oldid=424282616
- ↑ http://en.wikipedia.org/w/index.php?title=Enzyme&oldid=424282616
- ↑ Moss, G.P. "酶的命名法委员会建议". 酶的命名和分类的国际生物化学和分子生物学联盟. 检索于 2006-03-14.
- ↑ http://en.wikipedia.org/w/index.php?title=Enzyme&oldid=424282616
- ↑ Lovell SC; 等 (2003). “通过 Cα 几何形状进行结构验证:φ,ψ 和 Cβ 偏差”. Proteins. 50 (3): 437–450. doi:10.1002/prot.10286. PMID 12557186.
{{cite journal}}: 在|author=中显式使用“等” (帮助) - ↑ http://en.wikipedia.org/w/index.php?title=Protein_primary_structure&oldid=415921787
- ↑ Pauling L, Corey RB, Branson HR (1951). "蛋白质的结构:多肽链的两种氢键螺旋构型". Proc Natl Acad Sci USA. 37 (4): 205–211. doi:10.1073/pnas.37.4.205. PMC 1063337. PMID 14816373.
{{cite journal}}: CS1 maint: 多个名称: 作者列表 (链接) - ↑ Chiang YS, Gelfand TI, Kister AE, Gelfand IM (2007). “三明治状蛋白质超二级结构的新分类揭示了链组装的严格模式”. Proteins. 68 (4): 915–921. doi:10.1002/prot.21473. PMID 17557333.
{{cite journal}}: CS1 maint: 多个名称: 作者列表 (链接) - ↑ RAMACHANDRAN GN, RAMAKRISHNAN C, SASISEKHARAN V (1963 年 7 月). “多肽链构型的立体化学”. J. Mol. Biol. 7: 95–9
- ↑ http://en.wikipedia.org/w/index.php?title=Alpha_helix&oldid=423162580
- ↑ 蛋白质三级结构和折叠:第 4.3.2.1 节. 来自 蛋白质结构、比较蛋白质建模和可视化的原理
- ↑ Hutchinson EG, Thornton JM (1993 年 4 月). “希腊键基序:提取、分类和分析”. Protein Eng. 6 (3): 233–45. doi:10.1093/protein/6.3.233. PMID 8506258.
- ↑ SCOP:折叠:WW 结构域类似
- ↑ PPS '96 - 超二级结构
- ↑ Hutchinson, E. (1996). "PROMOTIF—A program to identify and analyze structural motifs in proteins". 蛋白质科学. 5 (2): 212–220. doi:10.1002/pro.5560050204. PMC 2143354. PMID 8745398.
{{cite journal}}: Cite has empty unknown parameter:|month=(help); Unknown parameter|coauthors=ignored (|author=suggested) (help) - ↑ Hutchinson EG, Thornton JM (1990). "HERA--a program to draw schematic diagrams of protein secondary structures". 蛋白质. 8 (3): 203–12. doi:10.1002/prot.340080303. PMID 2281084.
- ↑ http://en.wikipedia.org/w/index.php?title=Coiled_coil&oldid=427735447
- ↑ Steven Bottomley (2004). "交互式蛋白质结构教程". Retrieved January 9, 2011.
{{cite web}}: Check|authorlink=value (help); External link in|authorlink= - ↑ http://en.wikipedia.org/w/index.php?title=Protein_tertiary_structure&oldid=422486540
- ↑ http://en.wikipedia.org/wiki/Protein_quaternary_structure
- ↑ Crowfoot Hodgkin D (1935). "胰岛素的X射线单晶照片". 自然. 135: 591. doi:10.1038/135591a0.
- ↑ Kendrew J. C.; et al. (1958-03-08). "通过X射线分析获得的肌红蛋白分子三维模型". 自然. 181 (4610): 662. doi:10.1038/181662a0. PMID 13517261.
{{cite journal}}: Explicit use of et al. in:|author=(help) - ↑ "PDB 中的条目表,按实验方法排列".
- ↑ "PDB 统计". RCSB 蛋白质数据库. Retrieved 2010-02-09.
- ↑ Scapin G (2006). "结构生物学与药物发现". Curr. Pharm. Des. 12 (17): 2087. doi:10.2174/138161206777585201. PMID 16796557.
- ↑ Lundstrom K (2006). "膜蛋白的结构基因组学". 细胞与分子生命科学. 63 (22): 2597. doi:10.1007/s00018-006-6252-y. PMID 17013556.
- ↑ Lundstrom K (2004). "膜蛋白的结构基因组学:简要综述". 组合化学与高通量筛选. 7 (5): 431. PMID 15320710.
- ↑ http://en.wikipedia.org/w/index.php?title=Protein_sequencing&oldid=413170994
- ↑ Niall HD (1973). "自动艾德曼降解:蛋白质测序仪". Meth. Enzymol. 27: 942–1010. doi:10.1016/S0076-6879(73)27039-8. PMID 4773306.
- ↑ http://en.wikipedia.org/w/index.php?title=Protein_sequencing&oldid=413170994
- ↑ http://en.wikipedia.org/w/index.php?title=Protein_sequencing&oldid=413170994
- ↑ Astrid Sigel, Helmut Sigel 和 Roland K.O. Sigel,编辑 (2008). 镍及其在自然界中令人惊讶的影响. 生命科学中的金属离子. 卷 2. 威利. ISBN 978-0-470-01671-8.
- ↑ http://en.wikipedia.org/w/index.php?title=Hemeprotein&oldid=410476687
- ↑ Berman, H. M. (2000). "蛋白质数据库". Nucleic Acids Res. 28 (1): 235–242. doi:10.1093/nar/28.1.235. PMC 102472. PMID 10592235.
{{cite journal}}: 未知参数|coauthors=被忽略 (|author=建议) (帮助); 未知参数|month=被忽略 (帮助) - ↑ "RCSB PDB 新闻通讯存档". RCSB 蛋白质数据库.
- ↑ wwPDB, Consortium (2019). "蛋白质数据库:全球唯一的 3D 大分子结构数据存档". Nucleic Acids Res. 47 (D1): 520–528. doi:10.1093/nar/gky949. PMC 6324056. PMID 30357364.
{{cite journal}}: 未知参数|month=被忽略 (帮助) - ↑ http://en.wikipedia.org/w/index.php?title=Insulin&oldid=425481933
- ↑ http://en.wikipedia.org/w/index.php?title=Insulin&oldid=425481933