MINC/工具/mni autoreg
在诊断和研究应用中,当不同的数据集可以通过对等解剖平面的视觉检查进行比较时,人类大脑磁共振(MR)图像的解释会更容易。使用预定义的图谱模板进行定量分析通常需要先将图谱和图像平面对齐。不幸的是,在不同的扫描会话中获得的轴向平面在它们的相对位置和方向上往往不同,并且这些切片与图谱中的切片不共面。已经开发出一种完全自动的方法,基于多尺度三维(3D)互相关,将给定的体积数据集配准到与 Talairach 立体定向坐标系对齐的平均 MRI 大脑(n > 300)。一旦数据集通过算法恢复的变换进行重采样,图谱切片就可以直接叠加在重采样体积的相应切片上(见下文)。使用这种标准化空间还允许直接比较,逐个体素地比较,两个或多个被带入立体定向空间的数据集。
一个 Perl 脚本 (mritotal) 实施了多解析度拟合策略,该策略已用于将 500 多个大脑映射到蒙特利尔神经病学研究所的立体定向空间。此过程的核心是 minctracc,该程序自动找到将一个体积数据集(存储在 MINC 格式中,见下文)映射到另一个数据集的最佳线性变换。该程序使用对用户可选参数数量的优化来识别最佳(根据用户选择的目标函数)变换,将第一个数据集的体素值映射到第二个数据集。
配准的主要脚本是
- mritotal - 调用 mincblur 和 minctracc 来实现立体定向配准的脚本
- mritoself - 脑内配准
其他安装的脚本或程序是
- autocrop - 用于提取和操作 MINC 文件的边界
- make_model - 生成 `mritotal` 所需的适当模糊和子采样图像
- make_phantom - 生成椭球体或矩形幻影图像
- mincbbox - 提取图像的边界框
- mincblur - 用于使用高斯模糊核进行体积卷积的程序
- mincchamfer - 计算图像的 Chamfer 距离变换
- minctracc - 用于估计配准所需变换的程序
- xfmtool - 对变换 (.xfm) 文件执行各种操作
其中一些附带手册页,并且所有都响应 `-help`。
mritotal 需要两个外部文件才能运行。它们是配置文件 (mritotal.cfg) 和协议文件(默认值为 mritotal.default.cfg,但您可以使用 -protocol 选项指定其他文件)。如果您遵循以上所有说明,则这些文件将被正确自定义并安装到您的站点。
但是,如果出现问题,您应该知道以下内容
- 安装配置文件和协议文件(s) 的目录默认设置为 /usr/local/bic/etc/mni_autoreg。
- 模型的位置在 mritotal.cfg 中给出(或在 mritotal 中使用 -modeldir 选项给出)。
- 如果您发现有必要为您的站点数据创建新的协议(例如,如果您的 MRI 数据始终涵盖比大脑更多的内容,但默认协议使用的启发式方法不起作用 - 有关更多信息,请参阅 mritotal 手册页),那么您应该复制并更改默认协议文件。例如,您可能想将协议命名为“mysite” - 在这种情况下,将 mritotal.default.cfg 复制到 mritotal.mysite.cfg,并确保编辑 mritotal.cfg 以使它默认使用新的协议。也就是说,确保它具有“-protocol mysite”而不是“-protocol default”。
为了更好地理解 mni_autoreg 或线性配准是如何工作的,这里有一个简单的例子。假设我们有一张个人的图像,并且希望将其与模型对齐。这两张图像显示在下面,左边是个人图像 (indiv.mnc),右边是模型 (model.mnc)。请注意,这两个文件具有不同的采样和体素大小,如白色线条所示
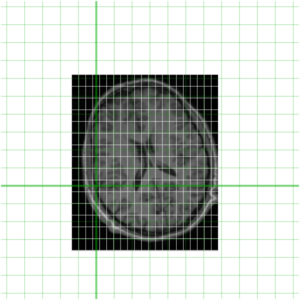
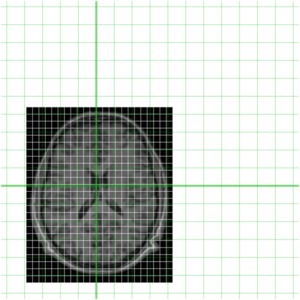
当然,作为 MINC 文件,这两个文件都代表了世界空间的采样(由起点、步长和方向余弦定义 - 使用 mincinfo 查看)。为了更好地理解这一点,我们的两个文件再次显示在下面,它们的相对位置在世界空间中用绿色显示。
为了了解它们目前是如何重叠的,我在下面将它们放在一起
很明显,它们没有相互对齐或“配准”,因此我们必须使用 minctracc 或 mritoself 等工具来对齐它们
为此,我们可以使用这样的命令
minctracc -lsq9 indiv.mnc model.mnc align.xfm
这里的 -lsq9 参数指示 minctracc 恢复 3 个旋转、3 个平移和 3 个缩放。请注意,此步骤的输出不是对齐的图像,而是输出(线性)变换文件,其中包含一个 4x4 变换矩阵。它由 3x3 仿射变换加上 3 个平移组成(如果需要的话)。下面是其中一个文件的示例
MNI Transform File Transform_Type = Linear; Linear_Transform = 0.995668319594959 -0.30943698305448 0.0180935790540566 8.9918311534818 0.320977051206147 0.94080272211886 0.00947446085537751 -39.3894632128575 -0.0222220364958048 -0.00403786869719625 1.15379667282104 -33.4353790283203;
MNI 变换文件有一个隐式的第 4 行,由 0,0,0,1 组成,但此数据未存储在文件中。为了了解此矩阵的分解是什么样的,我们可以使用 xfm2param
xfm2param align.xfm
这将生成如下输出
after parameter extraction -center 0.00000 0.00000 0.00000 -translation 8.99183 -39.38946 -33.43538 -rotation -0.20051 1.10337 17.25793 -scale 1.04280 0.99372 1.15402 -shear 0.02764 -0.00000 -0.00000
因此,现在我们有了变换,我们需要对原始输入数据进行重采样。为此,我们使用 mincresample,但是我们有几个关于输出文件采样的选择。第一个也是最常用的方法是这样的
mincresample -like model.mnc -transformation align.xfm source.mnc resampled.mnc
请注意,输出文件现在具有与原始模型相同的采样。
下一个选项是这样的
mincresample -tfm_input_sampling -transformation align.xfm source.mnc resampled.mnc
这指示 mincresample 通过输入变换重采样文件的原始采样,请注意,这将导致一个带有方向余弦的文件。
最后一个选项是使用个人的原始采样,如下所示
mincresample -use_input_sampling -transformation align.xfm source.mnc resampled.mnc
使用这种方法将保留原始采样,但也会保留文件的原始位置,这意味着您可能会或可能不会保留文件的完全“覆盖”。
因此,-like 选项是最常用的方法,因为它允许对生成的重采样文件使用基于体素的工具(如 minccalc 和 mincmath)进行进一步处理
版权所有 (C) 1993-2010 Louis Collins 和 Greg Ward,麦吉尔大学蒙特利尔神经病学研究所麦康奈尔脑成像中心。特此授予您在任何目的和无偿情况下使用、复制、修改和分发此软件及其文档的权限,但前提是在所有副本中保留上述版权声明。作者和麦吉尔大学对该软件的适用性不作任何陈述。它按“原样”提供,不提供明示或暗示的保证。对于因使用或误用此软件包而导致的任何数据丢失、设备损坏、财产损失或对受试者或患者的伤害,作者概不负责。所有使用 MNI_AutoReg 配准的结果都应该始终在所有三个空间维度上通过视觉检查仔细检查。MNI_AutoReg 配准决不能用于可能导致患者或受试者受伤或导致误诊的环境中。此包仅供研究使用。
我们不保证此包没有概念或编程错误。提供此包给您并不意味着或保证提供超出本文档中提供的任何额外支持。同样,它也不保证您会收到有关发现的任何编程错误的通知,或会收到软件的未来更新版本。作者不对因您使用蒙特利尔神经病学研究所自动线性配准包而造成的直接或间接损失承担任何责任。
我们希望至少引用以下文章来描述在使用 MNI_AutoReg 包分析数据的任何出版物中使用的配准方法
DL Collins,P Neelin,TM Peters 和 AC Evans,自动 3D 跨主体配准 MRI 体积数据到标准化 Talairach 空间,计算机辅助断层扫描杂志,18(2) p192-205, 1994
D. L. Collins, P. Neelin, T. M. Peters 和 A. C. Evans, ``自动 3D 跨主体配准 MRI 体积数据到标准化 Talairach 空间, 计算机辅助断层扫描杂志,18(2) pp192-205, 1994.