结构生物化学/基因表达
基因表达是将基因信息用于合成功能性基因产物的过程。这些产物通常是蛋白质,但在非蛋白质编码基因(如 rRNA 基因或 tRNA 基因)中,产物是功能性 RNA。基因表达过程被所有已知生命形式用来生成生命所需的生物大分子机制,包括真核生物(包括多细胞生物)、原核生物(细菌和古细菌)和病毒。
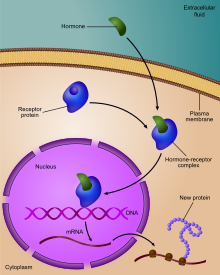

当基因在转录成 RNA 后被翻译成蛋白质时,它们就被表达了。一些基因在所有时间都表达,这些基因会受到组成性表达的控制。许多基因并非在所有时间都表达,而是在特定情况下才表达,无论是在特定细胞中还是在特定条件下。这些基因受到调控表达的控制。
基因组英属哥伦比亚制作的展示基因表达的视频
基因表达在转录过程中受到控制。基因是否被转录主要取决于特定 DNA 序列和与这些序列结合的特定蛋白质之间的相互作用。基因位于基因组中的特定位置,但这些位点没有任何区别特征。基因调控由调控位点控制,这些位点可以靠近或远离正在被转录的 DNA 区域。为了启动基因转录,调控蛋白将 RNA 聚合酶募集到基因组中的特定位点,这一过程发生在原核生物和真核生物中。

基因位于基因组中的特定位置,没有允许调控系统识别它们的任何区别特征,而是基因组中的其他序列有助于其调控。这些位点是特定 DNA 结合蛋白的结合位点,通常靠近正在被转录的 DNA,但它的邻近性并不必要。这些蛋白当成功地与位点结合时,会控制是否表达蛋白。
调控位点最初是在大肠杆菌中发现的。在大肠杆菌中,在存在乳糖的情况下,表达编码一种酶的基因,这种酶可以将乳糖转化为能量来源。这种酶的核苷酸序列几乎是完美的反向重复。
5'-...TGTGTGGAATTGTGAGCGGATAACAATTTCACACA...3'
3'-...ACACACCTTAACACTCGCCTATTCTTAAAGTGTGT...5'
像这样的对称调控位点通常对应于结合该位点的蛋白的对称性。
操纵子模型是由弗朗索瓦·雅各布和雅克·莫诺德在一系列实验后提出的,作为基因表达的常见机制。该模型由一个调控基因、一个作为调控 DNA 序列的操纵子位点和一组结构基因组成。调控基因编码一种阻遏蛋白,这种蛋白会与操纵子位点结合,从而阻止转录。操纵子位点和结构基因集合共同构成所谓的操纵子。操纵子是原核生物中常见的调控单元。
基因控制的主要方法是调控转录。转录可以通过多种策略进行调控。
- 特异性因子 - 通过改变 RNA 聚合酶对启动子的结合可能性来改变其特异性。
- 阻遏蛋白 - 结合到靠近或重叠 DNA 启动子区域的非编码序列上。它阻碍了 RNA 聚合酶的进程,从而阻断了基因的表达。
- 通用转录因子 - 这些因子将 RNA 聚合酶置于蛋白质编码序列的起始位置,并释放聚合酶转录 mRNA。
- 激活蛋白 - 加速基因表达速率
- 增强子 - 将特定启动子带到起始复合物
表达系统是一个系统,在该系统中,特定基因产物的产生是专门设计的。这种系统由一个基因组成,该基因通常由 DNA 编码,以及将 DNA 转录成 mRNA 并使用提供的试剂将 mRNA 翻译成蛋白质所需的分子机制。虽然这个过程在每个细胞中都被利用,但这个术语更常用于实验室工作。另一方面,表达系统从根本上来说是一个自然过程。
首先,染色质纤维指的是核小体串,这些核小体串通常与缠绕以进行压缩以及将基因组装配到细胞核中有关。根据各种研究人员的说法,这些核小体串(也称为 10nm 染色质纤维)构成了转录活性材料的模板。如今,随着染色质构象捕获和冷冻电子显微镜等新实验的开发,关于 30nm 染色质纤维在原位证据是否出现的问题出现了。
一位名为罗杰·科恩伯格的研究人员成功分离了包含组蛋白-DNA 复合物的染色质亚基,后来被称为核小体。这些相同的染色质亚基标志着 10nm 染色质纤维的“串珠”。一些实验,如 X 射线衍射、中子散射和能量损失电子显微镜,使研究人员能够观察到 10nm 纤维展示了 DNA 围绕核小体核心缠绕 1.75 圈。关于 10nm 染色质纤维的一些重要观察结果是:a) 组蛋白具有正电荷,导致 DNA 核心带正电,抵消了 DNA 负电骨架的电荷,b) 压缩是由于 DNA 围绕组蛋白核心缠绕而发生的,这使得基因组能够驻留在其中。
此外,另一位名为阿伦·克鲁格的研究人员指出,大量基因组拥有 30nm 纤维,但这些纤维是通过压缩将 10nm 纤维缠绕成螺线管而产生的。这种压缩通常取决于各种蛋白质和二价金属离子,如镁。
有多种模型可以帮助我们观察这些染色质纤维。这些模型包括单螺旋螺线管模型、双螺旋之字形模型、交联剂模型和超核小体模型。
1) 螺线管模型:处理 10 纳米纤维绕对称轴盘绕的情况,其中核小体紧密排列在一起。
2) 双螺旋之字形模型:处理两个核小体呈之字形排列,随后形成螺旋构象。
3) 交联剂模型:处理 DNA 在螺旋轴上交叉来回穿梭的情况,其中紧密排列的核小体彼此相对。该模型与双螺旋之字形模型非常相似。
4) 超核小体模型:处理由连接序列分隔的核小体聚集成团的情况。
-- 在这些模型中,只有螺线管模型和双螺旋之字形模型得到了广泛认可和一致性。
最早观察到 30 纳米纤维的实验之一是用海星精子进行的,该实验在体内通过电子显微镜和电子能谱成像技术进行。这些研究表明,10 纳米纤维折叠并扭曲成 30 纳米的宽度。
1) 冷冻电镜:另一种技术是冷冻电镜,它允许研究人员观察有丝分裂染色体及其在染色质完全浓缩形式下缺乏 30 纳米纤维。这种特定技术实际上有一些优势,例如样本完全水合且不需要使用交联剂进行化学固定。这使得研究人员更容易观察染色质,并且也推断出 10 纳米染色质可能在能量上更有利,因为它们存在于完全浓缩状态中。
2) 模型镊子实验:使用该实验的单分子力谱测量将包含 25 个核小体重复序列的 30 纳米染色质纤维转化为延伸的 10 纳米纤维所需的力。该实验提供亚皮牛顿力分辨率,这使得能够对染色质纤维构象之间的转变进行精确的数学建模。
3) 体外电镜研究:这些实验与核小体阵列的重建有关。这支持了双螺旋和螺线管模型。然而,该模型的缺点是它不清楚 30 纳米螺线管染色质纤维的规则间距如何能够适应体内存在的核小体不规则间距。
4) 电子能谱成像 (ESI):这是一种高对比度技术,不依赖于重金属对比剂。它仅基于与样品中特定元素相互作用的电子,生成高对比度的暗场图像,该图像随后可用于观察核小体。
5) 染色体构象捕获 (3C) 技术:说明了酵母基因组中的亚染色体域不存在为紧凑的 30 纳米纤维,而是存在为延伸的纤维。该技术发现的线性质量密度小于 FISH 实验中发现的线性质量密度,这与 30 纳米染色质纤维不一致。该技术只能找出感兴趣位点的长程染色质相互作用,因此存在技术局限性。
6) DNA 荧光原位杂交 (FISH):在这种技术中,假设 30 纳米纤维是体内染色质折叠的最低级。这通常在酵母中进行,在酵母中可以观察到灵活的 30 纳米样聚合物。然而,有人争论说,这种技术可能缺乏足够的解析度来确定与弯曲程度较小的 30 纳米纤维相比,高度弯曲和扭结的 10 纳米纤维。
7) Hi-C 技术:该技术由 Lieberman-Aiden 完成,是对 3C 方法的改编,它允许对人类淋巴母细胞系进行长程染色质相互作用的无偏识别。在这种技术中,使用限制性内切酶消化交联的 DNA 以创建填充有核苷酸的 5' 突出端。
1. 趋势生物化学科学; 2011 年 1 月,第 36 卷第 1 期,第 1-6 页,共 6 页 2. Berg, Jeremy M., John L. Tymoczko 和 Lubert Stryer. "基因表达的控制。"生物化学。第六版。纽约:W. H. Freeman,2007 年。第 892-99 页。印刷版。
