结构生物化学/蛋白质/冷冻电子显微镜
冷冻电子显微镜专门用于解释和可视化未染色生物复合物,例如病毒、小型细胞器和大分子生物复合物,这些复合物大小在 200 kDa 或更大,保存在玻璃态(即玻璃态或非晶态)冰中。基本目标是将其他电子显微镜技术与冷冻固定技术进行比较,以快速冷冻生物样品,以避免破坏其水性环境。这避免了超微结构改变、元素重新分布和物质洗脱。冷冻在玻璃态冰中的样本显示出与液态或天然状态相似的结构。近似天然成像条件允许对细胞机器进行三维重建。利用最先进的计算机控制的自动化显微镜、图像重建软件和可视化工具,可以实现大型生物复合物的亚纳米级分辨率结构。在冷冻电子显微镜中,电子束,即高能粒子流轰击样品。观察到的图像是在样品与该束相互作用的结果。形成高分辨率图像的大多数电子出现在弹性散射中,其中仅改变了它们的轨迹,但它们的能量不受影响。但是,一小部分电子将它们的部分能量转移到样品。这种能量积累并可以破坏分子键,一段时间后破坏样品。因此,为了进行高分辨率成像,低剂量参数要求在实际拍摄图像之前不暴露要成像的区域。
冷冻电子显微镜可以通过各种样品制备方法进行,两种常用的方法使用生物材料的薄膜和玻璃态切片。薄膜法要求将生物材料放置在电子显微镜网格上,并在接近液氮温度下快速冷冻。较大的样本(玻璃态切片)可以通过不同的方法进行玻璃化,包括高压冷冻。然后可以将这些样品切成薄片并放置在电子显微镜网格上,类似于薄膜。这些样品必须保持在液氮温度下才能进行高真空处理,并暴露于电子束中。
冷冻电子显微镜的一个分支是冷冻电子断层扫描(CET)。冷冻电子断层扫描在低温下进行,就像冷冻电子显微镜一样;CET 从 2D 图像构建 3D 样品。
冷冻电子显微镜用于各种领域。纳米粒子研究严重依赖电子显微镜来可视化小粒子。进行药物研究的制药公司利用电子显微镜来帮助预测药物和生物物质的行为。在药物研究的情况下,3D 可视化非常有用,并且在冷冻电子显微镜中,对样品的损伤最小,可以获得可用的图像。
- 允许检查生物样品的天然和水合结构特征。样品始终处于溶液中,并且从未与附着表面接触。因此,观察到的形状是水合分子在溶液中的真实形状,并且没有因其自身附着和压平在支撑膜上而发生扭曲。
- 在显微镜真空条件下提供良好的生物结构保存。
- 没有染色剂或化学固定剂会扭曲样品。当染色时,样品会以多种方式受到损坏,例如压平和扭曲。
- 当样品附着在碳网格上时,它可能以优先方向粘附。如果发生这种情况,那么最终图像集中将缺少信息(缺失锥体),并且计算模型在该方向上的分辨率将不存在。
- 与类似的糖包埋或冷冻干燥的样品相比,在室温下,辐射损伤降低了 2-5 倍。据认为,其背后的原因是减少了由键断裂导致的片段的温度依赖性重排或扩散。在固体冷冻状态下,重排或扩散减少,并且蛋白质构象更可能在更高的照射水平下保持。
- 可以观察到核酸、蛋白质和脂质之间的对比度得以区分。
- 使人们能够控制化学环境,以便可以检查分子的不同功能状态。
- 信噪比非常低。生物大分子通常由碳、氢、氧和氮组成。这些分子的电子吸收非常低。因此,图像对比度也非常低,在处理仅几幅图像时很难检测到特征。
- 难以从倾斜的样品获得图像。倾斜冷冻样品的冰横截面太厚,无法产生良好的图像。
- 成像倾斜冷冻样品时,充电现象更加普遍。
- 生成样品需要更多时间。但是,这通常不是一个大问题,尤其是在设计好工作方案并且易于获得良好样品之后。
- 如果无法轻松形成玻璃态冰,那么形成的立方冰会非常容易吸收电子,并且冷冻样品基本上毫无价值。
- 样品必须保持在低于 135 摄氏度。
以下是制备冷冻水合生物样品的一些通用程序:1)开发生物样品的薄层。 2)快速冷却样品至玻璃态。 3)将样品转移到电子显微镜中,不要重新加热到高于玻璃化温度。 4)在低于玻璃化温度下观察样品,电子剂量足够低以保护样品的结构。

断层扫描利用能量波对固体物体的效果和差异来产生内部结构的三维图像。冷冻电子断层扫描是冷冻电子显微镜的一个分支,其中在低温下,冷冻样品的二维投影被记录并用于通过计算机反投影来重建三维结构。这是使用透射电子显微镜来拍摄样品的连续图像,同时围绕轴线倾斜样品来完成的。 “投影定理”指出可以从沿不同方向的投影中检索 3D 物体。因此,要获得物体的 3D 描述,必须沿不同方向投影它;这是通过逐步倾斜样品来实现的。由于透射电子显微镜 (TEM) 的局限性,样品只能倾斜到 +- 60-70 度,而不是 90 度,这对于检索有关样品的全部 3D 信息是必要的。
冷冻电镜断层扫描 (Cryo-ET) 是一种非常精确的方法,可以确定样品的 3D 结构,因为快速冷冻样品和低温条件可以很好地保留结构并对某些过程进行良好的时间分辨率。例如,在细胞过程中的某个时刻快速冷冻细胞和组织可以很好地了解这些细胞和组织在该特定细胞过程中的某个时刻的结构和活性。这种断层扫描有助于以更动态的水平学习细胞及其细胞器。为了便于观看过程,细胞的每个细胞器都用不同的颜色呈现。冷冻细胞是为了使细胞保持其原始结构。冷冻此类样品的方法是将它们放置在网格上,在薄层水中将其吸干,然后将其浸入乙烷中,然后再将其储存在液氮中。冷冻电镜断层扫描用于研究几乎所有样品,例如病毒。该工具可以帮助理解病毒的复制状态,以及病毒可能变成的单个结构。最近的一项研究对病毒的刺突蛋白和刺突蛋白影响病毒的各种结构进行了研究。如今,冷冻电镜断层扫描被用于帮助寻找癌症的治疗方法,方法是评估蛋白质的构建块,即钙粘蛋白,这些蛋白质有助于阻止癌细胞在全身扩散。通过冷冻电镜断层扫描获得的信息有助于理解和了解许多细胞过程的结构基础,因此也能理解其功能。
冷冻电镜断层扫描存在一些局限性。主要局限性是样品的厚度。样品必须足够薄,才能使其冻结良好,以便可以用透射电镜(TEM)正确地收集它。如果样品太厚,则必须在温度仍很低的情况下将其切成更薄的切片,以防止再结晶。获得图像有两种方法:一种是通过固定倾斜增量,另一种是通过渐变倾斜增量。渐变倾斜增量更受青睐,这是因为倾斜增量与倾斜角度的余弦成正比。冷冻电镜断层扫描的另一个问题是辐射损伤。为了防止辐射损伤,应在低电子剂量条件下对样品成像,这导致获得的 3D 图像分辨率更低,并且也限制了冷冻电镜断层扫描所需的样品厚度。
二十面体重建
[edit | edit source]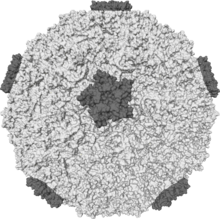
二十面体重建是指冷冻电镜在阐明具有适当(二十面体)对称性的颗粒的结构中的应用。二十面体样品的高内部对称性使确定对称元素的位置变得更容易,从而减少了确定样品 3D 结构所需的图像数量。 [1] 这可能看起来像是对显微镜的任意且不相关的应用,但实际上,存在着大量的颗粒包含这种对称性。二十面体颗粒的例子包括大多数人类病毒,以及一些分子,如十二氢-闭合-十二硼酸根离子 (B12 H122-) 和富勒烯。

许多病毒结构已经通过二十面体重建被预测并随后通过实验确定。二十面体属于高对称性群 Ih,该群包含 120 个对称操作,其中最独特的是六个五重对称轴。 [2] 该对称性群的性质对于冷冻电镜的应用至关重要。 [3]
螺旋重建
[edit | edit source]螺旋重建是一种利用冷冻电镜来开发某些“丝状”生物结构的三维结构的方法。只要存在螺旋对称性,该方法使用冷冻电镜的 2D 投影图像来生成这些 3D 图像。然而,该方法不能应用于包含“接缝”或“扰动”的结构。有一种称为不对称螺旋重建的新方法可以应用于包含“接缝”的螺旋结构。与传统的螺旋重建方法类似,傅里叶变换图像用于生成层线数据,然后这些数据用于生成 3D 结构。
螺旋重建允许通过一种类型蛋白质分子的规则接触形成大型基团。螺旋对称性可以在丝状病毒(例如,Pf1)中找到,在肌动蛋白、微管蛋白或其他细胞骨架的蛋白质中找到,或者在形成折叠在圆柱体表面的 2D 晶体的蛋白质中找到,例如乙酰胆碱受体或 CopA。
2D 与 3D 螺旋重建的基本思想:图 1 和图 2。链接:http://www.nysbc.org/facilities/CEM/cryoem-generalinfo.html 通过电子显微镜方法观察蛋白质的 2D 和 3D 图像。
电子晶体学
[edit | edit source]电子晶体学是一种显微镜形式,它使用电子束来构建小型固体(如蛋白质)的图像。该过程用于根据电子散射确定和预测蛋白质的结构和排列,这些结构和排列来自二级结构晶体,例如α螺旋或β折叠。它可以用来研究有机物和无机物,以及蛋白质结构。电子晶体学在许多方面补充了 X 射线晶体学,但也在 X 射线晶体学失败的地方取得了成功。例如,X 射线晶体学研究需要蛋白质的四级结构,而四级结构往往比二级结构难以获得。电子晶体学存在一个问题,即它会导致正在分析的蛋白质受到辐射损伤。这阻碍了显微镜过程的范围和功能。为了减少辐射损伤,实施了冷冻固定,在这种情况下,成像是在非常低的温度(如液氮的温度)下进行的。当特定蛋白质容易被显微镜中的电子变性或损坏时,此资源特别有价值。
晶体结构确定包括两个步骤:‘求解’,它使用 EM 图像找到大约 0.25 Å 内最重原子的模型;以及‘精修’,它使用选区电子衍射或会聚束电子衍射数据找到大约 0.02 Å 内的所有原子。
使用电子衍射来研究晶体结构始于 1937-1938 年的莫斯科,当时是由 Pinsker 和 Vainshtein 带领的一组科学家。他们的研究使用了他们自己的电子衍射相机,这些相机具有相对较低的加速电压来记录不同材料的电子衍射数据。根据这些数据,他们能够定位晶体结构中的氢原子,而这在使用 X 射线衍射时无法做到。为了求解未知结构,需要相位信息,该信息首先由 Hauptmann 和 Karle 于 1953 年引入,称为“直接方法”。将直接相位方法与现代计算机结合使用,电子晶体学在晶体和其他分子的结构确定方面取得了重大进展。
有两种不同的电子衍射技术:1)选区电子衍射 (SAED),它需要近似运动学条件,并适用于单位晶胞尺寸>10 Å 以及薄样品<200 Å;2)会聚束电子衍射 (CBED),它利用动态效应,并适用于单位晶胞尺寸<10 Å 以及厚样品>200 Å。
为什么使用电子?与 X 射线相比,电子与物质的相互作用强度高 10^4-10^5 倍;并且它们在高分辨率电子显微镜图像中存在相位。
与 X 射线晶体学相比,电子晶体学具有一些关键优势。其中一个优势是电子晶体学可以分析更小的晶体。这是因为电子与物质的相互作用比 X 射线更强。另一个优势是电子束可以通过磁透镜聚焦形成图像,而 X 射线无法形成图像。由于电子与物质相互作用的机制是基于电子检测晶体中的势分布,而 X 射线取决于 X 射线检测电子密度分布的机制,因此电子晶体学可以在 X 射线晶体学无法应用的某些情况下使用。例如,晶体中原子的氧化态。
使用电子晶体学来确定结构非常重要,因为它能够观察蛋白质的自然形式。通过利用电子晶体学,人们可以在蛋白质所在的结构中观察脂质-蛋白质双层中的蛋白质,从而可以更好地确定其功能。
单粒子电子显微镜
[edit | edit source]从一系列二维图像重建三维分子图像的技术被称为电子显微镜。由于其易于访问的特点,它为结构生物化学家提供了许多生物分子结构信息的深入见解。
为了从这种方法获得三维结构,必须满足两个要求。
1. 蛋白质的合理大小,可以形成大型宏观分子组装体,无需使用晶体。2. 分子必须以许多相同的拷贝存在。
电子断层扫描产生的分辨率较低,噪声较高。单颗粒电子显微镜的主要目标是确定收集到的投影图像之间的几何关系。在 2008 年,科学家们能够追踪多肽链的骨架并建立原子模型。单颗粒电子显微镜确实能够以接近原子分辨率提供结构信息。
三维结构将是以下步骤的结果:
1) 样品制备。这是收集样品并将其放置在(金属)板上以产生最佳对比度的步骤。有三种技术用于制备单颗粒电子显微镜的样品。a) 负染色:分子吸附在连续的碳膜上,将分子放入金属板上进行干燥。b) 玻璃化:将样品浸入液态乙烷中以在天然环境中保存分子,它产生低对比度。(将它们保存在完全水合的状态下)玻璃化是**最佳**的标本制备方法,但不适用于异质样品。c) 冷冻负染色:高对比度图像浸入高离子强度的饱和钼酸铵溶液中。它适合研究小而异质的样品。
2) 粒子挑选。这是所有过程中最繁琐的过程之一,因为电子显微镜学家必须根据其在方向上的相似性对粒子进行分类和分离;所有这些工作都是手工完成的,以实现最高效率。话虽如此,已经开发了自动挑选程序,但由于信噪比低,它们无法执行任务。此步骤的结果是收集了少量单个粒子图像。

维基百科编辑 _随机锥形倾斜_。
4) 细化。初步模型用于使用欧拉角计算更好的对齐,即粒子在平面内旋转/未旋转的位移。从细化后的数据中,重建了分子的新三维结构。当应用测试负温度时,会发生过度细化的风险。
单颗粒电子显微镜比电子晶体学具有经济上的优势。与晶体学不同,它不需要晶体——这意味着样品不需要是纯净的——这需要更多的工作才能实现。另一个优点是单颗粒电子显微镜只需要很少的样品,这总是让研究人员感到高兴。
然而,单颗粒电子显微镜的缺点之一是难以确定密度图的分辨率及其准确性。由于没有方法来检查准确性,因此通常只能做的事情是重复该过程并将结果与以前的结果进行比较。因此,结果只能根据一致性而不是准确性进行评估。
蛋白质-脂质阵列[edit | edit source]
电子晶体学已被用于通过分析称为蛋白质-脂质阵列的样品阵列来研究膜蛋白。这些阵列可以以多种不同的方式排列,并提供许多优点和缺点。对膜蛋白产生最有用密度图的两种形式是二维片状晶体阵列和管状晶体阵列。它们非常精确,可以揭示有关单个脂质分子和蛋白质侧链的信息,这是由于对片状或管状阵列图像中的许多晶胞进行了平均,从而增强了较差的信噪比。
在过去的几十年中,从去垢剂溶解的纯化蛋白质中创建管状和片状蛋白质-脂质阵列的方法有了很大的进步,但在筛选方面没有像易于操作的机器人筛选这样的进步。尽管该领域进行了大量研究,但他们仍然缺乏快速可靠地检查样品质量的能力。几个实验室通过增强现有软件,改进了这些晶体的分析方法,使该过程更加高效和用户友好。
尽管它们的螺旋阵列对称性在确定结构方面具有很大的优势,但管状晶体没有像片状晶体那样广泛使用。管状晶体的一幅图像包含许多相同分子的不同视图,足以在三维空间中重建它,而无需倾斜。为了纠正变形,管状晶体以与片状晶体类似的方式进行处理,其中两个重复长度被分成更短的片段,然后与参考结构进行比较,以确定识别完整结构所需的参数。此过程传统上使用傅立叶-贝塞尔方法来评估数据,使它们能够分析螺旋对称性保持的程度以及垂直于管轴的二重对称性,从而可以校正结构不同层级的聚焦变化。还开发了一种不使用傅立叶-贝塞尔方法,而是将片段视为单个粒子串的替代方法。这种替代方法正变得越来越流行,用于从无序的螺旋聚合物(如管状蛋白质-脂质晶体)中提取结构信息。这显示出从管状结构确定接近原子分辨率的结构的巨大潜力。
电子晶体学涉及的方法包括自由捕获以创建不同的构象状态。为了冷冻捕获标本,将电子显微镜网格放入液氮冷却的乙烷中,乙烷使标本快速冷却,从而可以捕获脂质-蛋白质阵列的结构,该结构的寿命为毫秒或更长。冷冻捕获的蛋白质可以通过光或适当的配体激活。近年来,氦冷却顶置冷冻捕获的发展导致数据收集图像更加清晰,并有望更详细地描述蛋白质-脂质双层的门控机制。
此外,分子断层扫描用于探索蛋白质的功能环境。通过从一系列倾斜视图中拍摄图像,可以创建整个场景的三维图像,从而创建更好的三维图像。
单分子方法[edit | edit source]
这是一种观察单个分子动力学行为以确定单个分子水平的作用机制,以及识别、分类和比较细胞内亚群和亚结构的方法。为了表征分子结构的动力学,科学家们观察单个分子的实时轨迹;通过观察其中许多分子,可以计算出总体上动力学特性的直方图。
与单分子方法相比,X射线晶体学或核磁共振提供了详细的结构视图,但受到静态分子视图和整体平均值的限制。
1. 单分子操作:在这种方法中,分子连接到外部探针,该探针对分子施加定义的力或扭矩,以表征其机械特性。这种方法也称为原子力显微镜 (AMF)。因为细胞被视为一个工厂,其中许多过程是由专门的机器完成的,这些机器将化学能转化为力、扭矩和机械功——探针将探测到这种动力学和机制。这种方法最近被用来研究 RNA 分子的折叠和展开以及催化这些反应的酶,以及研究 RNA 聚合酶。
2. 单分子检测:分子在两个位置用荧光标记,以“供体”和“受体”的形式存在,它们可以进行荧光共振能量转移 (FRET)。然后可以观察分子的轨迹,关于探针荧光强度的变化或关于 FRET 的变化。这种方法的另一个名称是荧光方法。这是一种强大的方法,可以研究分子的动态行为、它们的稳定性以及追踪粒子在细胞内外移动。例如,这种方法被用来研究核糖体在翻译过程中的多种相互作用。
在上述两种方法中,荧光检测方法更受研究人员的青睐,因为它需要更简单的仪器,但缺点是仪器收集的光子有限。
参考文献- [edit | edit source]
- ↑ Jiang, Wen, Zongli Li, Zhixian Zhang, Christopher R. Booth, and Matthew L. Baker. "Semi-automated Icosahedral Particle Reconstruction at Sub-nanometer Resolution." Journal of Structural Biology . 136. (2001): 214-225. Print.
- ↑ Miessler, Gary. Inorganic Chemistry. 3rd ed. Dehli: Pearson Education in South Asia, 2007. Print.
↑ Fuller, S.D., and S.J. Butcher. "Three-Dimensional Reconstruction of Icosahedral Particles-The Uncommon Line." Journal of Structural Biology . 116. (1996): 48-55. Print.
Yifand Cheng and Thomas Walz. 模板:单颗粒电子显微镜中接近原子分辨率的出现
Yoshinori Fujiyoshi and Nigel Unwin. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2651081/ 2008
Heel, Marin Van. http://sbio.uct.ac.za/Sbio/Arvind/Structure/Review_MvH.pdf 2006
Unknown. http://www.answers.com/topic/cryo-electron-microscopy. 2007
绝对天文学。 http://www.absoluteastronomy.com/topics/Electron_crystallography 2009
纽约结构生物学中心 http://www.nysbc.org/facilities/CEM/cryoem-generalinfo.html
Thomas E. Weirich,János L. Lábár,邹晓冬 http://books.google.com/books?id=rAocx9cElUcC&pg=PA3&lpg=PA3&dq=electron+crystallography&source=bl&ots=8rpqKYGhsK&sig=roVEpejLT5obY4Pi7UZthhn5-Wk&hl=en&ei=xpzaSqK3JYjYtgPHwJCyCQ&sa=X&oi=book_result&ct=result&resnum=3&ved=0CBQQ6AEwAg#v=onepage&q=&f=false
Cheng Y,Walz T。 http://www.ncbi.nlm.nih.gov/pubmed/19489732
什么是电子晶体学 - 邹晓冬 http://www.nanomegas.com/files/ElectronCrystallographyPRINCIPLES.pdf
在单一生物化学:当少即是多 http://www.annualreviews.org/doi/abs/10.1146/annurev.biochem.012108.120952?journalCode=biochem
