结构生物化学/蛋白质/X射线晶体学
X射线晶体学
X射线晶体学可以揭示数千种蛋白质的详细三维结构。 X射线晶体学分析中的三个组成部分是蛋白质晶体、X射线源和探测器。
X射线晶体学用于**通过研究分子生长固体晶体来研究分子结构。**晶体学家将高能X射线照射到包含数万亿个相同分子的微小晶体上。晶体将X射线散射到电子探测器上。电子探测器与在数码相机中捕获图像的类型相同。在每次持续几秒到几小时的X射线照射后,研究人员通过将所需的方位输入控制X射线设备的计算机来精确旋转晶体。这使科学家能够在三维空间中捕获晶体散射或衍射X射线的方式。每条衍射射线的强度被输入到计算机中,计算机使用数学方程来计算结晶分子中每个原子的位置。结果是分子的三维数字图像。
晶体学家用埃测量原子之间的距离。测量埃距离的完美“尺子”是X射线。晶体学家使用的X射线长约0.5到1.5埃,正好适合测量分子中原子之间的距离。这就是为什么使用X射线。
蛋白质X射线晶体学是一种用于通过其结晶形式的X射线衍射获得特定蛋白质的三维结构的技术。这种三维结构对于确定蛋白质的功能至关重要。制作晶体会在其中形成晶格,这种技术将数百万个蛋白质分子排列在一起,使数据收集更加敏感。就像拿一堆纸,用尺子测量宽度,然后用页数除以该长度来确定一张纸的宽度。通过这种平均技术,噪声水平降低,信噪比提高。[2]蛋白质的活性位点和结合位点的特异性完全取决于蛋白质的精确构象。X射线晶体学可以揭示蛋白质分子中大多数原子的精确三维位置,因为X射线和共价键具有相似的波长,因此目前提供最佳的蛋白质结构可视化。正是罗莎琳德·富兰克林的X射线晶体学,使J.D.沃森和F.H.C.克里克能够弄清楚DNA的双螺旋结构。
我们使用此程序来掌握细胞机制以及酶和其他大分子3-D结构的知识。至关重要的是,我们能够更好地理解细胞中发生的每个化学反应都需要特定酶才能发生。用于分析蛋白质结构的两种常见技术是核磁共振 (NMR)和 X射线晶体学。X射线晶体学可用于分析任何不同化合物,最高可达 106 (g/mol) 的分子量;而NMR则限于生物聚合物(由生物体产生的聚合物,如淀粉、肽、糖),分子量不超过 30,000 (g/mol)。它还可以测量非常小的化合物,因为测量分子中原子之间距离的适当尺寸为 0.5 到 1.5 埃。X射线用作辐射形式,因为它们的波长与共价键的顺序相同(约 1 Å 或 1 * 10−10m),这对于获得揭示分子结构信息的衍射图样是必需的。如果辐射的波长远大于或远小于共价键的键长,则光不会衍射,也不会获得关于结构的新知识。
完成 X射线晶体学分析所需的三个组成部分是**蛋白质晶体**、**X射线源**和**探测器**。
该过程从结晶感兴趣的蛋白质开始。蛋白质的结晶会导致所有蛋白质原子相对于彼此以固定的方式定向,同时仍保持其生物活性构象——这是 X射线衍射的必要条件。必须从溶液中沉淀或提取蛋白质。这里的经验法则是获得尽可能纯的蛋白质来生长大量晶体(这使得晶体具有带电特性,并且表面带电分布有利于更好的散射结果)。为了实现蛋白质结晶,需要采取 4 个关键步骤,它们是
- 纯化蛋白质。确定蛋白质的纯度,如果不纯(通常 > 99%),则必须进行进一步的纯化。
- 必须沉淀蛋白质。通常通过将蛋白质溶解在适当的溶剂(水缓冲溶液,含 2-甲基-2,4-戊二醇等有机盐)中来实现。如果蛋白质在水缓冲液或水有机缓冲液中不溶,则必须添加去垢剂(如十二烷基硫酸钠)。
- 必须使溶液达到过饱和(使蛋白质从其余溶剂中凝结,形成凝结核)。这通过向浓缩的蛋白质溶液中添加盐来实现,降低其溶解度,使蛋白质形成高度有序的晶体(这个过程被称为盐析)。其他方法包括分批结晶、液-液结晶、蒸气扩散和透析。
- 让实际的晶体生长。由于形成了核晶体,这将导致获得实际的晶体生长。
关于重结晶技术的说明,以获得更纯的蛋白质
重结晶是一种非常重要的技术,用于纯化物质。了解固体在特定溶剂中的溶解度是重结晶的关键。这种技术的应用在制药学和其他许多领域都有体现。例如,晶体学家使用核磁共振和X射线衍射方法来深入了解不同的化合物。X射线衍射需要形成纯净的晶体才能获得准确的结果。晶体学家可以通过X射线衍射深入了解蛋白质结构,但为了能够使用X射线检查它们的晶体,他们必须首先花费时间形成纯的蛋白质晶体。形成蛋白质晶体非常困难,甚至可能需要数年时间和极其特定的条件。温度、pH值和浓度必须非常具体,才能形成具有纯净结构的大型晶体。在这个过程中,重结晶对于去除晶格中的杂质至关重要。如今,科学家使用晶体学和重结晶技术来理解蛋白质结构,并帮助理解蛋白质一级结构中单个异常如何导致疾病。总而言之,纯化技术对于使用X射线衍射来理解结构至关重要。在本实验中,我们探讨了微观重结晶和宏观重结晶之间的差异。重结晶中使用的技术包括找到一个合适的溶剂、重力过滤、缓慢冷却和真空过滤。成功的重结晶的关键在于合适的溶剂。我们需要一种在低温下不会溶解样品但在高温下会溶解样品的溶剂。这使得溶质在溶液在温热温度下溶解后沉淀出来。由于溶质只溶于温热的溶质中,因此在冷却后会形成沉淀。重力过滤用于去除重结晶前溶液中残留的不可溶性杂质,它也用于过滤掉用于去除颜色杂质的活性炭。重力过滤是有效的,但我们必须避免在这个过程中结晶,以避免纯净晶体在滤纸中损失。缓慢冷却对于确保晶体的纯度和大小也至关重要。当溶液缓慢冷却时,溶解的杂质有时间与溶剂相互作用,而不是被困在晶格中。在快速冷却过程中,杂质可能会被困在晶格中,因为结晶发生得太快,杂质没有时间返回到溶剂中。在将晶体放入冰浴中以确保最大程度的重结晶后,使用真空过滤将溶液过滤,以从包含杂质的溶液中提取纯净的晶体。在真空过滤后,收集并称重纯净的晶体。微观重结晶与宏观重结晶的不同之处在于用于过滤纯净晶体的仪器和技术。微观重结晶包括使用克雷格管和离心分离,而不是真空过滤。它用于少于300毫克固体的重结晶。
下一步,产生X射线并将其照射到结晶的蛋白质上。X射线可以通过四种不同的方法产生:
- 用高能电子束轰击金属源
- 将物质暴露在初级X射线束中,以产生次级X射线荧光束
- 从产生X射线的放射性衰变过程中(伽马射线与X射线没有区别)
- 从同步加速器(具有恒定频率电场的回旋加速器)辐射源
第一种和最后一种方法利用了轫致辐射现象,即加速电荷会释放辐射。
然后,将X射线照射到蛋白质晶体上,导致一些X射线穿过晶体,而其余X射线则被散射到不同的方向。X射线的散射也被称为“X射线衍射”。这种散射是由辐射的电场和磁场与晶体原子中电子的相互作用造成的。
这些图案是衍射X射线之间干涉的结果,受布拉格定律控制:,其中是两个电子密度区域之间的距离,是衍射角,是衍射X射线的波长,是一个整数。如果反射角满足以下条件
,
衍射X射线将发生相长干涉。否则,将发生相消干涉。
以下是一个相长干涉的例子
以下是一个相消干涉的例子
相长干涉表示衍射X射线处于相位或彼此对齐,而相消干涉表示X射线不完全处于彼此的相位。结果是,测量的X射线强度随着角度和探测器与晶体之间距离的变化而增加和减少。
被散射到各个方向的X射线随后被X射线胶片捕获,胶片显示乳剂的变黑程度与照射到胶片的散射X射线的强度成正比,或者被固态探测器捕获,例如数码相机中的探测器。晶体被旋转,以便X射线能够从各个侧面和角度照射到蛋白质。胶片上的图案揭示了有关蛋白质结构的许多信息。这种技术背后的三个基本物理原理是
- 原子散射X射线。衍射X射线的振幅与原子中的电子数量成正比。
- 散射波重新组合。如果光束处于相位,它们将在胶片上相互加强,如果它们处于相位,它们将相互抵消。每个原子都会对散射光束做出贡献。
- 三维原子排列决定了光束如何重新组合。
因此,点的强度及其位置是分析的基本实验数据。
最后一步是根据胶片上衍射图案的测量强度创建电子密度图。可以将傅里叶变换应用于胶片上的强度,以重建晶体的电子密度分布。在这种情况下,傅里叶变换采用电子密度的空间排列,并以X射线胶片上衍射图案的形式给出空间频率(原子之间的间距)。傅里叶变换的一个日常例子是音乐播放器上的音乐均衡器。均衡器不是显示实际的音乐波形,而是显示各种频率带的强度,因为音乐波形难以可视化。通过傅里叶变换,电子密度分布被显示为一系列堆叠在一起的平行形状和线(等高线),就像地形图一样。该映射提供了通过X射线晶体学观察到的电子密度的三维表示。在解释电子密度图时,需要考虑分辨率。5Å - 10Å 的分辨率可以揭示多肽链的结构,3Å - 4Å 的分辨率可以揭示原子组,1Å - 1.5Å 的分辨率可以揭示单个原子。分辨率受晶体结构的限制,对于蛋白质而言约为 2Å。
蛋白质分子非常大,因此它们的晶体比小分子晶体散射X射线束少得多。由于较大的分子具有较少的晶体,因此衍射散射和发射的强度非常弱。蛋白质含有碳、氮和氧,因此是较轻的元素(即它们具有较少的电子/原子);这很重要,因为电子是造成衍射和强度的主要原因,因此它们的X射线散射强度比重元素弱。了解这一点后,蛋白质晶体学家使用高强度X射线源(如旋转阳极管或强大的同步加速器X射线源)来分析蛋白质晶体。
- 原子中电子的数量与波的振幅成正比。例如,比较碳原子和氢原子,你会发现碳原子的散射强度是氢原子的六倍。
- 如果波在薄膜处同相,则波会相互叠加;如果波异相,则波会在薄膜处相互抵消。
- 观察散射波如何重新组合时,唯一重要的是原子排列。
X射线的能量
- 其中 *f* 是频率,*λ* 是波长。能量的 SI 单位是焦耳 (J)。
由于 X 射线的波长较短,因此其能量高于可见光。
相位问题
[edit | edit source]X 射线与晶体中电子的相互作用会产生衍射图案,从数学上来说,它是电子密度分布的傅里叶变换。然而,用于测量 X 射线的探测器只能测量衍射 X 射线的振幅;相移是使用傅里叶变换并找到电子密度分布所必需的,但使用此方法无法直接测量。这在物理学界被称为“相位问题”。简单来说,无法从测量的 X 射线振幅中找到相位。为了获得电子密度图,必须进行其他推断并进行额外的实验。许多时候,有关化合物物理和化学性质的现有数据可以在密度图较差时提供帮助。另一种称为帕特森合成的 方法对于找出初始相位估计非常有用,并且对于在相位未知时确定蛋白质结构的初始阶段非常有用。通过使用帕特森合成找到一个原子(通常是一个重金属原子)可以简化问题,然后使用该原子的位置来估计初始相位并计算初始电子密度图,这可以进一步帮助对其他原子的位置进行建模并进一步改进相位估计。另一种方法称为分子置换;它定位了蛋白质结构在细胞中的位置。除了分子置换方法外,相位问题也可以通过同晶置换方法、多波长异常衍射方法、单波长异常衍射方法和直接方法来解决。
分子置换
[edit | edit source]如果有一个可以计算相位的原子模型,则可以解决相位问题。如果已知相关蛋白质结构,则可以获得模型。但是,为了构建此原子模型,需要确定模型在新的晶胞中的方向和位置。这时,分子置换 (MR) 技术就派上用场了。
分子置换,也称为 MR,是一种解决 X 射线晶体学中相位问题的 方法。MR 定位了蛋白质结构与其晶胞的方向和位置,其蛋白质结构与需要确定的未知蛋白质结构同源。获得的相位可以帮助生成电子密度图,并帮助生成蛋白质结构模型位置的计算强度,以与 X 射线晶体学实验中观察到的结构相对应。
MR 方法对于解决大分子晶体结构也同样有效。由于不需要制备重原子衍生物和收集数据,因此这种方法需要更少的时间和精力来确定结构。该方法很简单,模型构建也简化了,因为它不需要链追踪。
此方法包括两个步骤
- a. 旋转搜索以将同源模型定位到晶胞或目标中
- b. 平移目标,其中新的定向模型定位在晶胞中
帕特森基 (分子置换)
[edit | edit source]帕特森图是原子间矢量图,包含晶胞中每个相关原子的峰值。如果帕特森图是根据从电子密度图得出的数据生成的,则只有当模型正确定向并放置在正确位置时,两个帕特森图才应该紧密相关。这将使我们能够推断有关未知蛋白质结构与其细胞位置的信息。然而,分子置换存在一个问题,它有六个维度,三个参数用于指定方向和位置。使用帕特森图,可以将其划分为参数子集以分别查看每个部分。
旋转函数
[edit | edit source]旋转函数具有分子内矢量,这些矢量仅取决于分子的方向,而不取决于其位置,因为即使当分子在晶胞中平移时,所有原子都会移动相同的距离,但原子之间的矢量是相同的。比较未知蛋白质结构的帕特森图与同源已知蛋白质结构在不同方向上的帕特森图

经典旋转函数
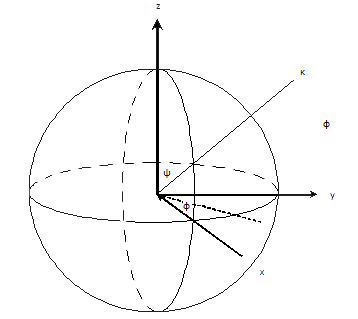
[edit | edit source]为了找到方向,确定旋转轴和绕该轴的旋转角。定义轴将需要两个参数(从球体中心到球体表面一点的矢量)。旋转轴最初与 z 轴平行,并绕 y 轴旋转角度 ᶱ,然后物体绕 z 轴旋转角度 ᶲ,最后绕旋转轴旋转角度 ᵠ。这些指定了单位球表面上的一个点。
快速旋转函数
[edit | edit source]可以通过比较两个帕特森图或这些帕特森图中的峰值来计算旋转函数。如果帕特森图用球谐函数表示,则可以通过傅里叶变换更快地计算旋转函数。
直接旋转函数
[edit | edit source]在直接旋转函数中,蛋白质结构可以放置在未知结构的晶胞中,并且将定向分子的帕特森图与整个未知结构帕特森图进行比较。
平移函数
[edit | edit source]一旦已知结构的方向,就可以对其模型(电子密度图)进行定向以计算结构因子,其中相关函数用于确定矢量以将模型平移到非对称单元内同源模型的顶部。
使用正确定向和平移的蛋白质结构相位模型,可以从导出的相位中准确地推导出电子密度图。电子密度图可用于构建和优化未知结构的模型。
多波长异常衍射
[edit | edit source]X射线是在称为同步加速器的巨大机器中产生的。同步加速器将电子加速到接近光速,并在一个大型的空心金属多边形环中穿行。在每个拐角处,磁铁弯曲电子流,导致以电磁辐射的形式发射能量。由于电子以光速运动,因此它们会发射高能X射线。
使用同步加速器的优点是,研究人员不必培养每种结晶分子的多个版本,而是只培养一种含有硒的晶体类型。然后,他们有能力调整波长以匹配硒的化学性质。这种技术被称为多波长反常衍射。然后用不同长度的波长多次轰击晶体,最终出现衍射图案,使研究人员能够确定硒原子的位置。该位置可以用作参考或标记来确定其余结构。这种优势使研究人员能够更快地收集数据。
该方法比较了原始蛋白质晶体与相同类型的晶体(至少添加了一个原子核数较高的原子)之间的 X 射线衍射图案。该方法用于确定小分子的结构,并最终由 Max Ferdinand Perutz(1914-2002)确定了血红蛋白的结构。完美的同晶是指原始晶体及其衍生物具有完全相同的蛋白质构象,分子的位置和方向以及晶胞参数。在完美的同晶中,晶体及其衍生物唯一的区别是由于在衍生物上添加了重原子而导致的强度差异。这些差异可以通过手动或通过自动帕特森搜索程序(例如 SIR 2002、SHELXD、nB 和 ACORN)来识别,此类信息对于确定蛋白质相位角非常重要。但是,由于晶胞尺寸的变化,完美的同晶几乎不会发生。对于含有重原子的蛋白质,其晶胞尺寸的容许变化为 dmin/4,其中 dmin 是分辨率极限。其他因素(例如旋转)也会导致非同晶。
- 制备几种蛋白质的衍生物,使其具有晶体结构。然后测量晶胞尺寸以检查同晶性。
- 收集原始蛋白质及其衍生物的 X 射线强度数据。
- 应用帕特森函数来确定重原子的坐标。
- 优化重原子参数并计算蛋白质的相位角。
- 计算蛋白质的电子密度。
衍生物通过两种不同的方法制成。首选的方法是在与母液成分相同的溶液中浸泡蛋白质晶体,但沉淀剂浓度略有增加。另一种方法是共结晶,但它不常用,因为晶体不会生长或异形生长。浸泡过程取决于晶体孔隙的宽度。孔隙应足够宽,以便试剂能够扩散到晶体中并到达晶体中所有蛋白质分子表面的反应位点。
多波长反常衍射(简称 MAD)是一种在 X 射线晶体学中使用的方法,它允许我们确定生物大分子(如蛋白质和 DNA)的结构,以解决相位问题。结构要求包括会导致 X 射线显着散射的原子;值得注意的是来自金属蛋白的硫或金属离子。由于硒可以取代天然硫,因此它更常用。使用这种技术极大地简化了晶体学家使用多同晶置换 (MIR) 方法,因为重化合物的制备是不必要的。
这种方法用于解决相位问题,当没有关于散射衍射的信息可用,除了振幅。此外,当重金属原子已经绑定在蛋白质内部,或者当蛋白质晶体不是同晶的,不适合 MIR 方法时,它被使用。该方法主要用于重金属溶液,这些金属酶通常来自第一过渡系列及其邻近元素。重要的是要有一个强大的磁场源来进行此实验,应考虑地下等环境。该方法还需要一种称为同步加速器的粒子加速器。
与多波长反常衍射 (MAD) 相比,单波长反常衍射 (SAD) 使用来自单个波长的单个数据集。MAD 和 SAD 之间的主要有利区别在于,晶体在 SAD 中花费在 X 射线束中的时间更少,这减少了对分子的潜在辐射损伤。此外,由于 SAD 只使用一个波长,因此比 MAD 更省时。
从单波长反常衍射数据推导出的电子密度图确实需要进行修改才能解决相位歧义。一种常见的修改技术是溶剂平滑,当 SAD 与溶剂平滑相结合时,由此产生的电子密度图与从完整 MAD 相位推导出的电子密度图具有可比的质量。溶剂平滑涉及调整蛋白质分子之间被溶剂占据的间隙区域的电子密度。假设溶剂区域与蛋白质相比相对无序且无特征。平滑溶剂区域的电子密度将增强蛋白质的电子密度到可解释的程度。这种方法被称为 ISAS,即迭代单波长反常散射。
直接法可以帮助使用它获得的数据来恢复相位。直接法使用三重关系来估计初始相位和扩展相位。三重关系是反射强度和相位与另外两个强度和相位之间的关系。使用此方法时,蛋白质结构的大小很重要,因为相位概率分布与原子数的平方根成反比。直接法是解决相位问题的最有用技术。
Paul Peter Ewald 和 Max von Laue 在 1912 年提出了使用晶体作为 X 射线的衍射光栅的想法。von Laue 提出,与可见光的较长波长相比,X 射线可能具有接近晶体晶胞间距的波长。他与 Walter Friedrich 和 Paul Knipping 合作,将硫酸铜晶体的 X 射线衍射记录到照相板上。von Laue 开发了散射角与晶胞间距大小及其在晶体中的方向之间的关系,并于 1914 年获得诺贝尔物理学奖。作为 von Laue 研究的结果,William Lawrence Bragg 开发了一项定律来连接晶体观察到的散射和从晶体中均匀间隔的平面上的反射。这可以用来推断原子结构,而布拉格定律对确定分子结构的重要性立即得到了认可。1914 年,第一个被解决的结构是食盐。它的电子分布证明了不仅共价化合物而且离子化合物都可以形成晶体。
1914 年,使用 X 射线解决了金刚石的结构,并表明碳到碳的单键长度为 1.52 埃。
X 射线晶体学首次用于在 1950 年代后期确定蛋白质结构。
John Kendrew 和 Max Perutz 都在剑桥大学工作,他们在 1945 年使用 X 射线晶体学发现了血红蛋白和肌红蛋白(肌肉中的氧气载体)的结构。他们于 1962 年获得诺贝尔化学奖。
通过 X 射线晶体学确定的第一个酶的三维晶体结构是鸡蛋清溶菌酶。这尤其重要,因为它提供了过渡态理论的视觉证据,因为它是过渡态几何形状互补于催化位点的物理证明。在第一个晶体结构之后,许多不同酶的 X 射线结构报告激增。[3]
药物发现和医学的许多进步很大程度上归功于 X 射线晶体学,通过识别当今流行的许多疾病中的药物靶点。例如,在 80 年代后期,科学家在使用 X 射线晶体学产生 HIV 蛋白酶的结构方面取得了突破,HIV 蛋白酶是一种对逆转录病毒生命周期至关重要的酶。该酶切割病毒蛋白链,这些蛋白链是未成熟病毒细胞的主要成分,将其切割成单独的成熟蛋白质,这些蛋白质可以继续形成更多成熟的和具有传染性的病毒颗粒。通过仔细观察其结构,特别是其对称性,研究人员开始制造与酶的活性位点相互作用的化合物,该活性位点位于其对称半部的中间,以关闭酶并阻止其正常运作。令人惊讶的是,到 90 年代中期,三种 HIV 蛋白酶抑制剂药物已上市,大大降低了艾滋病毒的死亡率。[4]
X射线晶体学不仅是药物发现的有力工具,也已被证实对改进药物疗效具有重要意义。例如,目前帕金森病的治疗方法是抑制单胺氧化酶B [MAO B],这种酶有助于神经递质的循环利用,通过去除一些有时至关重要的非活性分子成分来实现。虽然这种抑制剂有效,但会引起一些不良副作用,例如心率、血压、呼吸等的改变。然而,通过确定MAO B的三维结构,以及观察一些抑制剂如何与酶结合,埃默里大学的Dale Edmondson及其同事开始考虑开发新的药物,这些药物能更特异性地与酶结合,以最终减少副作用。[5]
此外,X射线晶体学有助于解释药物在体内的作用机制,它们如何相互作用,是什么让它们起作用等等。一个很好的例子就是广泛应用的阿司匹林。阿司匹林能通过阻断人体代谢和免疫系统中已知的环氧化酶(COX)来阻断前列腺素的产生,而前列腺素是一种在代谢中起着多种重要作用的信使分子。科学家们能够通过X射线晶体学研究COX酶并确定其结构,并由此清楚地了解酶结构的精确细节如何对其整体分子功能做出贡献。通过确定COX酶的三维结构,我们可以了解像阿司匹林这样的药物如何与之相互作用并阻断其活性。[6]
由美国宇航局进行的MIR实验表明,一些蛋白质在微重力环境下会产生更高质量的晶体。DCAM,即环境扩散控制蛋白质晶体生长法,是这些实验中使用的方法。DCAM采用液-液扩散法生长蛋白质晶体。这些蛋白质通过航天飞机轨道器被送往和平号空间站进行结晶。蛋白质晶体后来被带回地面进行X射线衍射分析。结果令人鼓舞。某些蛋白质所产生的最大晶体就是通过这些实验得到的。这些蛋白质包括溶菌酶、白蛋白和组蛋白八聚体。细菌视紫红质,一种与膜相关的蛋白质,产生了尺寸和质量都有所改善的晶体。此外,在微重力环境中进行的DCAM证明了成功地提供了异常大的蛋白质晶体,这些晶体可以用于中子衍射分析结构,而中子衍射技术无法用于在地球上生长的较小晶体。这些令人鼓舞的结果可以为地球上的人们带来许多益处。蛋白质晶体是制造治疗药物所必需的。科学家们可以通过在太空中培育蛋白质晶体来开发治疗疾病的新药,而这些晶体在地球上由于受重力的限制,无法生长到足够大的尺寸。
X射线晶体学的一些优势是,即使原子结构处于溶液中,该技术本身也能获得原子分辨率结构。这是因为晶体形式的结构与溶液中的结构相同。[需要引用]另一个优势是,原子结构包含大量关于结晶纯蛋白质的数据。从蛋白质结构中获得的信息可以提供比在细胞环境中寻找其特定位置更多的信息。
利用X射线晶体学来确定蛋白质结构,已经在结构生物化学领域取得了巨大而重大的突破。
虽然研究人员尚未找到治愈艾滋病的方法,但结构生物学极大地提高了他们对艾滋病毒的了解,并在开发治疗这种致命疾病的药物方面发挥了关键作用。艾滋病毒被迅速认定为逆转录病毒,这种病毒的遗传物质不是DNA,而是RNA。在艾滋病毒出现很久之前,世界各地的实验室研究人员就一直在研究逆转录病毒,其中一些逆转录病毒会导致动物患癌。这些科学家追踪了逆转录病毒的生命周期,并确定了病毒用来感染细胞的关键蛋白质。当艾滋病毒被确认为逆转录病毒时,这些研究为艾滋病研究人员提供了一个立即的助力,使他们能够将先前发现的病毒蛋白作为最初的药物靶点。
科学家们还在1989年确定了艾滋病毒蛋白酶的X射线晶体结构,这种病毒酶在艾滋病毒的生命周期中至关重要。制药科学家希望通过阻断这种酶,能够阻止病毒在体内传播。科学家们终于看到了他们的目标酶。通过将结构信息输入计算机模型程序,他们可以旋转酶模型,放大特定原子,分析其化学性质,甚至剥离或改变其部分结构。最重要的是,他们可以使用模型结构作为参考来确定可能阻断酶的分子类型。这种基于结构的药物设计策略有可能缩短传统的药物开发过程中试错过程所花费的数年时间和数百万美元。艾滋病毒蛋白酶的结构揭示了一个至关重要的发现,这种酶由两个相等的半部分组成。对于大多数这种对称分子来说,两个半部分都有一个活性位点,该活性位点执行酶的功能,但艾滋病毒蛋白酶只有一个活性位点位于两个半部分相遇的分子中心。制药科学家可以利用这一特点,通过用一个小分子阻断单个活性位点,理论上可以利用酶的结构形状作为指导,关闭整个酶并阻止病毒在体内的传播。
塞来昔布最初被设计用于治疗骨关节炎和成人类风湿性关节炎,并成为第一个被批准用于治疗一种罕见疾病FAP(家族性腺瘤性息肉病)的药物,该病会导致结肠癌。一个幸运的发现使科学家能够设计出保留非甾体抗炎药的抗炎特性的药物,而不会产生引起溃疡的副作用。通过在分子水平上研究这些药物,研究人员发现非甾体抗炎药会阻断两种密切相关的酶(称为环氧化酶)的作用:COX-1和COX-2。COX-2是在损伤或感染的情况下产生的,它会激活引发炎症和免疫反应的分子。通过阻断COX-2,非甾体抗炎药可以减少由关节炎、头痛和扭伤引起的炎症和疼痛。相反,COX-1会产生一种叫做前列腺素的分子,这种分子可以保护胃壁免受消化酸的侵蚀,因此当非甾体抗炎药阻断这一功能时,会导致溃疡。
为了创造一种不会引起溃疡的有效止痛药,科学家们意识到他们需要开发出新的药物,这些药物可以关闭COX-2,但不会关闭COX-1。通过结构生物学,他们可以确切地看到为什么塞来昔布会堵塞COX-2,而不会堵塞COX-1。COX-2和COX-1的三维结构几乎完全相同,除了COX-2活性位点中有一个氨基酸的改变,它会形成一个额外的结合口袋。正是这个额外的口袋可以与塞来昔布结合。除了向研究人员展示药物与其靶标结合的原子细节外,COX酶的结构也将继续为基础研究人员提供这些分子在体内如何发挥作用的见解。
X射线晶体学的一些缺点是,样品需要处于固态,样品必须以足够大的数量存在才能进行研究,而且样品经常会被用于研究它的X射线辐射破坏。这意味着气态或液态的任何物质都无法通过X射线晶体学进行分析。此外,稀有或难以合成的样品可能难以研究,因为可能没有足够的样品供辐射提供清晰的图像。第三,研究生物样品可能会有问题,因为用于研究样品的辐射很可能会损害或破坏活组织。
还需要考虑到,X射线晶体学对单个蛋白质结构的分析需要大量时间。每个步骤的大致时间估计如下:在完美执行和99%纯化的情况下,克隆和纯化蛋白质结构需要3-6个月。结晶过程可能需要1-12个月,这取决于蛋白质的物理特性,而这些特性完全取决于诱导晶体形成的有利焓值和晶体形成所处的溶剂。此外,对结晶蛋白质结构进行数据收集可能需要一个月的时间。最后,对溶液中的结构进行相位确定可能需要3个月的时间。综上所述,该过程并不快,这意味着当占用很长时间并使用各种实验室设备时,会产生财务问题。
- 要了解更多关于蛋白质X射线晶体学的信息,请参见Drenth, Jan: 蛋白质X射线晶体学原理 第三版。
- ↑ 美国卫生与公众服务部。生命的结构。2007年7月。<http://www.nigms.nih.gov>.
- ↑ Viadiu, Hector. "为什么我们需要晶体?" UCSD讲座。2011年11月。
- ↑ Kraut, Joseph. "酶是如何工作的?", 科学, 第242卷, 1988年10月28日, 第534页
- ↑ 美国国立卫生研究院, 生命的结构, 2007年, 第37-38, 40, 44页。
- ↑ 美国国立卫生研究院,“设计药物”, 2006年, 第31页。
- ↑ 美国国立卫生研究院,“设计药物”, 2006年, 第25-27页。
- ↑ 美国卫生与公众服务部。生命的结构。2007年7月。<http://www.nigms.nih.gov>.
- ↑ 美国卫生与公众服务部。生命的结构。2007年7月。<http://www.nigms.nih.gov>.
Berg, Jeremy M., John L. Tymoczko, 和 Lubert Stryer. 生物化学。第6版。纽约:W. H. Freeman and, 2006. 印刷版。









