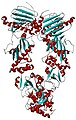
结构生物化学/蛋白质/蛋白质折叠
蛋白质折叠是一个多肽折叠成特定、稳定、功能性、三维结构的过程。它是蛋白质结构获得其功能性形状或构象的过程。
蛋白质由长链的 氨基酸 构成;它们存在于各种不同的结构中,这些结构通常决定着它们的功能。蛋白质遵循能量有利的途径来形成稳定的、有序的结构;这被称为蛋白质的天然结构。大多数蛋白质只有在折叠后才能执行其各种功能。蛋白质的折叠途径或机制是蛋白质为了达到其天然结构而经历的一系列结构变化。蛋白质折叠发生在细胞内高度拥挤、复杂、分子环境中,并且为了避免聚集或错误折叠,通常需要分子伴侣的帮助。蛋白质由各种类型的侧链的氨基酸组成,这些侧链可能是疏水的、亲水的或带电荷的。这些侧链的特征影响了蛋白质将形成的形状,因为它们在分子内和与周围环境之间的相互作用方式不同,从而使某些构象和结构比其他构象和结构更有利。科学家认为折叠蛋白质的指令编码在序列中。研究人员和科学家可以很容易地确定蛋白质的序列,但还没有破解控制折叠的密码(生命结构 8)。
蛋白质折叠理论与实验
[edit | edit source]早期研究 蛋白质组学及其结构的科学家推测,蛋白质具有导致其天然构象的模板。这一理论导致了对蛋白质如何折叠以获得其复杂结构的探索。现在众所周知,在生理条件下,蛋白质通常会自发地折叠成其天然构象。因此,蛋白质的一级结构是宝贵的,因为它决定了蛋白质的三维结构。通常,大多数生物结构不需要外部模板来帮助它们形成,因此被称为 自组装。
蛋白质复性
[edit | edit source]蛋白质复性自 1930 年代就为人所知。然而,直到 1957 年 克里斯蒂安·安芬森 对牛胰 RNase A 进行了一项实验,蛋白质复性才得以量化。RNase A 是一种单链蛋白质,由 124 个残基组成。在 2-巯基乙醇的 8M 尿素溶液中,RNase A 完全展开,其四个二硫键通过还原被裂解。通过对尿素进行 透析并将溶液引入 pH 8 的 O2 中,酶促活性蛋白在物理上无法从 RNase A 中识别出来。因此,该实验表明蛋白质自发地复性。
RNase A 复性的一个标准是其四个二硫键必须重新形成。RNase A 中的八个 Cys 残基之一与它的天然残基形成二硫键的可能性与其另外七个 Cys 残基相比是 1/7。此外,剩余的六个 Cys 残基中下一个随机形成下一个二硫键的可能性是 1/5,等等。因此,RNase A 随机重新形成四个天然二硫键的概率是 (1/7 * 1/5 * 1/3 * 1/1 = 1/105)。这个概率的结果表明,RNase A 中二硫键的形成不是随机的活动。
当 RNase A 重新氧化利用 8M 尿素时,允许二硫键在多肽链是无规卷曲时重新形成,那么当去除尿素后,RNase A 只有大约 1% 的酶促活性。然而,通过使用 2-巯基乙醇,当二硫键交换反应发生并且蛋白质回到其天然状态时,蛋白质可以再次变得完全活跃。RNase A 的天然状态在生理条件下是热力学稳定的,特别是由于比天然状态更稳定的蛋白质需要更大的 活化能垒,并且动力学上无法获得。
通过使用酶 蛋白质二硫键异构酶 (PDI),随机化的 RNase A 所需的时间缩短到大约 2 分钟。这种酶有助于促进二硫键交换反应。为了使 PDI 能够发挥作用,它的两个活性位点 Cys 残基需要处于 -SH 形式。此外,PDI 帮助蛋白质在获得热力学有利构象时随机裂解和重新形成二硫键。
翻译后修饰的蛋白质可能无法复性
[edit | edit source]处于“混乱”状态的蛋白质会通过蛋白二硫键异构酶 (PDI) 重新折叠,而处于天然状态的蛋白质则不需要 PDI,因为天然蛋白质已经处于稳定的构象。然而,经过翻译后修饰的蛋白质需要二硫键来稳定其相对不稳定的天然形式。例如,胰岛素 是一种多肽激素。这种由 51 个残基组成的多肽具有两个二硫键,被 PDI 失活。以下链接显示了带有两个二硫键的胰岛素图像。通过观察这种现象,科学家们发现胰岛素是由胰岛素原 生成的,胰岛素原是一种含有 84 个残基的单链。这个链接 提供了更多关于胰岛素原结构及其转化为胰岛素过程的信息。胰岛素原的二硫键需要在通过蛋白水解切割 其内部的 33 个残基的 C 链之前保持完整。然而,根据两项发现,C 链不是决定 A 和 B 链折叠的关键,而是将它们连接在一起,以促进二硫键的形成。一方面,在合适的重新折叠条件下,混乱的胰岛素能够以 30% 的产率恢复到其天然形式。如果 A 和 B 链进行交联,这种产率可以提高。另一方面,通过分析来自许多物种的胰岛素原序列,发现 C 链的突变率是 A 和 B 链的八倍。
蛋白质折叠的决定因素
[edit | edit source]有各种相互作用有助于稳定天然蛋白质的结构。具体而言,重要的是要研究形成蛋白质结构的相互作用是如何组织的。此外,只有少量的多肽序列可以形成稳定的构象。因此,很明显,在生物系统中,特定的序列是通过进化而被使用的。
螺旋和片层在蛋白质中占主导地位,因为它们有效地填充空间
[edit | edit source]平均而言,大约 60% 的蛋白质含有大量的α-螺旋 和β-折叠片。通过疏水相互作用,蛋白质能够形成致密的非极性核心,但它们没有能力指定哪些多肽被限制在特定的构象中。如卷曲形式的多肽片段中所见,氢键的数量并不比α-螺旋和β-折叠片少。这一观察结果表明,多肽的不同构象形式不受氢键需求的限制。 Ken Dill 提出,螺旋和片层的出现是由于凝聚聚合物中空间位阻 造成的。通过实验和对简单柔性链的构象进行模拟,可以确定β-折叠片和α-螺旋的比例随着链的复杂程度的增加而增加。因此,可以得出结论,螺旋和片层在蛋白质的复杂结构中很重要,因为它们在蛋白质折叠中是致密的。不同力的耦合,例如氢键、离子配对和范德华相互作用,进一步促进了α-螺旋和β-折叠片的形成。
蛋白质折叠受内部残基的指导
[edit | edit source]通过研究蛋白质修饰,可以确定不同类别的氨基酸残基在蛋白质折叠中的作用。例如,在一项特定研究中,RNase A 的游离伯氨基被衍生化,与由 8 个残基组成的多聚-DL-丙氨酸链结合。多聚-丙氨酸链很大且水溶性,因此可以使 RNase 的 11 个游离氨基连接起来,而不会干扰蛋白质的天然结构或其重新折叠的能力。因此,可以得出结论,蛋白质的内部残基促进了其天然构象,因为 RNase A 的游离氨基位于外部。此外,研究表明,发生在残基表面的突变很常见,并且与发生在内部残基的改变相比,更不可能改变蛋白质的构象。这一发现表明,蛋白质折叠主要是由疏水力 驱动的。
蛋白质结构是按等级组织的
[edit | edit source]George Rose 证明了蛋白质结构域由亚结构域组成,并且进一步具有亚亚结构域等等。因此,很明显,大型蛋白质具有连续的、致密的、物理上可分离的结构域。当将天然蛋白质中的多肽片段可视化为一条带有许多缠结的绳子时,在将绳子切成两段时可以看到一个平面。当用蓝色和红色突出显示 n 个残基结构域中的 n/2 个残基时,可以重复此过程。随着此过程的重复,可以看出,在所有阶段,蛋白质的红色和蓝色区域都不会相互渗透。以下链接 显示了 HiPIP(高电位铁蛋白)的 X 射线结构及其前 n/2 个残基在 n 个残基蛋白质上的颜色分别为红色和蓝色。此外,第二行和第三行中显示的后续结构显示了如所示的 n/2 残基分裂的重复过程,其中蛋白质的左侧具有其第一个和最后一个一半,分别为红色和蓝色,而链的其余部分为灰色。通过这个例子,可以清楚地看到蛋白质结构是按等级组织的,这意味着多肽链被视为亚结构域,它们本身是致密的结构,并与相邻的结构相互作用。这些相互作用形成了一个更大的井然有序的结构,这主要是由于氢键相互作用,并且在理解多肽 如何折叠形成其天然结构方面起着重要的作用。
蛋白质结构是可适应的
[edit | edit source]由于侧链 在球状蛋白质 内部以高度互补的方式相互配合,因此其堆积密度几乎与有机晶体相同。因此,为了确认这种高堆积密度现象是否是影响蛋白质结构的重要因素,Eaton Lattman 与 George Rose 试图验证球状蛋白质中是否存在侧链之间的相互作用。他们分析了 67 种经过充分研究的球状蛋白质结构,并得出结论,没有发现偏好的相互作用。该实验表明,堆积不是决定天然折叠的原因,而是天然折叠是球状蛋白质堆积的必要条件。这一观点可以得到进一步的支持,因为蛋白质家族的成员即使没有序列相似性和远缘关系,也会产生相同的折叠。
此外,结构实验数据表明,蛋白质内部残基以有效的方式紧密结合在一起有各种方式。布莱恩·马修斯 基于T4溶菌酶(由 噬菌体T4 产生)进行了一项广泛的研究,结果发现T4溶菌酶残基的变化只会影响局部位移,不会导致任何全局结构变化。以下 链接 提供了T4溶菌酶的X射线视图和对其结构的简要生化描述。马修斯研究了164个残基的T4溶菌酶的超过300种不同突变体,并将它们进行比较。此外,还观察到T4溶菌酶可以承受大约4个残基的插入,同时仍然不会对整体蛋白质结构或酶活性产生任何重大结构变化。此外,通过使用测定技术表明,在进行的2015个单残基替换中,只有173个T4突变体表现出酶活性显着降低。通过这些实验,可以明显看出蛋白质结构具有极强的耐受性。
莱文萨尔悖论
[edit | edit source]莱文萨尔悖论是一个思想实验,也是蛋白质折叠理论中的自指。1969年,赛勒斯·莱文萨尔注意到,由于展开的多肽链具有非常多的自由度,因此该分子具有天文数量的可能构象。在他的一篇论文中估计了3300 或10143 个构象。
莱文萨尔悖论观察到,如果蛋白质通过依次采样所有可能的构象来折叠,那么即使以快速速率采样构象,也需要花费大量时间才能完成折叠。基于蛋白质折叠速度远快于此的观察结果,莱文萨尔随后提出,不会发生随机构象搜索,因此蛋白质必须通过一系列亚稳态中间状态来折叠。
1969年,赛勒斯·莱文萨尔计算出,如果蛋白质要从展开状态到天然状态折叠时随机采样所有可能的构象,即使蛋白质每秒达到1000亿个构象,也将需要天文时间。观察到蛋白质在相对较短的时间内折叠,莱文萨尔提出蛋白质以固定且定向的过程折叠。我们现在知道,虽然蛋白质折叠不是一个随机过程,但似乎并没有单一的固定蛋白质折叠途径。这一观察被称为莱文萨尔悖论。这个悖论清楚地表明,蛋白质不会通过尝试所有可能的构象来折叠。相反,它们必须遵循至少部分定义的折叠路径,该路径由完全变性蛋白与其天然结构之间的中间体组成。
累积选择
[edit | edit source]摆脱莱文萨尔悖论的关键在于认识到累积选择。理查德·道金斯曾问,一只猴子随机敲击打字机需要多长时间才能打出“Methinks it is like a weasel”,这是哈姆雷特对波洛尼乌斯的一句台词。需要大量击键,约为1040 个。然而,如果我们假设每个正确的字符都保留下来,只让猴子重新打出错误的字符,平均只需要几千次击键。这两种情况之间的关键区别在于,第一种情况使用完全随机搜索,而第二种情况保留了部分正确的中间体。这也表明蛋白质折叠的本质是保留部分正确的中间体,尽管蛋白质折叠问题比莎士比亚例子中提出的问题要困难得多。
成核-凝聚模型
[edit | edit source]为了正确理解蛋白质折叠问题,我们必须考虑蛋白质的某些特征。由于蛋白质只有轻微的稳定性,因此典型1000个残基的蛋白质的折叠状态和展开状态之间的自由能差为42 kJ mol−1,因此每个残基平均只贡献0.42 kJ mol−1 的能量来维持折叠状态。这个能量值小于室温下的热能,即2.5 kJ mol−1。这种微不足道的稳定能量意味着正确的中间体,尤其是折叠早期形成的中间体,可能会丢失。然而,导致协同折叠的相互作用可以随着结构的建立而稳定中间体。因此,具有显着结构偏好的局部区域,虽然它们本身可能不稳定,但往往会采用其偏好的结构,并且随着它们的形成,可以相互作用,导致稳定性的提高。成核-凝聚模型指的是解决蛋白质折叠挑战的这个概念框架。
分子内相互作用在折叠机制中的作用
[edit | edit source]蛋白质 折叠形成能量上有利的结构,这些结构由 疏水相互作用 聚集、 氢键 和 范德华力 在 氨基酸 之间的相互作用所稳定。蛋白质折叠首先形成二级结构,如α螺旋、β折叠和环。不同的氨基酸具有不同的倾向,它们会根据氨基酸的极性和旋转势垒而形成α螺旋、β折叠或β转角。例如,缬氨酸、苏氨酸、异亮氨酸等氨基酸由于空间位阻会导致α螺旋不稳定。因此,它们更倾向于向β折叠而不是α螺旋发生构象变化。氨基酸在二级结构中的相对频率根据它们对α螺旋、β折叠或转角的偏好进行分组(表1)。 表1:氨基酸残基在二级结构中的相对频率 这些结构反过来又折叠形成三级结构,并通过形成分子内氢键来稳定。 共价 键也可能在折叠成三级结构期间通过形成二硫键或金属簇而发生。根据罗伯特·佩恩的“蛋白质折叠机制”,分子也经常会通过形成疏水坍塌(其中所有疏水侧链突然滑入蛋白质内部或聚集在一起)形成的中间“熔融球状体”状态,然后达到其天然构象。然而,这意味着所有主链NH和CO基团都被埋藏在非极性环境中,但它们更喜欢水性环境,因此二级结构必须很好地配合在一起,以便通过氢键和 范德华力 相互作用的稳定性超过它们的亲水性倾向。氢键在蛋白质中的强度会根据它们在结构中的位置而变化;在疏水核心形成的氢键比暴露于水性环境中的氢键对天然状态的稳定性贡献更大。
水溶性蛋白质折叠成具有非极性疏水核心的紧凑结构。蛋白质内部包含中心非极性残基(例如-亮氨酸、缬氨酸、甲硫氨酸和苯丙氨酸),而外部主要包含极性带电残基(例如-天冬氨酸、谷氨酸、赖氨酸和精氨酸)。这样,极性带电分子就可以与周围的水分子相互作用,而疏水分子可以免受水性环境的包围。最小化结构外部的疏水侧链数量使蛋白质结构在热力学上更有利,因为疏水分子更喜欢在水性环境中聚集在一起(例如-疏水效应)。跨越生物膜的蛋白质(例如-孔蛋白)具有与水溶性天然结构相反的内外分布,它们具有疏水残基覆盖的外部表面,中心充满水,衬里是带电和极性氨基酸。
膜蛋白的折叠
[edit | edit source]在“折叠场景调查:膜蛋白”这篇文章中,作者帕梅拉·J·布斯和保罗·库尔诺试图回答具有α螺旋结构的跨膜蛋白的折叠机制是如何运作的。研究膜蛋白的折叠一直很困难,因为这些蛋白通常很大,并且由多个亚基组成。这些蛋白具有高度的构象灵活性,这对它们在细胞中发挥作用是必要的。此外,这些蛋白既有疏水表面,面向膜,也有亲水表面,面向膜两侧的水性区域。这些蛋白在侧向移动,并共享其嵌入的脂质双层的弹性特性。为了研究这些蛋白,布斯和库尔诺认为必须操纵脂质双层,并结合动力学和热力学研究方法。
可逆折叠和线性自由能 蛋白质折叠的自由能通过可逆化学变性来衡量。蛋白质的可逆折叠取决于这种自由能。对于正在研究的α螺旋蛋白质,已经证明它们遵循可逆的二态过程。bR(一种称为细菌视紫红质的α螺旋膜蛋白)在将SDS(一种变性剂,也是一种阴离子去垢剂)添加到混合脂质、去垢剂胶束中时会可逆地展开。该二态反应涉及部分展开的SDS状态和折叠的bR状态。通过比较展开和折叠速率的对数以及SDS摩尔分数,产生了线性图,证明了线性关系。该图证明bR在膜外具有非常高的稳定性,证明它出乎意料地稳定。此外,bR在膜外非常稳定,在没有添加变性剂的情况下,它不会在合理的时间段内展开。
与水溶性蛋白质的比较 Booth和Curnow研究了三种信息量最多的膜蛋白:bR、DGK(大肠杆菌二酰基甘油激酶)和KcsA(链霉菌利迪亚斯钾通道)。这三种膜蛋白与水溶性蛋白质(通过2或3态动力学折叠)进行了比较。在没有变性剂的情况下,展开的总自由能变化对于水溶性蛋白质和类似大小的膜蛋白是相同的。这证明决定蛋白质稳定性的不是力的类型,而是弱力的平衡。事实证明,膜蛋白中的氢键与水溶性蛋白质中的氢键强度相似,而不是像预期的那样在膜蛋白中更强。
机械强度和在施加力下的展开 动态力显微镜可用于测量蛋白质特定区域在施加力下的机械响应。在这种情况下,展开力取决于活化能垒。这种展开与蛋白质的热力学稳定性无关。对于在施加力下的展开,膜蛋白(尤其是bR)似乎遵循哈蒙德行为规则。该反应两个连续状态之间的能量差减小,状态在结构上变得相似。
周围膜的影响 膜蛋白受其周围膜的影响很大。如果脂质在去垢剂胶束中掺入,从而增加脂质结构的稳定性,则蛋白质及其折叠都将被稳定。不同脂质的不同组合会导致膜蛋白的不同稳定性或折叠。膜的大小也会影响膜蛋白。不同类型的脂质会导致不同的膜特性。一种称为PE脂质的脂质比第二种称为PC脂质的脂质具有更高的自发曲率。通过将PE脂质添加到PC脂质中,双层的单层曲率增加。增加脂质双层的曲率会增加蛋白质折叠的稳定性。
蛋白质在生物膜中的转运
[edit | edit source]在线粒体中,由核糖体制造的蛋白质直接从胞质溶胶中吸收。线粒体蛋白首先在胞质溶胶中完全合成,作为线粒体前体蛋白,然后被摄取到膜中。线粒体蛋白在其N端含有特定的信号序列。这些信号序列通常在进入膜后被去除,但进入具有外膜、内膜、膜间空间的膜的蛋白质具有内部序列,这些序列在内膜内的转运中起主要作用。
蛋白质转运在将蛋白质转运穿过线粒体膜中起主要作用。在外膜和内膜中发现了四个主要的多分支蛋白复合物。TOM复合物位于外膜,两种类型的TIM复合物整合在内膜中:TIM23和TIM22。这些复合物充当线粒体前体蛋白的受体。
TOM:导入所有细胞核编码的蛋白质。它主要启动信号序列向膜间空间的转运,并将跨膜蛋白插入外膜空间。然后,一个称为SAM复合体的β桶复合物负责在外膜中正确折叠蛋白质。TIM23位于内膜,调节可溶性蛋白质插入基质,并促进跨膜蛋白质插入内膜。TIM23是另一个内膜复合物,促进由转运蛋白组成的内膜蛋白的插入,这些转运蛋白在线粒体膜之间转运ADP、ATP和磷酸盐。OXA是另一个内膜复合物,帮助插入线粒体本身合成的内膜蛋白,以及插入首先被转运到基质空间的内膜蛋白。File:Translocation.jpg
在核糖体上折叠
[edit | edit source]蛋白质链开始折叠的地方是一个被广泛研究的主题。当新生链穿过核糖体的“出口隧道”进入细胞环境时,链何时开始折叠?将讨论核糖体隧道中协同翻译折叠的概念。蛋白质的新生链通过其C端与肽基转移酶中心(PTC)结合,并将以矢量方式出现。隧道非常狭窄,对新生链施加了一定的刚性,随着每个氨基酸的添加,蛋白质的构象空间增加。协同翻译折叠可以帮助通过帮助蛋白质在仍然位于核糖体隧道中时获得显着水平的天然状态来大大减少可能的构象空间。蛋白质的长度也可以很好地估计其三维结构。较小的链倾向于有利于β折叠,而较长的链(如达到119/153个残基的链)倾向于有利于α螺旋。
核糖体隧道长度超过80Å,宽度约为10-20Å。在隧道内部,有辅助分子,如L23、L22和L4蛋白质,它们与新生链相互作用,帮助折叠。隧道也具有亲水性,并帮助新生链穿过它,不会受到阻碍。尽管很硬,但隧道不是被动的导管,但它是否具有促进蛋白质折叠的能力尚不清楚。最近一项涉及冷冻电镜的实验表明,隧道中存在折叠区。在出口口(距PTC约80Å),新生链已采用优选的低阶构象。这强化了链可以在某些区域具有折叠程度的建议。尽管可以发生一些低阶折叠,但天然状态的采用发生在隧道外,但并不一定是在新生链被释放时。结合的新生链(RNC)采用部分折叠结构,在拥挤的细胞环境中,这会导致链自缔合。然而,这种自缔合通过沿着出口隧道排列的交错核糖体得到缓解,这最大限度地增加了RNC之间的距离。
生成用于研究的RNC
一种生成RNC并拍摄其从隧道中出现的快照的技术是停止翻译。使用没有终止序列的截短的DNA。这允许新生链保持绑定,直到需要为止。为了确定链的残基,可以通过碳-13或氮-15对其进行标记,然后通过核磁共振波谱检测。另一种技术是PURE方法,它包含翻译所需的最小成分。这种方法已被用于研究链与辅助分子(如TF伴侣)之间的相互作用。该方法与石英晶体微天平技术相结合,用于通过质量分析合成。生成RNC链的体内技术可以通过在高细胞密度下刺激它来完成。这最初是在未标记的环境中完成的,然后将细胞转移到标记的培养基中。RNC由SecM生成。通过亲和层析纯化RNC,并通过SDS-PAGE或免疫印迹检测。
通过生成RNC,可以进行许多实验来更多地研究出现的新生链。如上所述,链以矢量方式从出口隧道中出现。这使链能够对天然折叠进行采样,并增加了折叠到天然状态的概率。除了这种矢量折叠之外,伴侣还有助于有利的折叠速率和正确的折叠。
内质网中的蛋白质折叠
[edit | edit source]蛋白质进入哺乳动物内质网:内质网(ER)是蛋白质成熟的主要检查点,以确保只有正确折叠的蛋白质被分泌并递送到作用部位。蛋白质进入内质网始于识别N'端信号序列。特别地,这种序列被信号识别蛋白(SRP)检测到,导致核糖体/新生链/SRP复合物与内质网膜结合。然后,该复合物通过一个称为Sec61转运蛋白的蛋白质孔,该孔允许多肽链进入内质网的腔室部分。
蛋白质折叠过程中相互冲突的过程:蛋白质进入内质网后,蛋白质分解成折叠中间体的集合。这些中间体采取三种不同的路线。它们要么被正确折叠并被发送到从内质网(ER)输出到胞质溶胶中,要么被聚集起来,要么被挑选出来降解。这三个过程在竞争中正确分泌蛋白质。为了使蛋白质被正确分泌,折叠、聚集和降解之间的竞争必须有利于折叠,以便折叠比其他过程更快地发生。这种平衡被称为蛋白稳态。可以通过使用更小的分子来稳定蛋白质(称为辅因子)或增加折叠因子的浓度来将蛋白稳态的平衡倾斜为有利于折叠。这种控制蛋白稳态的能力使科学家能够克服一些蛋白质折叠疾病,如囊性纤维化。
正确折叠的蛋白质已准备好进行顺行转运,并通过内质网膜分泌到胞质溶胶中,方法是通过识别正确折叠的蛋白质的货物受体。折叠不正确的蛋白质不会被分泌,要么被靶向降解,要么被聚集。聚集的蛋白质能够重新进入蛋白质集合的阶段,准备折叠,以便它们可以再次尝试正确折叠。

内质网中的折叠因子
关于折叠途径的生化研究提供了一个关于内质网中蛋白质折叠所涉及的折叠因子或伴侣的综合清单。折叠因子根据它们是否催化某些步骤或它们是否与折叠途径中的中间体相互作用进行分类。通常将一般的蛋白质折叠因子分为四组:热休克蛋白作为伴侣或伴侣蛋白、肽酰脯氨酰顺式/反式异构酶(PPIases)、氧化还原酶和糖基结合蛋白。
许多折叠因子之所以很棒是因为它们具有多功能性。一个折叠因子可以处理折叠途径的不同区域。不幸的是,这会导致冗余,因为不同类型的蛋白质执行重叠的功能。这种功能冗余使得难以理解单个折叠因子在帮助客户蛋白成熟中的具体作用。折叠因子也倾向于在成熟过程中协同作用,这进一步模糊了每个因子的个体作用。由于这些作用尚不清楚,因此很难确认即使一个折叠因子处理了一个蛋白质中的特定反应,该折叠因子也会在另一个蛋白质中执行相同的函数。
除了帮助蛋白质的非共价折叠和展开外,内质网中的折叠因子有时会延迟与蛋白质的相互作用。这为新生蛋白质正确折叠提供了时间,并使折叠的蛋白质能够在其折叠途径上回溯,这延长了未折叠状态的平衡,防止蛋白质被保持在非天然状态。
内质网后的折叠:虽然内质网只提供正确组装的蛋白质以分泌,但也有一些例子表明蛋白质在高尔基体和更远的地方改变构象。通常,新折叠的蛋白质在内质网中很敏感且易于展开,但在退出后抗展开。在没有伴侣蛋白和其他折叠酶的环境中,蛋白质是紧凑的,并且在退出内质网后对改变相对抵抗。然而,这并不一定意味着蛋白质折叠结束,因为一些分子伴侣蛋白如Hsp 70s和Hsp 90s在蛋白质存在的整个过程中继续帮助蛋白质构象。

研究蛋白质折叠的一种策略是在高浓度的化学变性剂(如盐酸胍)中展开蛋白质分子。然后迅速稀释溶液,直到变性剂浓度降低到使天然状态再次热力学稳定。之后,可以观察蛋白质折叠的结构变化。理论上,这听起来很简单。然而,此类实验很复杂,因为展开的蛋白质在化学变性剂中具有无规卷曲状态。此外,分析样品中发生的结构变化可能很困难,因为所有分子可能具有显著不同的构象,直到反应的最后阶段。因此,分析必须在几秒钟内完成,而不是通常允许推断单个天然蛋白质构象结构的天数或数周。为了避免这个问题,可以在蛋白质展开后还原二硫键,并在氧化条件下重新形成。然后,可以通过质谱等标准技术识别蛋白质,以得出有关二硫键形成时的折叠阶段存在的结构的结论。
多种技术用于监测重折叠过程中的结构变化。例如,在圆二色性中,远距离的紫外光用于提供折叠过程中二级结构出现的测量。近距离的紫外光监测芳香残基紧密堆积环境的形成。 核磁共振也是一种有用的技术,可以表征单个氨基酸残基水平的构象。它还可以用于监测结构的形成如何保护酰胺氢免受溶剂交换的影响。
圆二色性:这种类型的光谱法测量圆偏振光的吸收,因为蛋白质的结构,例如α螺旋和β折叠,是手性的,可以吸收这种光。光的吸收指示蛋白质折叠程度。这种技术还可以通过测量吸光度随变性剂浓度或温度的变化来测量蛋白质的平衡展开。变性剂熔解测量展开的自由能,而温度熔解测量蛋白质的熔点。这种技术是研究蛋白质折叠最普遍和最基本的方法。
双偏振干涉测量法:这种技术使用限制在波导中的激光束的倏逝波来探测已吸收到波导表面的蛋白质层。激光光聚焦在两个波导上,一个感知光束并具有暴露的表面,另一个用于创建参考光束并激发波导的偏振模式。对干涉图的测量可以帮助计算蛋白质密度或折叠,吸收层的尺寸,并推断关于亚原子分辨率的分子相互作用的结构信息。当通过两个波导的光结合时,在远场中获得二维模式。
质谱法:使用 质谱法 研究蛋白质折叠的优势包括能够检测具有不同氘含量的分子,这使得能够研究蛋白质折叠反应的异质性。它还可以测量与分子伴侣蛋白结合的折叠中间体的构象,而不会破坏复合物。质谱法还可以直接比较重折叠特性,因为如果两种蛋白质的分子量足够不同,则可以在没有分离的情况下研究蛋白质混合物。
高时间分辨率:这些是快速时间分辨技术,其中展开的蛋白质样品被触发快速折叠。然后研究产生的动力学。实现这一点的方法包括快速混合溶液、光化学方法和激光温度跳跃光谱法。
蛋白质三级结构的计算预测:这是一种与蛋白质折叠相关的蛋白质结构分析的不同形式。这些程序可以模拟冗长的折叠过程,提供统计势的信息,并再现折叠途径。
蛋白质错误折叠是指蛋白质未能有效地实现其紧密堆积的天然构象,或者由于环境变化或突变导致稳定性降低而未能维持该构象。已经确定,蛋白质折叠失败是在高温和其他压力情况下的一种普遍现象。错误折叠蛋白质的两个最常见结果是降解和聚集。当多肽从细胞中出现时,它可能会折叠成天然状态、被蛋白水解降解或与其他分子形成聚集体。蛋白质处于不断的动态平衡状态,因此即使折叠过程已完成,在细胞环境中也会发生展开。展开的蛋白质通常会重新折叠回其天然状态,但如果控制过程失败,错误折叠会导致细胞功能障碍,并最终导致疾病。与错误折叠相关的疾病涵盖多种病理状况,例如囊性纤维化,其中编码结果的基因发生突变,导致折叠成一种构象,其分泌被细胞中的质量控制机制阻止。大约 50% 的癌症与 p53 蛋白的突变有关,最终导致细胞周期控制的丧失,并导致肿瘤生长。蛋白质无法保持折叠会导致聚集,这是被称为淀粉样蛋白沉积症的一组遗传性、散发性和感染性疾病的常见特征。聚集通常导致无序的物质,可以在生物体中降解,但它也可能导致在组织中积累的高度不溶性纤维。已知大约有 20 种疾病是由淀粉样蛋白物质的形成引起的,包括阿尔茨海默病、II 型糖尿病和帕金森病。淀粉样蛋白纤维是有序的蛋白质聚集体,由于分子间氢键具有广泛的β折叠结构,并且与它们衍生的蛋白质具有总体相似的外观。淀粉样蛋白纤维的形成是由于长时间暴露于至少部分变性的条件下。
阿尔茨海默病:这种神经退行性疾病是由 斑块 和 缠结 在大脑神经细胞中积累引起的。[1] 斑块几乎完全由一种蛋白质组成,是β-淀粉样蛋白在神经细胞间隙之间聚集,而缠结是tau蛋白在神经细胞内部聚集。缠结在广泛的神经细胞疾病中很常见,而神经炎斑块更特异于阿尔茨海默病。尽管科学家不确定斑块和缠结在阿尔茨海默病形成中起什么作用,但一种理论是,这些积累的蛋白质阻碍了神经细胞彼此通信的能力,并使其难以存活。研究表明,斑块和缠结在人们衰老时自然发生,但在患有阿尔茨海默病的人群中观察到更多。这种增加的原因尚不清楚。
克雅氏病(疯牛病):这种疾病是由称为朊病毒的异常蛋白质引起的,它们会侵蚀大脑并形成洞状病变。朊病毒(proteinaceous infectious virion)被发现是具有改变构象的蛋白质。科学家推测,这些感染性因子可以与其他类似蛋白质结合,并诱导它们的构象发生改变,从而传播新的感染性蛋白质。[2] 朊病毒对热、紫外线和辐射高度耐受,这使得它们难以消除。在克雅氏病中,潜伏期长达数年,然后迅速发展为抑郁、行走困难、痴呆和死亡。目前还没有针对朊病毒病的有效治疗方法,所有朊病毒病都是致命的。[3]
帕金森病:编码α-突触核蛋白的基因发生突变是某些罕见家族性帕金森病的病因。迄今已发现三种点突变:A53T、A30P 和 E46K。此外,该基因的重复和三联体重复也可能是其他帕金森病血统的病因。帕金森病患者的主要症状是由于基底神经节对运动皮层的刺激减少,而基底神经节的刺激通常是由多巴胺的不足产生和作用引起的。多巴胺是在大脑的多巴胺能神经元中产生的。患有这种疾病的人会发生脑细胞丢失(多巴胺能神经元死亡),这可能是由异常积累的蛋白质α-突触核蛋白与受损细胞中的泛素结合造成的。这使得α-突触核蛋白-泛素复合物无法被引导到蛋白酶体。新的研究表明,内质网和高尔基体之间蛋白质的错误运输可能是α-突触核蛋白导致多巴胺能神经元丢失的原因。
囊性纤维化:弗朗西斯·柯林斯于 1989 年首次发现了这种遗传性基因突变。问题出现在囊性纤维化跨膜传导调节因子 (CFTR)中,该调节因子调节盐含量并防止细菌生长,当 CFTR 的解离被扰乱时,CFTR 作为一种蛋白质调节跨细胞膜的氯离子转运。[4] 缺失的氨基酸无法杀死肺部的细菌,从而导致慢性肺部感染,最终导致早逝。[5] 科学家已经使用核磁共振波谱 (NMR)来研究囊性纤维化及其影响。
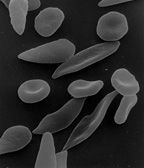
镰状细胞贫血:镰刀状红血球粘附在狭窄的血管壁上,阻碍血液流动,这就是镰状细胞贫血的定义。血液中红血球数量不足以及携氧血液缺乏会导致严重的医疗问题。通过检测两个缺陷的遗传基因的存在,可以检测到血红蛋白基因的缺陷。当血红蛋白释放氧气时,就会形成镰刀状形状,从而导致僵硬的红血球形成杆状结构。一些症状包括:疲劳、呼吸急促、任何关节或身体器官的疼痛持续时间不等、眼部问题可能导致失明、皮肤和眼睛发黄,这是由于红血球快速分解造成的。幸运的是,镰状细胞贫血可以通过简单的血红蛋白电泳血液测试检测出来。尽管目前还没有治愈方法,但输血、口服抗生素和羟基脲可以减轻疼痛。[6]
亨廷顿病:也称为三核苷酸重复病,亨廷顿病是由亨廷顿蛋白中谷氨酰胺重复造成的。大约 40 个或更多个 C-A-G(谷氨酰胺)的重复会导致亨廷顿病,因为正常数量在 10 到 35 个之间。在突变的亨廷顿蛋白 (mHTT) 的翻译后修饰过程中,一小部分多聚谷氨酰胺扩展会错误折叠形成包涵体。包涵体对脑细胞有毒。这种亨廷顿蛋白的改变没有明确的影响,只是它影响神经细胞的功能。[7] 这种无法治愈的疾病影响肌肉协调和一些认知功能。
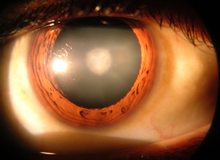
白内障:眼晶状体由称为晶状体蛋白的蛋白质组成。晶状体蛋白在晶状体细胞质中具有果冻状的质地。白内障是目前世界上导致失明的主要原因,当晶状体蛋白分子形成聚集体,散射可见光,导致眼晶状体变得浑浊时就会发生白内障。紫外线和氧化剂被认为会导致白内障,因为它们可能会改变晶状体蛋白的化学结构。在儿童中,人们观察到 αB-晶状体蛋白的缺失或突变会促进白内障的形成。随着年龄的增长,患白内障的可能性呈指数级增长。Pain, Roger H. (2000). 蛋白质折叠机制. 牛津大学出版社. pp. 420–421. ISBN 019963788. Retrieved 2009-10-18. {{cite book}}: Check |isbn= value: length (help)
淀粉样蛋白纤维。
[edit | edit source]蛋白质错误折叠是由折叠效率下降引起的,这会导致可用于执行其正常作用的蛋白质数量减少,并形成淀粉样蛋白纤维,即聚集的蛋白质结构,形成交叉β结构,可以产生许多生物学功能。蛋白质聚集可以来自翻译后发生的各种过程,包括内质网 (ER) 质量控制系统中降解可能性增加、蛋白质运输不当,或特定肽和蛋白质从其可溶性功能状态转换为其高度组织化的聚集纤维。
结构。
X 射线晶体学。
通过 X 射线晶体学,形成了淀粉样蛋白纤维结构的三维晶体,并检查了肽的形成结构以及分子如何堆积在一起。在一个特定的片段中,发现晶体包含平行 β-折叠片的部分,其中每个肽贡献一个单一的 β-链。β-链堆叠在一起,形成的 β-折叠片是平行的,侧链 Asn2、Gln4 和 Asn6 以一种使水从两个 β-折叠片之间的区域排除的方式相互作用,而其余的侧链在外部被水合并远离下一个 β-折叠片。
固态核磁共振 (SSNMR)。
通过固态核磁共振 (SSNMR) 以及其他方法的帮助,例如计算能量最小化、电子顺磁共振以及定点荧光标记和氢氘交换、质谱、有限蛋白水解和脯氨酸扫描诱变,淀粉样蛋白纤维的结构被认为是四个β-折叠片,相隔约 10Å。
通过 NMR 与计算能量最小化,确定在 pH 7.4 和 24 ˚C 下淀粉样β肽的 40 个残基形式为纤维的核心贡献了一对 β-链,该 β-链通过一个蛋白质环连接。淀粉样β肽以平行方式相互堆叠。
从定点自旋标记与电子顺磁共振 (SDSL-EPR) 实验中发现,该分子在纤维中结构非常完整,并且排列平行。SDSL-EPR 以及氢氘交换、质谱、有限蛋白水解和脯氨酸扫描诱变表明,该结构在 N 末端侧具有高灵活性并暴露于溶剂,但在结构的其他部分则很刚性。
通过 SSNMR 与荧光标记和氢氘交换进行的实验确定,C 末端参与了纤维结构的核心,每个分子贡献了四个 β-链,其中链 1 和 3 形成一个 β-折叠片,链 2 和 4 形成另一个 β-折叠片,相隔约 10Å。

使用 SSNMR 技术进行的进一步实验在原子水平上进行了研究,结果在光谱中产生了非常窄的共振线,表明纤维中的分子具有某种程度的均匀性,肽显示出扩展的 β-链与纤维相连。
结论。
通过 X 射线晶体学或 SSNMR 确定的结构与先前从胰岛素形成的冷冻电子显微镜 (EM) 中提出的结构类似。EM 利用电子密度图,揭示了结构中未扭曲的 β 折叠。这些实验中发现的结构相似性表明许多淀粉样纤维可能具有相似的特征,例如侧链堆积、β 链对齐和 β 折叠的分离。 [8] Annu. Rev. Biochem. 2006.75:333-366. www.annualreviews.org. Retrieved 24 Oct 2011</ref>
形成
形成被认为具有遗传性的淀粉样蛋白结构的能力源于发现越来越多蛋白质没有显示与蛋白质相关的疾病迹象。人们发现,淀粉样蛋白可以从具有功能而不是疾病相关特征的自身蛋白质转化而来,存在于活生物体中。
在这些蛋白质突变中,影响淀粉样纤维形成的不同因素和不同的链以不同的速度形成淀粉样纤维。在不同的多肽分子中,疏水性、亲水性、电荷变化、溶剂暴露程度、芳香族侧链数量、表面积和偶极矩都会影响蛋白质的聚集速度。人们发现,蛋白质浓度、溶液的 pH 值和离子强度以及蛋白质所在的氨基酸序列决定了从非结构化、非同源蛋白质序列中聚集的速度。
侧链疏水性的增加或减少会改变蛋白质聚集的倾向。
蛋白质中的电荷可以通过多肽链与周围其他大分子相互作用而产生聚集。此外,β 折叠形成的低倾向性以及 α 螺旋形成的高倾向性也有助于淀粉样蛋白的形成。
人们发现,蛋白质序列暴露于溶剂的程度往往会影响淀粉样蛋白的形成。暴露于溶剂的蛋白质似乎会促进聚集。即使蛋白质的其他部分具有较高的聚集倾向,但并没有参与聚集,它们似乎至少部分未暴露于溶剂,而暴露于溶剂但未参与聚集的其他区域则具有较低的形成淀粉样纤维的倾向。
甚至有人提出,蛋白质序列随着时间的推移而进化,通过交替疏水和亲水区域的模式来避免形成疏水残基簇,从而降低蛋白质聚集发生的倾向。 [8]
序列对淀粉样蛋白形成的影响
淀粉样蛋白的形成主要来自所有肽和蛋白质中相似的多肽链的特性,但有时序列会影响分子构象状态的相对稳定性。在这种情况下,具有不同序列的多肽链会以不同的速度形成淀粉样纤维。序列差异会影响蛋白质聚集的行为,而不是影响蛋白质折叠的稳定性。各种物理化学因素会影响未折叠多肽链淀粉样结构的形成。
侧链的疏水性会影响未折叠多肽链的聚集。当聚集位点区域的氨基酸在突变或折叠位点增加或减少疏水性时,它们会改变序列的聚集能力。随着时间的推移,序列已经进化来避免产生疏水残基团块,通过交替蛋白质的疏水区域。
电荷会影响淀粉样蛋白折叠的聚集。高净电荷可能具有阻碍蛋白质自缔合的可能性。减少正净电荷的突变可能导致与增加正净电荷相反的聚集形成效果。人们发现,多肽链可以通过与高电荷大分子相互作用来运行,这表明蛋白质聚集的电荷很重要。
蛋白质的二级结构也会影响淀粉样蛋白的聚集。研究表明,形成 α 螺旋结构的低概率和形成 β 折叠结构的高概率是淀粉样蛋白形成的促成因素。然而,人们发现,自然界并不特别有利于 β 折叠的形成,因为很少发现亲水和疏水残基序列模式的交替。
氨基酸序列的特征会影响淀粉样纤维结构和聚集速度。据称,不同的突变,包括芳香族侧链数量的变化、暴露表面积和偶极矩的变化,会改变许多多肽链的聚集速度。
未折叠区域在促进部分折叠蛋白质聚集中起着至关重要的作用。人们发现,一些易于弯曲或暴露于溶剂的区域喜欢聚集。其他未参与聚集的区域被发现没有暴露,而是半埋藏,即使它们具有很高的聚集可能性,而结构中暴露且未参与聚集的区域则具有较低的聚集淀粉样纤维的可能性。纤维倾向于通过未折叠多肽片段的缔合而聚集在一起,而不是通过对接结构元素。
总的来说,人们发现,未折叠蛋白质的疏水性低于折叠蛋白质,净电荷高于折叠蛋白质。倾向于不形成 β 折叠结构蛋白质二级结构的残基似乎会抑制淀粉样蛋白聚集的发生。人们发现,蛋白质浓度、pH 值和离子强度与氨基酸序列有关,这会影响聚集速度。
环境影响
[edit | edit source]人们已经了解,蛋白质的一级结构(氨基酸序列)使蛋白质倾向于特定的三维结构,以及它如何从未折叠形式折叠到天然状态。盐浓度、温度、主要溶剂的性质、大分子拥挤和伴侣蛋白的存在都会影响折叠机制以及未折叠蛋白质与天然状态蛋白质的比例。最重要的是,这些环境因素会影响任何单个蛋白质达到正确最终结构的可能性。
放置在适当环境(特定溶剂、溶质浓度、pH 值、温度等)中的孤立蛋白质倾向于“自折叠”成正确的天然构象。改变任何这些环境特征都会破坏结构和/或干扰折叠机制。超出给定蛋白质“正常”范围的 pH 值会使特定氨基酸电离或干扰原本会稳定结构的极性和偶极-偶极分子内力。过量的热量(烹饪)蛋白质会破坏蛋白质二级结构所必需的氢键。
极端环境或化学变性剂的存在(如可以破坏二硫键的还原剂)会导致蛋白质变性并失去其二级和三级结构,形成“无规卷曲”。在某些条件下,完全变性的蛋白质可以恢复到其天然状态。有意变性用于分析生物分子的各种方法中。
细胞内的复杂环境通常需要伴侣蛋白和其他生物分子,以便蛋白质正确形成天然状态。
蛋白质是生命体必不可少的组成部分。人体发育需要与蛋白质的合成同步进行。然而,蛋白质包含着许多尚未解开的谜团,例如蛋白质折叠。折叠是蛋白质必要的活动,它们需要折叠才能继续发挥生物活性。折叠也是每个蛋白质为了获得稳定构象而经历的过程。但有时这个过程会发生错误,科学家称之为蛋白质错误折叠。蛋白质错误折叠会导致人类、动物和生物体出现多种疾病,例如阿尔茨海默病和疯牛病。正因为如此,蛋白质折叠和错误折叠的研究变得至关重要。在揭示蛋白质、折叠、错误折叠及其影响的过程中,科学家们取得了许多成功,蛋白质的奥秘正逐渐被解开。科学家 W. A. (Bill) Thomasson 在文章《揭示蛋白质折叠的奥秘》中记录了许多关于蛋白质的重要内容;在这篇文章中,他阐述了阿尔茨海默病和疯牛病,以及蛋白质错误折叠的一些影响,并介绍了科学在这些领域取得的成果。Thomasson 博士首先从一般性的蛋白质折叠和错误折叠开始介绍。首先,蛋白质由氨基酸序列组成。科学家们已经发现了 20 种出现在蛋白质中的氨基酸。蛋白质结构以两种基本形状而闻名,即 α 螺旋和 β 折叠。“大多数蛋白质可能在到达稳定构象的路上会经历几个中间状态”(Campbell 和 Reece,79)。蛋白质需要折叠才能继续发挥活性。科学家们列出了三种蛋白质折叠类型:蛋白质可以是折叠的、部分折叠的或错误折叠的。在折叠过程中,“被称为伴侣蛋白的蛋白质与靶蛋白相关联;然而,一旦折叠完成(甚至之前),伴侣蛋白将离开其当前的蛋白质分子,继续支持另一个蛋白质的折叠”(Thomasson)。这篇文章的作者记录了 Anfinsen 关于蛋白质错误折叠的重要结论。在他看来,错误折叠发生在折叠过程中,当折叠出错时就会出现。蛋白质错误折叠的研究集中在温度敏感性突变上;科学家们观察到噬菌体 P22 随着温度的变化而发生突变。他们得出结论,突变蛋白比正常蛋白稳定性差。这意味着,他们在噬菌体尾部突起的结论是,错误折叠的蛋白质比正确折叠的蛋白质稳定性差,而且它们很难达到正确折叠的状态。当蛋白质发生错误折叠时,会导致许多疾病。聚集可能与错误折叠的出现同时出现,并且它会出现在大脑中,导致阿尔茨海默病和疯牛病,正如许多科学家认为的那样。蛋白质错误折叠对人类生活的一个影响就是阿尔茨海默病。这是一种老年病。根据科学家的研究,这种疾病发生在淀粉样蛋白前体蛋白发生错误折叠时。这种蛋白质会被加工成可溶性肽 Aβ。科学家们还没有完全了解这种疾病的起因。但导致错误折叠的主要原因是血液中的载脂蛋白 E (apoE)。apoE 蛋白有三种形式,即 apoE2、apoE3 和 apoE4。每种形式的 apoE 对 Aβ 的影响尚未发现,但科学家认为 apoE 可以与 Aβ 结合。在错误折叠过程中,β 淀粉样蛋白形成,在阿尔茨海默病患者中形成“神经纤维缠结”。这种疾病只发生在老年人身上,因为在淀粉样蛋白过程中,核的形成非常缓慢。这种蛋白质的突变不稳定,会导致疾病。关于 apoE 的研究仍然是一个谜,因为一些科学家表明,这种蛋白质的一种形式会导致疾病的发展,而另一种形式则会降低疾病的发展。最后,关于阿尔茨海默病的研究仍在继续,目的是确认 apoE 蛋白对 Aβ 的影响,并成功找到治疗这种疾病的方法。蛋白质错误折叠的另一个影响是疯牛病。这是一种非常危险的疾病,因为它可以从动物传播到人类。这种疾病是由朊病毒的错误折叠引起的。错误折叠的过程是朊病毒的自我复制。朊病毒是含有 DNA 和 RNA 的蛋白质颗粒。突变出现在折叠过程中,朊病毒自我复制并导致蛋白质错误折叠。它们含有 DNA 和 RNA。这是蛋白质的一种特殊情况;它可以作为自己的伴侣蛋白。由于复制,朊病毒随着正常蛋白质的增加而迅速繁殖。这种疾病表明,蛋白质折叠可以在没有遗传学的情况下发生,例如在绵羊身上进行的实验。Thomasson 博士在文章中继续介绍了有关错误折叠的更多信息,以及科学家揭示其奥秘的方法。他提供了关于 p53 蛋白及其突变的信息。它会导致癌症,也是一种蛋白质错误折叠。Thomasson 博士想表达的是他关于药物的想法,这种药物可以使蛋白质错误折叠变得更加稳定,并最大限度地减少蛋白质的错误折叠。这个想法似乎很好,但它的结果就像蛋白质折叠的奥秘一样是一个谜。关于蛋白质折叠的研究对我们的生活至关重要。错误折叠是导致许多危险疾病的主要原因之一,但我们还没有找到有效的治疗方法。蛋白质折叠的研究越来越成功,帮助人类能够摧毁由错误折叠引起的疾病。由蛋白质错误折叠引起的疾病已经成为人类需要解决的问题之一。
分子伴侣主要以辅助蛋白质折叠而闻名。伴侣蛋白不仅参与蛋白质生命周期的初始阶段。分子伴侣参与生产、维持和回收蛋白质伴侣的结构和单元。伴侣蛋白存在于胞质溶胶中,但也存在于细胞隔室中,例如膜结合的线粒体和内质网。伴侣蛋白对蛋白质正确折叠的作用或必要性各不相同。许多原核生物只有很少的伴侣蛋白,并且伴侣蛋白类型的冗余度较低,而真核生物则有大量的伴侣蛋白家族,其中包含一些冗余度。据推测,一些伴侣蛋白对于蛋白质的正确折叠至关重要,例如原核生物的例子,它们具有较少的伴侣蛋白家族变异。其他伴侣蛋白发挥的作用不那么重要,例如在真核生物中,伴侣蛋白家族内部存在更多的变异,并且会产生效率或亲和力的梯度。这种冗余或低效伴侣蛋白的存在可能在一种状态中存在,但伴侣蛋白的有效性也是其环境的函数。pH 值、空间、温度、蛋白质聚集和其他外部因素可能会使原本无效的伴侣蛋白变成更重要的伴侣蛋白。这些环境因素表明了模拟细胞体内条件或天然状态以了解需要使用伴侣蛋白的条件的重要性。这简要概括了分析和比较伴侣蛋白体内与体外功能的困难。模拟体内或细胞内的环境不仅因为物理因素,如 pH 值或温度,还因为伴侣蛋白开始使多肽构象化的時間。一些伴侣蛋白靠近核糖体,并立即附着到多肽上,以防止错误构象。其他伴侣蛋白允许多肽自己开始折叠,并在稍后附着。因此,每个伴侣蛋白的作用变得与其靠近多肽的距离以及其辅助折叠的时间和地点有关。最近的研究表明,核仁中的伴侣蛋白不仅催化蛋白质折叠,而且还催化其他对维持健康细胞重要的功能。这些核仁伴侣蛋白被称为核仁多功能蛋白 (NoMP)。例如,热休克蛋白不仅帮助其他蛋白质折叠,而且在压力时刻发挥作用,以调节蛋白质稳态。此外,有证据表明,伴侣蛋白在网络中协同工作以监督某些功能,例如处理毒素、饥饿或感染。
核仁伴侣网络被划分为不同的分支,每个分支都有其特定的功能。该网络是动态的,其网络组分的浓度或位置会随着细胞生理和环境的变化而变化。热休克蛋白 (HSP) 根据其分子量进行分类,是伴侣网络的组成部分。HSP 70s 和 90s 通过确保蛋白质正确折叠并防止蛋白质毒性来维持蛋白质稳态,而蛋白质毒性是指由于蛋白质错误折叠而导致的细胞功能损伤。HSP70s 帮助折叠新合成的蛋白质,而 HSP90s 则在折叠过程的后期发挥作用。核仁网络还包含参与核糖体生物发生或细胞中核糖体合成的伴侣。HSP70 和 DNAJ 家族中的蛋白质,它们帮助处理前 rRNA,经常在处理酿酒酵母 (一种酵母菌) 中的前 rRNA 的蛋白质复合物中被发现。其他 HSP 对于核糖体生物发生也很重要,包括 HSP90,它与 TAH1 和 PIH1 协同作用以创建小的核仁核糖核蛋白。核仁伴侣网络提供完成细胞存活所需的生物学任务所需的组织和帮助,如果它不能正常运作,可能会出现很多问题。例如,当癌细胞的 rRNA 合成水平升高时,核糖体生物发生就会增加。科学家正在研究化合物 CX-3543,该化合物可以阻止核仁与 rDNA 结合并阻止 RNA 合成,从而导致细胞死亡。有可能使用针对核仁伴侣网络中特定分支的药物来治疗功能失调的细胞。其他伴侣网络包括专门参与从头蛋白质折叠的网络,这意味着它们帮助折叠新合成的蛋白质,以及对受损蛋白质的重新折叠。在肿瘤细胞线粒体中存在的一个伴侣网络包含 HSP90 和 TRAP1,它们保护线粒体并防止细胞死亡,使癌细胞能够继续不受控制地扩散。[9]
示例:分子伴侣 (HSP 70)
[edit | edit source]HSP 70 是热休克蛋白家族中的蛋白质,与 HSP 90 一起作用。它与 HSP 90 协同作用以支持蛋白质稳态。它在折叠过程的早期与新合成的蛋白质结合。它具有三个主要结构域:N 末端 ATP 酶结构域、底物结合结构域和 C 末端结构域。N 末端 ATP 酶结合并水解 ATP,底物结合结构域对长达七个残基的中性疏水性氨基酸残基具有亲和力,而 C 末端结构域充当底物结合结构域的盖子。当 HSP 70 与 ATP 结合时,该盖子处于打开状态,当 hsp 70 与 ADP 结合时,该盖子处于关闭状态。HSP70 或 DnaK 是细菌伴侣,可以通过夹紧肽段来帮助折叠。[10]
-
HSP 70
示例:GroEL 和 GroES
[edit | edit source]GroEL 和 GroES,或 60kDa 和 10kDa,都是细菌伴侣。GroEL 和 GroES 的结构都是堆叠的环,中间为空心。蛋白质适合在这个空心中心。然后,腔室内的构象变化可以改变蛋白质的形状和折叠。[10]
示例:分子伴侣 (HSP 90)
[edit | edit source]HSP 90 是热休克蛋白家族中的蛋白质。然而,这种特殊的蛋白质与其他伴侣蛋白不同,因为 HSP90 在分子伴侣的折叠方面受到限制。相反,Hsp 90 至关重要,需要对其进行研究和理解,因为许多癌细胞已经能够接管并利用 Hsp 90 才能在许多毒性环境中生存。因此,如果有人对 Hsp90 抑制剂进行结构研究并以某种方式对其进行靶向治疗,那么就可能找到一种方法来阻止癌细胞的扩散。此外,已经进行了许多研究以测试 Hsp 90 伴侣循环是否是由 ATP 结合和水解驱动还是由其他因素驱动。但是,经过 Southworth 和 Agard 的大量研究,有足够的证据表明 HSP90 蛋白可以在没有核苷酸结合的情况下发生构象变化,而是平衡的稳定是将 Hsp90 改变为封闭状态、紧凑状态或开放状态的因素。通过 X 射线晶体学以及单电子粒子显微镜,以及通过研究酵母 Hsp90、人 Hsp90 和细菌 Hsp 90 (HtpG) 的三态构象变化,发现 Hsp90 的三种构象,很明显,特定物种存在不同的构象变化。总体而言,Hsp90 是一种伴侣蛋白,它更多地参与维持细胞内的稳态,而不是参与蛋白质折叠。Hsp90 在未来药物开发领域具有上升的潜力,因为它在帮助癌细胞存活方面发挥着至关重要的作用。
-
HSP 90
示例:分子伴侣 (TF)
[edit | edit source]这是第一个与新链从核糖体通道出来时相互作用的伴侣蛋白。在没有新链的情况下,TF 会循环打开和关闭,但是一旦存在新链,它就会与新链结合,在新链周围形成一个保护腔。为了发挥其功能,TF 会扫描新链的任何暴露的疏水性片段,并且它也可以重新与新链结合。在 TF 存在的情况下,折叠效率更高,但是,这是以速度为代价的,它可以与新链保持 30 秒以上。当疏水性部分被掩盖时,随着折叠向天然状态进展,会触发新链的释放。
示例:分子伴侣 (YidC、Alb3、Oxa1)
[edit | edit source]YidC、Alb3 和 Oxa1 是促进蛋白质插入质膜的蛋白质。YidC 是一种仅包含两个多肽链的蛋白质。其结构的形成得到了特定磷脂的支持。YidC 蛋白可以在革兰氏阴性和革兰氏阳性细菌中找到。Oxa1 可以在线粒体的内膜中找到。Alb3 位于叶绿体内部的类囊体膜中。实验表明,YidC 蛋白积极地促进 Pf3 衣壳蛋白的插入。此外,YidC 还直接接触 Pf3 衣壳蛋白的疏水性片段。虽然 Oxa1 只能在线粒体中找到,但它也可以促进核内膜蛋白的插入。YidC 和 Alb3 的作用似乎是可以互换的,因为 Alb3 可以替代 E. coli 中的 YidC。此外,YidC、Oxa1 和 Alb3 都支持 Sec 独立蛋白质的插入。Oxa1 仅支持 Sec 独立蛋白质的插入,因为酵母细胞中的线粒体没有 Sec 蛋白。
NLR
[edit | edit source]富含亮氨酸的核苷酸结合域 (NLR) 提供了一种植物和动物中都存在的病原体传感机制。它们可以通过隐秘的机制直接或间接地由病原体分子衍生物触发。研究表明,HSP90、SGT1 和 RAR1 等分子伴侣是 NLR 蛋白的主要稳定成分。HSP90 可以通过三种实用的方式监测其对应客户(适用于 NLR 蛋白)的功能:促进功能阈值的稳态、激活刺激依赖性活性以及提高进化能力。
植物包含许多 NLR 基因,这些基因被认为在 LRR 结构域中是多态性的,以便熟悉高度多样化的病原体效应子。NLR 传感器的稳定性将是决定病原体识别的机制。HSP90 系统对植物有利,因为它会将亚稳态 NLR 蛋白耦合起来,并在信号传导能力强的条件下稳定它们。这将允许掩盖可能造成损害的突变。
蛋白质折叠中底物结合的分子伴侣机制
[edit | edit source]众所周知,伴侣蛋白协同工作,帮助蛋白质折叠,防止错误折叠。然而,伴侣蛋白如何帮助蛋白质折叠的机制尚未完全了解。最近关于Hsp40和Hsp70的研究为伴侣蛋白及其底物的机制提供了更多见解。Hsp40家族包含许多具有不同J结构域的Hsp40。当Hsp40与Hsp70结合时,不同的J结构域将执行不同的Hsp70 ATP酶活性。在蛋白质折叠过程中,未折叠的多肽与Hsp40伴侣蛋白结合。从那里,Hsp40的J结构域与Hsp70的核苷酸结合域(NBD)结合。当HSP70 NBD上的ATP水解为ADP时,Hsp70底物结合域发生构象变化。这导致Hsp70对多肽底物的亲和力更高,并从Hsp40上解离底物。当ADP被ATP交换时,多肽底物从Hsp40释放。研究表明,核苷酸交换因子以降低Hsp70对ADP的亲和力的方式改变了Hsp70 ATP酶域上的叶瓣。一旦多肽从Hsp70释放,它就可以折叠到其天然状态,或者如果发生错误折叠,它可以被伴侣蛋白重新折叠。如果与Hsp70结合的多肽被E3泛素连接酶CHIP识别,它将被降解。[11]
小热休克蛋白和α-晶状体蛋白作为分子伴侣蛋白
[edit | edit source]众所周知,小热休克蛋白 (sHSPs) 和相关的 α-晶状体蛋白 (αCs) 几乎是无处不在的蛋白质,它们被各种压力强烈诱导,但也构成性地在许多生物体的多种细胞类型中发挥作用。广泛的研究表明,大多数 sHSPs 和 αCs 可以通过结合变性蛋白质和逆转变性来充当 ATP 独立的分子伴侣蛋白。这种方法从而保护细胞免受不可逆蛋白质聚集造成的损害。许多遗传性疾病被发现是由 sHSP/αCs 中的缺陷引起的,这些蛋白质在神经退行性疾病和其他与蛋白质错误折叠相关的疾病中积累。sHSP/αC 蛋白的大小范围从 ~12 到 42 kDa,并且是位于 C 末端的约 90 个氨基酸的结构域,称为 αC 结构域。sHSP-底物复合物可以通过尺寸排阻色谱观察。它们很大且异质,并且它们的大小分布取决于 sHSP/αC 与底物的比率以及底物聚集速率,底物聚集速率受浓度和温度的影响。底物结合通常通过 sHSP/αC 上可用疏水表面的增加来促进,这似乎在没有明显损失定义的 sHSP/αC 二级和三级结构的情况下发生。sHSP/αCs 上没有单个特定的底物结合表面。相反,似乎许多位点有助于底物相互作用,并且结合可能因不同底物而异,具体取决于底物展开时暴露的表面的构象。然而,一些 sHSP/αCs 识别几乎任何展开的蛋白质,这表明它们作用于任何不稳定或受损的细胞成分。
蛋白质折叠的能量景观
[edit | edit source]如果蛋白质随机且不可预测地折叠,则到达天然构象所需的时间将远远大于实际所需的时间。关于蛋白质如何自然有效地折叠的当前理论涉及某种“漏斗”——这个想法是,存在着并非一步一步地达到正确的三维结构的方法,而是从上到下逐渐变窄的许多路径。漏斗从顶部开始,从顶部能量不利折叠向下进行,到底部能量有利于正确折叠。
1961 年,克里斯蒂安·安芬森进行的实验激发了蛋白质依靠能量学和热力学来达到其天然折叠的想法,当时他发现核糖核酸酶在被变性后可以自发地重新折叠成其适当的结构,而无需其他分子的帮助。蛋白质折叠不是随机的这一理论的进一步理论证明见于莱文撒尔悖论,该悖论指出,一个长 100 个氨基酸的蛋白质大约需要 10^81 年才能达到正确的构象,而实际上,它只需要毫秒到一天的时间。
这些漏斗模型(如 Go 型模型)显示出具有山丘和凸起的漏斗,代表蛋白质在能量漏斗中向下移动时走最小的阻力路径。这些凸起被称为“挫折点”。人们认为,具有最少挫折点或凸起的漏斗折叠成其天然形式的速度更快,因为存在的能量边界更少。虽然这些模型是简化的尝试,并没有考虑到错误折叠,但它们在许多蛋白质的情况下仍然证明是准确的。
另一个使用算法和计算机的模型是经验力场。该模型使用数十万台闲置运行的计算机以惊人的精度计算 50 个氨基酸以下的蛋白质折叠场景。但是,这些计算机模型有时会高估不太可能的折叠结构或产生很少或从未见过的折叠模式。例如,一些模拟/算法往往会卡在局部最小值中,无法到达全局最小值,即正确折叠的蛋白质。Go 型模型等简单模型不仅可以预测折叠的蛋白质,还可以预测确定蛋白质折叠速率的过渡状态。
这些模型才刚刚开始展示蛋白质折叠中间阶段的动力学。因此,这是一个正在进一步研究的领域。对蛋白质折叠动力学的理解还不太成熟,蛋白质在初始氨基酸链和最终产物之间的运动也是一个正在研究的领域。能量景观模型也难以解释拥挤和聚集等外部因素。一种这样的外部相互作用的例子,称为“多米诺骨牌交换”,涉及从一种蛋白质交换单体到另一种蛋白质,以激活两种蛋白质的正确折叠。
最近的研究将人力和计算机能力结合起来,正确预测蛋白质构象。像 fold.it 这样的网站,由华盛顿大学计算机科学系监督,将折叠问题变成了视频游戏,让世界各地的人们像解谜游戏一样解决蛋白质折叠问题。用户被提供部分折叠的蛋白质,通常是那些卡在局部有利构象中,而这种构象对于计算机来说似乎是最优的,并被要求将蛋白质重新配置成看起来更稳定的形状。利用计算机的计算能力和速度以及人类在空间中操纵物体的能力,在帮助更有效地解决蛋白质折叠问题方面显示出希望。
协同性和蛋白质折叠速率
[edit | edit source]蛋白质折叠中表达的协同性是蛋白质折叠最引人注目的方面之一。与传统的蛋白质折叠涉及复杂和异质机制的观点相反,许多蛋白质表现出的协同两态折叠动力学相对简单。由于其简单性,最近通过应用协同两态折叠动力学来理解决定协同性和蛋白质折叠速率多样性的努力。
蛋白质的协同性通常是指一种机制,在这种机制中,结构区域的存在使得在蛋白质折叠中增加有序性更加有利。如前所述,小球状蛋白质的协同两态折叠动力学相对简单,成为许多科学家的研究兴趣。对能够对单个分子进行激发的敏感实验,可以对过渡时间进行估计,揭示了两种状态的协同性。
由两态折叠蛋白质揭示的一般趋势可以总结为以下两点。首先,拓扑结构更复杂的蛋白质往往比拓扑结构更简单、局部的蛋白质折叠得更慢;其次,更大的蛋白质往往比更小的蛋白质折叠得更慢。这里蛋白质的大小和大小是根据其链长定义的。
蛋白质折叠动力学受过渡态能量增益和熵损失决定的自由能垒控制。在描述这种模式时,科学家引入了能量景观理论的最小挫折原则。该理论指的是天然结构在蛋白质折叠过程中比其他随机构象具有更低的自由能的概念。因此,天然结构有利于蛋白质快速折叠,并作为向天然状态、蛋白质的功能形式或三级结构的驱动力。该原则可以用漏斗能量景观来表达。
漏斗能量景观
漏斗能量景观将折叠蛋白质的能量景观描绘为一个粗糙的漏斗。粗糙来自蛋白质折叠过程中的非天然接触。该景观本质上是多维的,因此漏斗是在二维图上的投影。漏斗的深度表示构象状态的能量;漏斗的宽度表示熵的度量。漏斗的瓶颈表示折叠蛋白质的过渡态构象,而漏斗的底部表示蛋白质的天然状态。当蛋白质走向其天然状态时,它会经历熵损失,并达到更低的能量状态。漏斗能量景观为科学家提供了方便的图示,让他们可以想象蛋白质折叠过程的热力学和动力学。
φ (phi) 值
另一个在蛋白质折叠动力学研究中起作用的概念是φ (phi) 值。该值指的是过渡态构象中天然结构含量的近似测量。与 φ 值的比较是检查研究蛋白质折叠动力学的各种模型的方法之一。
一般观察结果
第一个提到的趋势可以从熵的角度很容易理解。拓扑结构更复杂的蛋白质,或具有长程相互作用的蛋白质,与具有短程相互作用的蛋白质相比,在折叠方面的熵成本预计会更高。第二种趋势最近通过专注于蛋白质大小对折叠速率影响的实验得到了证实。研究发现,仅基于链长的简单模型可以粗略地预测蛋白质的折叠速率和稳定性。
Go¯模型
粗粒度拓扑模型(Go¯模型)被广泛用于研究蛋白质折叠的协同性和动力学,因为人们注意到天然蛋白质的拓扑结构决定了折叠机制。典型的 Go¯模型简化了蛋白质,其中只有一种相互作用稳定折叠的蛋白质。早期的模型经常检查蛋白质折叠中起作用的非加性力,例如侧链排序和疏水效应。近年来,更多种类的 Go¯模型被用于研究蛋白质折叠动力学。
- 项目符号列表项
- 具有蛋白质天然接触之间非成对加性相互作用的 Go¯模型(这里指的是 Eastwood 和 Wolynes 的模型)表明,短程多体相互作用可以增加自由能垒,并使过渡态构型更加局部化。
- 另一方面,晶格 Go¯模型表明,局部和核心埋藏相互作用的耦合促进了协同性,并增加了与接触顺序的相关性。
- 具有成对加性相互作用的 Go¯模型,特别是那些专注于三体相互作用强度变化的影响和 φ 值的模型,表明三体相互作用增加了能垒,并增加了与测量的 φ 值的一致性。
- 此外,溶剂介导的相互作用也被引入 Go¯模型。当接触之间的相互作用被溶剂分离的最小值和脱溶能垒取代时,观察到蛋白质功能的动力学和协同性随着脱溶能垒高度的增加而增加。溶剂介导的 Go¯模型的优点是它有助于区分短程接触和长程接触,从而区分具有简单拓扑结构的蛋白质和具有更复杂拓扑结构的蛋白质。在溶剂介导的 Go¯模型的研究中,经常使用“化学计量图”。化学计量图是一种用不同浓度的变性剂来表示蛋白质折叠动力学数据的图形,这种变性剂会破坏蛋白质的天然结构。
- 变分 Go¯模型通过排除处于天然状态下紧密接触的残基之间的体积力来提高协同性。在这个模型中,实现以下目标:a)长程接触的协同性更强;b)计算速率的范围更广;c)计算的 φ 值得到改善。还有一些 Go¯模型完全专注于蛋白质折叠能量景观的漏斗方面,而忽略了非天然接触的影响。
其他模型,例如毛细管模型,假设折叠核的体积与单体的数量成比例。在这种模型中,表明协同性增加往往会减慢动力学并使能量景观平滑。
结论。
近年来,具有非加性力的拓扑模型的发展正在成为理解蛋白质折叠速率协同性的更流行和可靠的方法。该模型的改进显示出其在更明确和深入地理解决定蛋白质折叠速率和机制方面的光明前景。能够实现长程接触的 Go¯模型变得更加协同,φ 值更加准确,需要在蛋白质折叠动力学和折叠机制的研究中得到进一步改进和更多关注。
蛋白质序列、结构和功能之间的关系
[edit | edit source]在过去的 20 年中,人们开发了几种蛋白质预测方法。还没有开发出适用于所有蛋白质的通用方法,因为每种方法都有其优点和缺点。开发这种方法的难度在于我们对蛋白质序列、结构和功能之间高度错综复杂的关系的理解还不够完善。
Anfinsen 提出了将氨基酸序列与其结构相关联的理论。他证明了一种变性(未折叠)蛋白质可以自发地恢复其天然三级结构。这种方法也是将功能分配给蛋白质结构的有用贡献者。蛋白质研究人员可以预测疏水底物可能与蛋白质的疏水区域结合,反之亦然,对于带电区域也是如此。这种方法的问题是,它没有考虑某些因素,例如非典型环境条件。
人们认为,相似的排序意味着相关的结构。这种理论只对少数蛋白质适用。研究人员发现,蛋白质折叠的相似性并不总是与其蛋白质序列相关。由于这些发现,1995 年提出了“帕拉塞尔苏斯挑战”。“帕拉塞尔苏斯挑战”背后的理论是开发两种蛋白质,它们的序列相似性超过 50%,但它们的折叠完全不同。该挑战在 1997 年得到了满足,当时出现了两个共享 88% 序列同一性(GA88 和 GB88)的蛋白质序列。最近的研究表明,只要 3 个突变就足以诱导不同的折叠模式。尽管“帕拉塞尔苏斯挑战”的结果非常有趣,但它们很少在自然界中发生。
功能收敛导致将特定功能分配给结构的问题。各种结构可以采用相似的功能,但有些结构也可以采用非常不同的功能。但是,某些折叠与特定功能之间存在显着相关性。功能预测中有两个主要变量:(1)结合位点的位置,以及(2)位点上的功能范围。对功能有贡献的金属、离子、辅助因子和其他蛋白质也必须考虑在内。在通过晶体学确定结构时,会遇到一个问题。PROCOGNATE 资源和 PIDA 数据库为这个问题提供了一个解决方案。
定义蛋白质功能的一种广泛使用的方法源自基因本体,它包含三个图结构,其中定义了功能术语和它们之间的关系。基因本体的局限性出现在非位置蛋白质,以及当蛋白质在晶体结构中没有与配体之间定义的关系时。试图弥合这一差距的其他发展包括蛋白质特征本体 (PFO [29]) 和分布式注释系统 (DAS[30])。
两种方法用于确定功能位点:(1)在不知道位点在哪里或它结合什么的情况下,或者(2)在事先知道相互作用伙伴的情况下。最常用的方法涉及生物信息学,例如 SOIPPA 方法。序列保守性是将功能分配给蛋白质的一个非常重要的贡献者,但很难确定残基是由于结构原因还是功能原因而保守的。另一种方法涉及基于能量的方法。最近开发的 ProFunc 服务器结合了 InterProScan 和 BLAST 搜索等方法。
预测结合位点(本身极其复杂)只是拼图的第一步。下一步是根据生化功能确定整体功能,更具挑战性的是确定其生物学作用。当研究人员发现蛋白质功能不仅取决于其最终折叠产物时,分析蛋白质功能的难度增加了另一层复杂性。蛋白质在其部分变性和完全变性状态下可能具有功能。综上所述,可以肯定地说,我们还有很多关于蛋白质序列、结构和功能之间关系的知识需要学习。
结构域交换、折叠和错误折叠
[edit | edit source]蛋白质中发生的结构域交换可能在蛋白质的折叠或错误折叠过程中很重要。结构域交换发生在两个或多个相同的蛋白质链相互交换时。结构域交换可以被认为是单体和寡聚体交换的一种机制。寡聚体交换中发生的是,来自一种蛋白质的一个单体将与来自另一种蛋白质的另一个相同的单体交换。这种结构域交换机制已在各种蛋白质中观察到,超过 40 种不同的蛋白质。交换机制对某些蛋白质的功能很重要。例如,对于特定蛋白质 p13suc1,人们已经观察到交换和聚集是相关的,这意味着它们具有共同的机制。P13suc1 在细胞周期进程中是细胞周期蛋白依赖性激酶 (Cdk) 所必需的。P13suc1 具有两种不同的状态,一种是单体,另一种是交换的二聚体。结构域交换部分是一个 β 链,它不是一个独立折叠的结构域。在研究过程中,人们发现 β4 在与 β2 接触时起着至关重要的作用,因为它们在折叠过程的早期阶段相互配对。因此,对于 p13suc1,已经证明,已交换的区域负责蛋白质的折叠和错误折叠。蛋白质中似乎存在折叠和错误折叠之间的竞争,因为多肽链可以折叠成结构或错误折叠成淀粉样纤维。在蛋白质折叠中,更重要的是折叠核的存在,折叠核在过渡态中形成蛋白质链的一部分。人们还发现了参与蛋白质折叠核位置的残基与淀粉样蛋白区域之间的相关性,以及纤维形成和蛋白质折叠可能包含关键残基的重要信息。通过对蛋白质折叠进行建模并观察寡聚形式中的可交换区域,可以看到这种关系是折叠和错误折叠的原因。这可能使研究人员更接近解决蛋白质求解问题并了解蛋白质是如何获得其折叠指令的。参考资料:http://www.benthamscience.com/open/tobiocj/articles/V005/27TOBIOCJ.pdf
-
结构域交换
死亡折叠超家族包含 4 种亚家族结构。它们分别是死亡结构域 (DD)、死亡效应结构域 (DED)、半胱天冬酶募集结构域 (CARD) 和 PYrin 结构域 (PYD)。这些亚家族结构参与多聚体复合物的组装,这些复合物可能与细胞炎症和死亡有关。
死亡折叠结构域的结构和功能
目前已知有 102 种蛋白质具有死亡折叠超家族结构域。这些结构域包含同型相互作用。这些蛋白质包括 39 个 DD、8 个 DED、33 个 CARD 和 22 个 PYD。尽管这些结构域在序列上最多有 90% 的差异,但它们都具有典型的死亡折叠。这种折叠“包含一个球状结构,其中 6 个两亲α螺旋以反平行α螺旋束的形式排列,具有希腊键拓扑结构”(Peter Vandenabeele 等人,2012 年)。构成这些亚家族之一的死亡结构域之间的差异在于 α 螺旋的长度和方向以及疏水残基和带电残基在复合物表面上的分布。
死亡折叠结构域的推测功能是介导大型寡聚信号复合物的组装。在这些复合物中,半胱天冬酶和激酶的活性增加。在此之前,人们对具有死亡折叠结构域的蛋白质组装体的结构构象知之甚少。
三种不同的相互作用类型
I 型相互作用:来自一个死亡折叠结构域的螺旋 1 和 4(Patch Ia)的残基与来自另一个死亡折叠结构域的螺旋 2 和 3(Patch Ib)的残基相互作用。II 型相互作用:来自一个死亡折叠结构域的螺旋 4 和螺旋 4 和 5 之间的环(Patch IIa)的残基与来自另一个死亡折叠结构域的螺旋 5 和 6 之间的环(Patch IIb)的残基相互作用。III 型相互作用:来自一个死亡折叠结构域的螺旋 3(Patch IIIa)的残基与位于另一个死亡折叠结构域的螺旋 1 和 2 之间的环以及螺旋 3 和 4 之间的环(Patch IIIb)的残基相互作用。
先前的理论认为,这三种相互作用类型在整个死亡折叠超家族中是保守的,但现在看来,相同类型死亡折叠结构域之间的相互作用存在差异。
死亡折叠结构域的晶体分析
只有三种 DD 复合物的晶体结构被分析过。它们是 PIDDosome、MyDDosome 和 Fas/FADD-DISC。对这些结构的分析表明,DD 可以参与多达六种相互作用。
死亡结构域和药物
死亡结构域已被证明可以促进导致炎症和细胞死亡的多聚体复合物的组装。了解这些结构可以通过阻止或触发这些寡聚体复合物的形成,从而产生治疗益处。可能受这些相互作用影响的疾病包括神经退行性疾病和炎症性疾病,以及许多其他具有炎症或过度细胞死亡特征的疾病。
虽然折叠通常是蛋白质功能的主要贡献者,但有些蛋白质并不折叠成特定的结构,但仍然具有功能。这些蛋白质通常在不同的形式之间转换,或者具有不保持特定形状的无序区域,而不是特定的结构。
正如蛋白质的折叠由其氨基酸序列决定一样,非折叠蛋白质之所以是非折叠的,也是因为它们的序列。这些蛋白质的某些氨基酸含量往往比折叠蛋白质少得多,而另一些氨基酸则多得多。具体来说,非折叠蛋白的形成疏水核心的氨基酸含量较少,而表面氨基酸含量较多。疏水核心的形成是大多数蛋白质折叠的第一步,一旦形成,核心往往会提供稳定最终结构的驱动力。如果没有形成核心的氨基酸,蛋白质就不会被驱动到特定的结构。
一些在非压力条件下完全折叠且不活跃的分子伴侣被称为条件性无序蛋白。当这些伴侣暴露于不同的压力条件下时,它们会呈现部分无序的构象。这种无序非常重要,因为它们能够保护细胞免受压力源的影响。对这些无序伴侣的研究使人们对蛋白质无序在分子识别中的功能作用有了更多了解。X 射线晶体学是一种有用的技术,有助于可视化蛋白质的结构。根据这种技术,超过 95% 的整个分子由 25% 的晶体结构表示,而所有其他分子由于这些区域上的多种构象,其序列中超过 5% 的电子密度缺失。蛋白质实际上具有一些无序构象,这些无序蛋白在连续光谱上从非常灵活的结构状态到静态结构状态的一端。这种无序中可能只有一部分蛋白质或整个完整的多肽链。因此,仅研究蛋白质的某些部分并不能帮助总结蛋白质的灵活性。“条件性无序”一词表示蛋白质的无序可能在某些特定条件下发生,而在其他条件下可能不会发生。在蛋白质中看到内在无序的情况非常普遍。例如,估计真核蛋白质的 30% 到 50% 具有超过 30 个氨基酸,这些氨基酸在体外违反了已定义的二级结构,并且已经预测存在许多完整的无结构蛋白质。尽管使用了许多计算方法,但仍然很难验证细胞内蛋白质折叠状态。许多蛋白质在细胞内部分或完全折叠,而实际上在细胞内是无结构的。这些机会的数量仍然不确定。然而,人们认为,适当的结合对的存在将使无序蛋白进入其折叠状态,这意味着体外固有无序蛋白的百分比在细胞内可能更低。细胞内的稳定相互作用可能会降低无序的程度。通过化学位移、残余偶极偶合和顺磁共振增强测量,NMR 成为提供有关蛋白质无序程度的详细信息的一种好方法。
无序蛋白有两种状态。一种表现出高度的灵活性,而另一种状态是蛋白质表现出更多有序的状态。因此,为了了解无序和功能之间的因果关系,研究这两种状态至关重要。许多无序蛋白,如 DNA、蛋白质和膜,一旦找到结合伙伴就会重新折叠。此外,还可以发生有序到无序到有序的转变。参与多种结合的蛋白质是条件性无序的很好的例子。与已经井然有序的结合表面相比,在结合之前无序的结合表面能够更好地折叠成与其他伙伴不同的构象。“构象选择假说”表明,构象集合的不同成员可以通过不同伙伴的结合而稳定。另一方面,“结合折叠”模型提出,蛋白质在与不同伙伴结合时可能能够折叠成不同的构象。
对整个蛋白质组的预测表明,真核生物中无序蛋白的频率远大于原核生物,而两组原核生物,古细菌和细菌,的频率相似。在哺乳动物中,大约一半的蛋白质被预测具有大型无序区域,其中大约四分之一是完全无序的。
无序蛋白在信号传导和调控中普遍存在,特别是在与生物分子(如核酸和其他蛋白质)的相互作用中。分子识别、蛋白质组装和修饰通常涉及具有无序区域的蛋白质。这些蛋白质能够与多个分子伴侣相互作用,这意味着它们在蛋白质-蛋白质网络中也很常见,要么作为枢纽蛋白,要么作为与枢纽蛋白相互作用的蛋白质。
无序蛋白与多种人类疾病有关。特别是淀粉样蛋白疾病,涉及错误折叠蛋白质的积累,似乎与无序蛋白有关,这可能是因为它们的变异区域使其更有可能具有有利于其积累的结构。这一类别包括许多神经退行性疾病,如阿尔茨海默病和帕金森病。
可以使用计算机算法通过氨基酸的一级结构或氨基酸序列来分析和比较氨基酸的结构或折叠,以及由此延伸的功能。使用计算机可以增强对未知折叠模式的氨基酸序列与具有相似氨基酸序列的已知折叠的比较。一种名为蛋白质基本局部比对搜索工具,或 蛋白质 BLAST 的计算机自动化工具,是一个向公众开放的免费搜索工具,允许在线数据库中快速比较氨基酸序列。此工具的输出是氨基酸的匹配百分比和序列匹配的已知特性。此外,由于氨基酸序列基于 DNA 序列,三个碱基编码一个氨基酸,因此可以使用 DNA BLAST 在 DNA 水平上分析所研究的蛋白质。公共氨基酸和 DNA 序列数据库的整合以及计算机算法的应用,通过允许世界各地的科学家共享和分析序列,加速了基因组和蛋白质组领域的发展。
附录
计算机的作用
创建 BLAST 程序的科学家是来自 NIH 的 Webb Miller、David J. Lipman、Warren Gish、Eugene Myers 和 Stephen Altschul
分子伴侣
Pain, Roger H. 蛋白质折叠机制。第 2 版。364-85
蛋白质折叠的能量景观
Cho, Samuel S. “蛋白质折叠、结合和聚集的能量景观:简单漏斗及超越”。加州大学圣地亚哥分校博士论文(2007)。
Cheung, Margaret S. “与分子功能相关的蛋白质折叠动力学的能量景观方面”。加州大学圣地亚哥分校博士论文(2003)。
Yang, Sichun. “扩展蛋白质折叠动力学理论框架”。加州大学圣地亚哥分校博士论文(2006)。
分子内相互作用
Pain, Roger H. “蛋白质折叠机制” 第 2 版。
http://www.nature.com/horizon/proteinfolding/background/importance.html
Berg “生物化学” 第 6 版
计算机模拟研究帮助确定了共翻译折叠途径的几个特征。首先,确定了体内蛋白质折叠是一个矢量过程,这是一个分散变化。其次,从 N 端到 C 端发育中的多肽的共翻译矢量折叠导致从核糖体通道中出现的多肽不同区域的顺序结构。第三,在蛋白质合成过程中,发育中的多肽链与核糖体的连接减少了生长链的构象空间和自由度。这限制了可能的中间体的数量,并减少了可能的折叠途径的数量。第四,共翻译蛋白质折叠在核糖体上多肽链合成过程的早期开始,一些元素在核糖体通道内形成。第五,折叠催化和分子伴侣在生长链从通道中出现后立即与其相互作用。这加速了蛋白质折叠中的缓慢步骤,并防止蛋白质错误折叠。
- ↑ “阿尔茨海默病”. 俄亥俄州立大学医疗中心. 2009. 检索于 2009-10-09.
{{cite web}}: 外部链接位于|publisher= - ↑ Lindquist, Susan (1999). “什么是朊病毒?”. 检索于 2009-10-09.
- ↑ “疯牛病和变异型克雅氏病”. eMedicine 健康. 检索于 2009-10-09.
{{cite web}}: 外部链接位于|publisher= - ↑ “揭开蛋白质折叠之谜” (PDF). [Thomasson, W.A. “揭开蛋白质折叠之谜”]. 检索于 2009-10-18.
- ↑ “折叠解决囊性纤维化”. [1]. 检索于 2009-10-18.
{{cite web}}: 外部链接位于|publisher= - ↑ “遗传疾病概况:镰状细胞贫血”. [2]. 检索于 2009-10-18.
{{cite web}}: 外部链接位于|publisher= - ↑ “亨廷顿舞蹈症基础”. [3]. 检索于 2009-10-18.
{{cite web}}: 外部链接位于|publisher= - ↑ a b c 无效的
<ref>标签;未为名为annu的引用提供文本 - ↑ Piotr Banski, Mohamed Kodiha and Ursula Stochaj (2010). “核仁中的伴侣蛋白和多任务蛋白:为了生存而相互连接?”. 检索于 2010-10-16.
- ↑ a b Joan L. Slonczewski, John W. Foster. "Microbiology: An Evolving Science."
- ↑ Summers, Daniel W., and Peter M. Dougla (2009). "Polypeptide Transfer from Hsp40 to Hsp70 Molecular Chaperones". Retrieved 2010-10-24.
{{cite web}}: CS1 maint: multiple names: authors list (link) - ↑ 11. Kersse K, Verspurten J, Vanden Berghe T, Vandenabeele P. The death-fold superfamily of homotypic interaction motifs. Trends in biochemical sciences. 2011;36(10):541–52. Available at: http://www.ncbi.nlm.nih.gov/pubmed/21798745. Accessed October 29, 2012.
12. Small heat shock proteins and α-crystallins: dynamic proteins with flexible functions. Basha E, O'Neill H, Vierling E. Trends Biochem Sci. 2012 Mar;37(3):106-17. Epub 2011 Dec 14
Conditional disorder in chaperone action. Bardwell JC, Jakob U. Trends Biochem Sci. 2012 Sep 24. pii: S0968-0004(12)00127-2. doi: 10.1016/j.tibs.2012.08.006. [Epub PMID 23018052 [PubMed - as supplied by publisher] "Molecular Biology of the cell." Fifth Ed-Alberts, Johnson, Lewis, Raff, Roberts, Walter. pg. 716-717
Braakman, Ineke, and Neil J. Bulleid. "Protein Folding and Modification in the Mammalian Endoplasmic Reticulum." Annual Review of Biochemistry. 80. (2011): 71-99. Web. 29 Oct. 2011. <http://www.annualreviews.org/doi/pdf/10.1146/annurev-biochem-062209-093836>.
Cabrita LD, Dobson CM, Christodoulou J. Protein folding on the ribosome. Current Opinion in Structural Biology 2010, doi:10.1016/j.sbi.2010.01.005
A Keith Dunker, Israel Silman, Vladimir N Uversky, Joel L Sussman. "Function and structure of inherently disordered protein." Curr Opin Struct Biol. 2008 Dec;18(6):756-64
Booth Paula J, Curnow Paul. Folding Scene Investigation: Membrane Proteins. Current Opinion in Structural Biology 2009, doi:10.1016/j.sbi.2008.12.005
Heijne, Gunnar Von. "Membrane Protein Folding and Insertion." Annual Review of Biochemistry 80 (2011): 157-160. 26 Oct. 2011 <http://www.annualreviews.org/doi/full/10.1146/annurev-biochem-111910-091345?url_ver=Z39.88-2003&rfr_id=ori:rid:crossref.org&rfr_dat=cr_pub%3dpubmed>
Kuhn, Andreas, Rosemary Stuart, Ralph Henry, and Ross E. Dalbey. "The Alb3/Oxa1/YidC protein family: membrane-localized chaperones facilitating membrane protein insertion?" TRENDS in Cell Biology 13 (2003): 510-516. 26 Oct. 2011 <http://www.cell.com/trends/cell-biology/abstract/S0962-8924(03)00196-X>
表 1:Berg,Jeremy。二级结构中氨基酸残基的相对频率。2012。生物化学,纽约。印刷。
Voet,Donald,Judith G. Voet。生物化学第三版。新泽西州:John Wiley & Sons,Inc,2004。印刷。